Dynamic and Programmable DNA-Templated Boronic Ester Formation†
Anthony R. Martin
Institut des Biomolécules Max Mousseron (IBMM), UMR 5247 CNRS–Universités Montpellier 1 et 2, Place Bataillon, 34095 Montpellier (France), Fax: (+33) 4-6714-2029
Search for more papers by this authorDr. Ivan Barvik
Institute of Physics, Faculty of Mathematics and Physics, Charles University, Ke Karlovu 5, 121 16 Prague 2 (Czech Republic)
Search for more papers by this authorDr. Delphine Luvino
Institut des Biomolécules Max Mousseron (IBMM), UMR 5247 CNRS–Universités Montpellier 1 et 2, Place Bataillon, 34095 Montpellier (France), Fax: (+33) 4-6714-2029
Search for more papers by this authorCorresponding Author
Dr. Michael Smietana
Institut des Biomolécules Max Mousseron (IBMM), UMR 5247 CNRS–Universités Montpellier 1 et 2, Place Bataillon, 34095 Montpellier (France), Fax: (+33) 4-6714-2029
Institut des Biomolécules Max Mousseron (IBMM), UMR 5247 CNRS–Universités Montpellier 1 et 2, Place Bataillon, 34095 Montpellier (France), Fax: (+33) 4-6714-2029Search for more papers by this authorCorresponding Author
Dr. Jean-Jacques Vasseur
Institut des Biomolécules Max Mousseron (IBMM), UMR 5247 CNRS–Universités Montpellier 1 et 2, Place Bataillon, 34095 Montpellier (France), Fax: (+33) 4-6714-2029
Institut des Biomolécules Max Mousseron (IBMM), UMR 5247 CNRS–Universités Montpellier 1 et 2, Place Bataillon, 34095 Montpellier (France), Fax: (+33) 4-6714-2029Search for more papers by this authorAnthony R. Martin
Institut des Biomolécules Max Mousseron (IBMM), UMR 5247 CNRS–Universités Montpellier 1 et 2, Place Bataillon, 34095 Montpellier (France), Fax: (+33) 4-6714-2029
Search for more papers by this authorDr. Ivan Barvik
Institute of Physics, Faculty of Mathematics and Physics, Charles University, Ke Karlovu 5, 121 16 Prague 2 (Czech Republic)
Search for more papers by this authorDr. Delphine Luvino
Institut des Biomolécules Max Mousseron (IBMM), UMR 5247 CNRS–Universités Montpellier 1 et 2, Place Bataillon, 34095 Montpellier (France), Fax: (+33) 4-6714-2029
Search for more papers by this authorCorresponding Author
Dr. Michael Smietana
Institut des Biomolécules Max Mousseron (IBMM), UMR 5247 CNRS–Universités Montpellier 1 et 2, Place Bataillon, 34095 Montpellier (France), Fax: (+33) 4-6714-2029
Institut des Biomolécules Max Mousseron (IBMM), UMR 5247 CNRS–Universités Montpellier 1 et 2, Place Bataillon, 34095 Montpellier (France), Fax: (+33) 4-6714-2029Search for more papers by this authorCorresponding Author
Dr. Jean-Jacques Vasseur
Institut des Biomolécules Max Mousseron (IBMM), UMR 5247 CNRS–Universités Montpellier 1 et 2, Place Bataillon, 34095 Montpellier (France), Fax: (+33) 4-6714-2029
Institut des Biomolécules Max Mousseron (IBMM), UMR 5247 CNRS–Universités Montpellier 1 et 2, Place Bataillon, 34095 Montpellier (France), Fax: (+33) 4-6714-2029Search for more papers by this authorThe MENRT (A.R.M.), the Région Languedoc-Roussillon (D.L.), and the CNRS are gratefully acknowledged for financial support. I.B. thanks the Ministry of Education, Youth and Sports of the Czech Republic (project no. MSM 0021620835). We thank Dr. C. Mari for helpful discussions.
Graphical Abstract
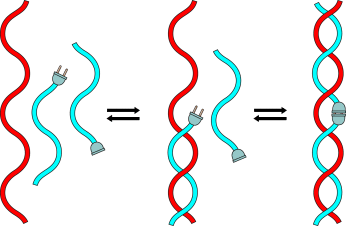
Steckerverbindung: Eine Helix mit einer Boronat-Verknüpfung wurde mithilfe eines DNA- oder RNA-Templats aufgebaut, indem geeignete Halbsequenzen – eine mit einer Boronsäure am 5′-Ende und eine mit einem Ribonucleotid am 3′-Ende – „zusammengesteckt“ wurden. Die zwei Hybridisierungsschritte in der Drei-Komponenten-Anordnung (siehe Bild) lassen sich reversibel durch externe Reize, z. B. eine Säure oder Base, Cyanidionen oder Fructose, steuern.
Supporting Information
Detailed facts of importance to specialist readers are published as ”Supporting Information”. Such documents are peer-reviewed, but not copy-edited or typeset. They are made available as submitted by the authors.
| Filename | Description |
|---|---|
| ange_201007170_sm_miscellaneous_information.pdf8.8 MB | miscellaneous_information |
Please note: The publisher is not responsible for the content or functionality of any supporting information supplied by the authors. Any queries (other than missing content) should be directed to the corresponding author for the article.
References
- 1G. P. Chiusoli, Rend. Lincei-Sci. Fis. 2009, 20, 199–209.
- 2
- 2aL. E. Orgel, Trends Biochem. Sci. 1998, 23, 491–495;
- 2bX. Y. Li, D. R. Liu, Angew. Chem. 2004, 116, 4956–4979; Angew. Chem. Int. Ed. 2004, 43, 4848–4870;
- 2cA. P. Silverman, E. T. Kool, Chem. Rev. 2006, 106, 3775–3789;
- 2dC. Dose, S. Ficht, O. Seitz, Angew. Chem. 2006, 118, 5495–5499;
10.1002/ange.200600464 Google ScholarAngew. Chem. Int. Ed. 2006, 45, 5369–5373;
- 2eM. Röthlingshöfer, E. Kervio, T. Lommel, U. Plutowski, A. Hochgesand, C. Richert, Angew. Chem. 2008, 120, 6154–6157;
10.1002/ange.200801260 Google ScholarAngew. Chem. Int. Ed. 2008, 47, 6065–6068;
- 2fJ. P. Schrum, A. Ricardo, M. Krishnamurthy, J. C. Blain, J. W. Szostak, J. Am. Chem. Soc. 2009, 131, 14560–14570.
- 3
- 3aY. Ura, J. M. Beierle, L. J. Leman, L. E. Orgel, M. R. Ghadiri, Science 2009, 325, 73–77;
- 3bH. D. Bean, F. A. L. Anet, I. R. Gould, N. V. Hud, Origins Life Evol. Biosphere 2006, 36, 39–63.
- 4J. M. Lehn, Chem. Soc. Rev. 2007, 36, 151–160.
- 5
- 5aA. Bugaut, K. Jantos, J. L. Wietor, R. Rodriguez, J. K. M. Sanders, S. Balasubramanian, Angew. Chem. 2008, 120, 2717–2720;
10.1002/ange.200705589 Google ScholarAngew. Chem. Int. Ed. 2008, 47, 2677–2680;
- 5bN. Sreenivasachary, D. T. Hickman, D. Sarazin, J. M. Lehn, Chem. Eur. J. 2006, 12, 8581–8588;
- 5cA. Bugaut, J. J. Toulme, B. Rayner, Angew. Chem. 2004, 116, 3206–3209;
10.1002/ange.200454041 Google ScholarAngew. Chem. Int. Ed. 2004, 43, 3144–3147;
- 5dS. Lena, G. Brancolini, G. Gottarelli, P. Mariani, S. Masiero, A. Venturini, V. Palermo, O. Pandoli, S. Pieraccini, P. Samori, G. P. Spada, Chem. Eur. J. 2007, 13, 3757–3764;
- 5eD. T. Hickman, N. Sreenivasachary, J. M. Lehn, Helv. Chim. Acta 2008, 91, 1–20;
- 5fE. Buhler, N. Sreenivasachary, S. J. Candau, J. M. Lehn, J. Am. Chem. Soc. 2007, 129, 10058–10059;
- 5gN. Sreenivasachary, J. M. Lehn, Proc. Natl. Acad. Sci. USA 2005, 102, 5938–5943.
- 6
- 6aR. J. Jones, S. Swaminathan, J. F. Milligan, S. Wadwani, B. C. Froehler, M. D. Matteucci, J. Am. Chem. Soc. 1993, 115, 9816–9817;
- 6bZ. Y. J. Zhan, D. G. Lynn, J. Am. Chem. Soc. 1997, 119, 12420–12421;
- 6cZ. Y. Li, Z. Y. J. Zhang, R. Knipe, D. G. Lynn, J. Am. Chem. Soc. 2002, 124, 746–747.
- 7
- 7aN. Christinat, E. Croisier, R. Scopelliti, M. Cascella, U. Rothlisberger, K. Severin, Eur. J. Inorg. Chem. 2007, 5177–5181;
- 7bW. J. Niu, M. D. Smith, J. J. Lavigne, J. Am. Chem. Soc. 2006, 128, 16466–16467;
- 7cN. Iwasawa, H. Takahagi, J. Am. Chem. Soc. 2007, 129, 7754–7755;
- 7dN. Nishimura, K. Kobayashi, Angew. Chem. 2008, 120, 6351–6354;
10.1002/ange.200802293 Google ScholarAngew. Chem. Int. Ed. 2008, 47, 6255–6258;
- 7eD. Roy, J. N. Cambre, B. S. Sumerlin, Chem. Commun. 2008, 2477–2479.
- 8
- 8aK. Severin, Dalton Trans. 2009, 5254–5264;
- 8bN. Fujita, S. Shinkai, T. D. James, Chem. Asian J. 2008, 3, 1076–1091;
- 8cS. Jin, Y. Cheng, S. Reid, B. Wang, Med. Res. Rev. 2010, 30, 171–257;
- 8dD. Luvino, D. Gasparutto, S. Reynaud, M. Smietana, J.-J. Vasseur, Tetrahedron Lett. 2008, 49, 6075–6078;
- 8eD. Luvino, M. Smietana, J. J. Vasseur, Tetrahedron Lett. 2006, 47, 9253–9256.
- 9
- 9aR. Scorei, V. M. Cimpoiasu, Origins Life Evol. Biosphere 2006, 36, 1–11;
- 9bB. E. Prieur, C. R. Acad. Sci. 2001, 4, 667–670;
- 9cA. Ricardo, M. A. Carrigan, A. N. Olcott, S. A. Benner, Science 2004, 303, 196;
- 9dM. Sugiyama, Z. Hong, L. J. Whalen, W. A. Greenberg, C.-H. Wong, Adv. Synth. Catal. 2006, 348, 2555–2559;
- 9eS. A. Gabel, R. E. London, J. Biol. Inorg. Chem. 2008, 13, 207–217.
- 10
- 10aD. Luvino, C. Baraguey, M. Smietana, J. J. Vasseur, Chem. Commun. 2008, 2352–2354;
- 10bA. R. Martin, K. Mohanan, D. Luvino, N. Floquet, C. Baraguey, M. Smietana, J. J. Vasseur, Org. Biomol. Chem. 2009, 7, 4369–4377.
- 11J. Yan, G. Springsteen, S. Deeter, B. Wang, Tetrahedron 2004, 60, 11205–11209.
- 12
- 12aR. Badugu, J. R. Lakowicz, C. D. Geddes, Dyes Pigm. 2005, 64, 49–55;
- 12bE. Galbraith, T. D. James, Chem. Soc. Rev. 2010, 39, 3831–3842.
Citing Literature
This is the
German version
of Angewandte Chemie.
Note for articles published since 1962:
Do not cite this version alone.
Take me to the International Edition version with citable page numbers, DOI, and citation export.
We apologize for the inconvenience.