Entschlüsselung der Polymer-Interaktionen in Biokonjugaten unterschiedlicher Architektur durch zeitaufgelöste limitierte Proteolyse mit massenspektrometrischer Analyse
Abstract
Protein-Polymer Konjugate erlauben eine pharmakokinetische Optimierung von therapeutischen Proteinen. Allerdings sind die Interaktionen des Polymer- und des Proteinteils des Biokonjugates oftmals unbekannt, so dass eine Optimierung des molekularen Designs weitestgehend auf empirische Beobachtungen beschränkt ist. Dieser Artikel schließt diese analytische Lücke durch Kombination einer limitierten Proteolyse mit massenspektrometrischer Analyse (LiP-MS). Zur Bestätigung der LiP-MS wurden zunächst Interaktionen des IFN-α2a mit seinem Rezeptor (IFNAR2) bestimmt, die sich mit publizierten Strukturen des Komplexes deckten. Interaktionen konnten dann detailliert am Beispiel von Biokonjugaten des Interferon-α2a (IFN) mit Poly(ethylenglykol) (PEG) und PEG alternativen Polymeren beschrieben werden. Dazu wurde ein IFN-Biokonjugat zusammen mit einem „schweren“ 15N-IFN-Wildtyp (IFN-WT; interne Kontrolle) durch Trypsin verdaut und die Schnitterfolge am Biokonjugat mit denen am Wildtyp differentiell analysiert. Die Biokonjugate wurden mit 10 kDa PEG, linearem Poly(glycerol) (LPG) oder Poly(2-oxazolin) (POX)über Cyclooctinlinker (BCN oder DBCO) ortsspezifisch mit Azid-funktionalisiertem IFN-α2a synthetisiert. LiP-MS zeigte, dass die Interaktionsmuster der resultierenden Biokonjugate für PEG und LPG vergleichbar waren, nicht aber für POX, welches geringere Interaktionen mit dem Protein im Biokonjugat aufwies. Die mittels LiP-MS gefundenen Wechselwirkungsprofile wurden durch atomistische Molekulardynamiksimulationen bestätigt und lassen kurzlebige Interaktionen des Polymerteils mit dem Proteinteil vermuten (<50 ns), zeigen aber auch die Abhängigkeit der Wechselwirkungen von dem verwendeten Polymer. Zusammengefasst gelingt es mittels zeitaufgelöster LiP-MS-Analyse, ein detailliertes Wechselwirkungsprofil des Polymerteils und des Proteinteils in Biokonjugaten zu kartieren. Die gut zu implementierende LiP-MS Methode könnte dazu beitragen, die heute weitgehend noch empirischen Optimierungen von Biokonjugaten durch ein Hypothesen-getriebenes Vorgehen zu ergänzen.
Einleitung
Die Konjugation von Poly(ethylenglykol) (PEG) an therapeutische Proteine erfolgt regelmäßig zu deren pharmakokinetischer (PK) Optimierung. PEG ist ein nicht toxisches, hydrophiles Polymer mit nicht biologisch abbaubarem Rückgrat. Personen, die einer andauernden Exposition durch dieses Polymer ausgesetzt sind, z. B. durch Kosmetika, könnten möglicherweise ein erhöhtes Risiko haben, anti-PEG neutralisierende Antikörper zu entwickeln. Dann würden PEGylierte Therapeutika durch das Immunsystem erkannt, es käme zu einer raschen Clearance und vielleicht sogar zur Komplementaktivierung und Hypersensitivität.1 Das ist einer der Gründe, weshalb PEG-alternative Polymere in der Biomedizin erforscht werden – z. B. Poly(2-oxazoline) (POX),2 Polysarkosin (PSar),3 lineares Polyglycerol (LPG),4 zwitterionische Polymere,5 Poly(methylacrylate)6 oder Polypeptide.7
Die Entwicklung von Protein-Polymer-Konjugaten erfolgt derzeit weitestgehend empirisch, im Wesentlichen getrieben durch Überlegungen zur Wirksamkeit. Dieses Vorgehen bestimmt die Auswahl des Polymertyps, dessen Architektur, die Konjugationsschemie und die daraus resultierende Polymer-Linker-Struktur.6, 8 Wünschenswert wäre eine Ergänzung dieses empirischen Vorgehens durch Hypothesen-basierte chemisch/biochemische Entwicklung, zum Beispiel eine Maximierung der Wirksamkeit durch gezielte Verankerung eines rational ausgewählten Polymers auf der Proteinoberfläche.9 Ortsspezifische Biokonjugationsmethoden stehen dahingehend zur Verfügung,10 z. B. durch rekombinante Techniken die einen erweiterten genetischen Code nutzen. Damit können nichtproteinogene Aminosäuren an definierten Positionen eines Proteins positioniert werden, eine gentechnische Herstellung, die in verschiedenen Expressionssystemen erfolgreich ist – z. B. in Escherichia coli,11 Hefe12 und Säugetierzellen.13 So konnten wir bereits verschiedene Biokonjugate mit ortsspezifischer Polymer-Konjugation herstellen, u. a. Interleukin 4 (IL-4),14 IFN-α2a8, 15 und den Fibroblasten-Wachstumsfaktor 2 (FGF-2).16
Trotz der wachsenden Literatur zur Polymerchemie sowie synthetischer und abiotischer Routen für die Synthese neuartiger, maßgeschneiderter Protein-Polymer-Konjugate bleiben Fragen zur molekularen Aufklärung dieser Strukturen offen. Cattani et al. zeigte mit Kernspinresonanzspektroskopie (NMR) und Röntgenstrukturanalyse, dass das β-faltblattreiche Plastocyanin und eine an diesem Protein ortsspezifisch konjugierte PEG-Kette als unabhängige, also nicht oder wenig wechselwirkende Domänen nebeneinander vorliegen.17 Ähnliches beschreiben Pai und Mitarbeiter für mono-PEGyliertes Lysozym und humanen Wachstumsfaktor; die Polymerkette wurde als proteinunabhängiger, hantelformartiger Random Coil beschrieben („Dumbbell modell“).18 Weitere Untersuchungen von Polymer-Protein-Interaktionen in Biokonjugaten nutzen NMR-Analysen des Proteinrückgrats am Beispiel von Galectin und Acrylaten.6 Neben diesen spektroskopischen Ansätzen, wurden MS-basierte Methoden erfolgreich zur Kartierung von Interaktionen und zugänglichen Epitopen auf Makromolekülen genutzt - das sogenannte „Protein Footprinting“. Diese Methoden werden üblicherweise genutzt, um den Zugang kleinerer Moleküle auf die Oberfläche eines Proteins zu analysieren, wie D2 (HDX-MS), OH-Radikale (Hydroxylradikal-Protein Footprinting (HRPF)/„Higher Order Structure“ (HOS)) oder chemische Crosslinker (X–Link).19 Durch „Bottom-Up“-MS erfolgt dann die Kartierung für die Bereiche der Interaktion zwischen Protein und Polymer, die für die kleinen Moleküle zugänglich sind.20 „Top-Down“-Proteomik liefert Informationen zu strukturellen Informationen aus der Gasphase. Dabei werden intakte (d. h. unverdaute) Komplexe mittels Elektrosprayionisations-MS (ESI-MS) ionisiert. Die Komplexe werden dann in der Gasphase fragmentiert, bspw. mittels Kollision (kollisionsinduzierte Dissoziation, Kollisionen höherer Energien), ladungsbasierter Fragmentierung (Elektronentransfer-Dissoziation, „Electron Capture“-Dissoziation) oder Laserbestrahlung (z. B. UV-basierte Photodissoziation). Aus den resultierenden Fragmenten werden Bereiche mit erhöhter oder erniedrigter Stabilität abgeleitet.19a
Eine qualitative massenspektrometrische Analyse nach limitierter Proteolyse (LiP-MS) nutzen wir bereits zur Beschreibung von Polymer-Protein-Interaktionen auf molekularer Ebene für verschiedene IFN-Biokonjugate.8 PEGyliertes IFN ist eines der ersten von der FDA zugelassenen Biokonjugate zur Therapie von Hepatitis C Erkrankungen.21 Die damaligen LiP-MS Experiment waren qualitativ und erlaubten bereits eine Charakterisierung der Kinetik des Konjugat-Verdaus durch Proteinase K allerdings mit Einschränkungen, so dass wir damals Interaktionsbereiche zwischen dem Protein und dem Polymer im Biokonjugat nicht quantitativ beschreiben konnten.8 Die nun folgende Studie hat das Ziel, diese Lücke zu schließen. Es gelang, intramolekulare Polymer-IFN-Interaktionen in 14N-Polymer-IFN-Konjugaten mittels 15N-markiertem IFN-WT als internen Standard exakt über die Zeit zu quantifizieren und so präzise Kartierungen der Wechselwirkungsprofile zwischen Polymer- und Proteinteil in den Biokonjugaten durchzuführen.
Ergebnisse und Diskussion
Synthese ortsspezifisch konjugierter IFN-Polymer-Konjugate
Die rekombinante Herstellung des humanen, IFN-α2a erfolgte mit der nichtproteinogenen Aminosäure N6-((2-Azidoethoxy)carbonyl)-L–Lysin (NAEK) mittels Amber Stop-Codon Technologie, welche ein Lysin des Wildtyps an Position K31 ersetzte (Abbildung 1, S1).8, 15a Position K31 wurde dann mit BCN oder DBCO modifiziertem Polyethylenglykol, Poly(2-oxazolin) oder Polyglycerol umgesetzt, wodurch Konjugate mit äquivalenter Bioaktivität in vitro und in vivo erhalten wurden.8, 15a In dieser Arbeit haben wir so zur quantitativen LiP-MS-basierten Strukturanalyse sechs verschiedene IFN-Biokonjugate synthethisiert: PEG (10 kDa), LPG (10 kDa oder 11.7 kDa) und POX (PetOx; 7.8 kDa oder 8.6 kDa), jeweils konjugiert mittels kupferfreier Azid-Alkin-Cycloaddition unter Verwendung von am Polymer befindlichen Cyclooctinlinkern, DBCO und BCN. Die Reinheit der hergestellten Konjugate wurde mittels SDS-PAGE analysiert (Abbildung S1).
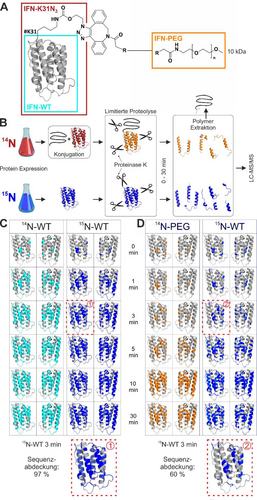
(A) Biokonjugationsschema für die ortsspezifische Konjugation an K31 zur Herstellung von 14N-IFN-DBCO-PEG. (B) Exemplarische Darstellung der LiP-MS-Analyse des IFN-DBCO-PEG-Konjugats in Kombination mit 15N-IFN-WT und in Gegenwart von Proteinase K. LiP-MS-Analyse von (C) 14N-IFN-WT in Kombination mit 15N-IFN-WT und (D) 14N-IFN-PEG zusammen mit 15N-IFN-WT. Der zeitliche Verlauf des Auftretens von Schnittstellen ist farblich hervorgehoben und der prozentuale Anteil der Sequenzabdeckung (rote Box) von 15N-IFN-WT ist nach einer Dauer von 3 min des Verdaus mit Proteinase K gezeigt. Die IFN-Tertiärstruktur entstammt der PDB-Datei 1ITF.
Quantifizierung des LiP-MS-basierten Proteinverdaus mit Proteinase K
LiP-MS wird zur Detektion struktureller Veränderungen in komplexen Proteomen z. B. als Antwort auf externe Stimuli verwendet.22 Hierzu wird zumeist die unspezifische Protease Proteinase K genutzt.23 Die Anwendbarkeit dieser Protease für IFN-Biokonjugate wurde in dieser Studie mittels LiP-MS getestet. Als Voraussetzung für eine vergleichende Quantifizierung wurde dem Proteinverdau ein interner Standard (IFN-WT) zugegeben, der 15N-markiert war (Abbildung 1A).
15N-IFN-WT wurde rekombinant durch metabolische Markierung mit Ammonium-15N-Chlorid-Substitution hergestellt und das aufgereinigte Protein wurde über Natriumdodecylsulfat-Polyacrylamid-Gelelektrophorese charakterisiert (SDS-PAGE), wobei es eine vergleichbare Auftrennung zum IFN-WT (14N-IFN-WT) zeigte (Abbildung S1). Die Markierungseffizienz in 15N-IFN-WT, quantifiziert mittels MS, war 99.6 % (Tabelle S1).
Zunächst wurde der Verdau durch Proteinase K von 14N-IFN-WT und seinem 15N-Isotop in einem 1 : 1-Verhältnis durchgeführt (Abbildung 1B). Wie erwartet ergab sich ein konsistentes Bild zwischen 14N- und 15N-IFN-WT bezüglich des zeitlichen und quantitativen Verlaufs detektierter Peptide (Abbildung 1C, Tabelle S2). Jede Peptidvariante wurde der 15N-markierten IFN-Struktur zugeordnet, was zu einer Sequenzabdeckung von über 90 % nach 3 min Verdau durch Proteinase K führte (Abbildung 1C). Das Verhältnis des Verdaus in Helices und Schleifen war über die Zeit vergleichbar zwischen beiden IFN-Varianten (Abbildung S2). Basierend auf diesen konsistenten Ergebnissen nutzen wir nachfolgend 15N-IFN-WT als internen Standard. Danach verdauten wir 14N-IFN-DBCO-PEG parallel mit 15N-IFN-WT mit Proteinase K für 30 min und analysierten die resultierenden Peptide (Abbildung 1D). Die Kartierung des generierten Peptid-Pools des internen Standards 15N-IFN-WT auf der 3D-Struktur von IFN zeigte eine reduzierte Sequenzabdeckung von 60 % nach 3 min Verdau mit Proteinase K. Ebenfalls nahm die Geschwindigkeit des Verdaus ab (~90 % nach 10 min), verglichen mit dem Verdau des unkonjugierten 14N-IFN-WT (~90 % nach 1 min) (Tabelle S2). Das durch 15N-IFN-WT gewonnene LiP-MS-Profil deutet auf eine durch PEG reduzierte Proteinase K-Aktivität hin. Der Mechanismus der Inhibition hängt offenbar nicht mit der Konjugation des Polymers am IFN zusammen, sondern könnte auf die zunehmende Konzentration löslichen PEGs während des proteolytischen Verdaus zurückzuführen sein. Ähnliche Befunde des hemmenden Effektes von PEG auf die Aktivität von Proteasen wurden in der Vergangenheit beobachtet.24 Obwohl Proteinase K zuvor für die LiP-basierte Strukturanalyse verwendet wurde (bspw. für apo-Myoglobin25) erwies sich Proteinase K für unsere strukturelle LiP-Analyse von PEGylierten Biokonjugaten durch die mögliche Polymerinteraktion als ungünstig.
Fingerprinting molekularer Interaktionen zwischen IFN und seinem hochaffinen Rezeptor (IFNAR2) mittels trypsinbasierter LiP-MS
IFN ist unter physiologischen Bedingungen geladen (pI=6.8) und hat 20 über seine Proteinoberfläche gleichmäßig verteilte positiv geladene Aminosäuren (R und K, Abbildung 2A). Daher ist das Protein für einen Trypsin basierten LiP-MS-Verdau geeignet. Die Aktivität von Trypsin wird durch PEG in PEGylierten Proteinen nicht beeinflusst26 und die Konformation des IFN wird durch Bindung an IFNAR2 nicht beeinträchtigt27 – beides ideale Voraussetzungen für unseren 15N-IFN-WT-basierten Quantifizierungsansatz (Abbildung S3). Wir etablierten LiP-MS, indem wir die spezifischen Interaktionen von IFN mit seinem endogenen hoch-affinen Rezeptor IFNAR2 als Ausgangspunkt weiterer Experimente wählten. Durch Trypsinverdau und nachfolgender MS-Analyse gelangten wir zu einem Interaktionsprofil des IFN-Rezeptorkomplexes, welches mit einer publizierten 1H NMR -Struktur abgeglichen wurde.27
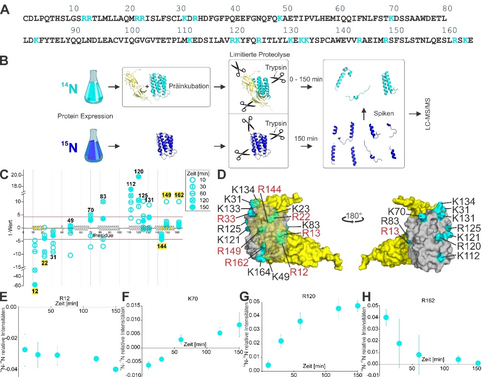
(A) Aminosäuresequenz von IFN im Buchstabencode. Trypsin-Schnittstellen (K und R) sind in cyan gefärbt. (B) Schematische Abbildung des Trypsin basierten LiP-MS-Ansatzes zur Analyse von IFN-Interaktionen mit IFNAR2. (C) t-Statistiken aus gepaarten t-Tests für ausgewählte IFN-Schnittstellen für den Verdau von 14N-IFN-WT (10, 30, 60, 120 und 150 min) mit Trypsin zusammen mit IFNAR2, verglichen mit dem Verdau von 15N-FN-WT für 150 min. Die Sekundärstruktur ist auf der x-Achse skizziert, Aminosäuren im Bereich der Rezeptor-Bindestelle sind dort gelb markiert. (D) Struktur des Komplexes von IFN (grau) mit dem extrazellulären Part des IFNAR2 (gelb, PDB: 2LAG27). Mögliche Trypsin-Schnittstellen sind in cyan gefärbt, Schnittstellen in der Nähe des Rezeptors zudem rot gefärbt. Differenzen zwischen den relativen Intensitäten von 14N- und 15N-Peptiden über die Zeit hinweg wurden für verschiedene Positionen beobachtet: (E) R12, (F) K70, (G) R120 und (H) R162. Die Ergebnisse sind als Mittelwerte mit Standardabweichungen dargestellt (n=3).
Die Kinetik des Auftretens tryptischer Peptide des 14N-IFN-WT-IFNAR2-Komplexes wurde in einem molaren 1 : 5-Verhältnis untersucht und nach 10, 30, 60, 120 und 150 min analysiert. Dann wurden die Peptidfragmente aus einem Trypsinverdau von 15N-IFN-WT nach 150 min jeder Probe des 14N-IFN-WT-IFNAR2-Komplexes als interner Standard hinzugefügt und eine MS-Analyse durchgeführt (Abbildung 2B). In diesem Falle entschieden wir uns die Peptidfragmente nach 150 Minuten als interne Kontrolle und nicht für eine gleichzeitige Inkubation von 15N-IFN-WT mit dem IFNAR2 und intaktem 15N-IFN-WT, da es dadurch zu einem Ligandenaustausch gekommen, und die Analyse unmöglich gewesen wäre (diese Einschränkung galt nicht für die Polymer-IFN-Biokonjugate; siehe unten). Die mittlere Anzahl identifizierter Peptide, das mittlere totale extrahierte Ionenchromatogramm (XIC) der 14N- und 15N-Peptide, sowie die Intensitäten der Peptide mit ungeschnittener Trypsin-Schnittstelle über die Zeit hinweg sind in Abbildung S4–S6 dargestellt. Die Quantifizierung von Schnittstellen des 14N-IFN-WT-IFNAR2-Rezeptor-Komplexes, beschrieben durch t-Statistik (Signifikanzniveau bei α=0.05, entsprechend einem t-Wert von 4.303), zeigte auf, dass die Proteolyse in Abhängigkeit der lokalen Proteinstruktur erfolgte (Abbildung 2C, S7). Die N- und C-terminalen α-Helices (Aminosäuren 10–21 und 137–157) von IFN sind im IFNAR2 Komplex geschützt27, 28 und unsere Experimente bestätigten dies (Positionen R12 und R22). Die negativen t-Werte an diesen beiden Positionen zeigten, dass Trypsin nicht an der IFN-IFNAR2 Interaktionsfläche schneiden konnte (Abbildung 2D). Diese Beobachtung wird durch die unveränderte Schneidrate an R12 bestätigt (Abbildung 2E). Gleichzeitig blieben die Intensitäten (und t-Werte) an der Schnittstelle K70 über das analysierte Zeitintervall hinweg ähnlich ausgeprägt (Abbildung 2F). Wir vermuten, dass die Umgebung der K70-Schnittstelle weniger zugänglich für Trypsin ist. Einige Schnittstellen (z. B. R120), die nicht an der Bindung mit IFNAR2 beteiligt sind, wurden zugänglicher für den Trypsinverdau (Abbildung 2C, 2G). Ein ähnliches Muster wurde für die benachbarte Schnittstelle K112 beobachtet (Abbildung 2C). Beide Stellen sind in der α-Helix D lokalisiert (Aminosäuren 110–132). R162 im Bereich der C-terminalen Helix wurde ebenfalls schnell geschnitten, erkennbar an einem positiven t-Wert nach 10 min des Verdaus und einer Reduktion der Intensität über die Zeit hinweg (Abbildung 2H). Diese Schnittstelle ist direkt am Randbereich der IFN-IFNAR2 Interaktionsfläche lokalisiert (Abbildung 2D). Zusammengefasst konnten also interagierende und nicht interagierende Bereiche innerhalb des IFN-IFNAR2-Komplexes mithilfe unseres LiP-MS-Ansatzes erfolgreich abgegrenzt werden.
Fingerprinting von Polymer-Protein-Interaktionen in IFN-Polymer-Konjugaten
Nach der Bestätigung unseres LiP-MS-Ansatzes mittels des oben beschriebenen IFN-IFNAR2 Komplexes, kartierten wir nun die Interaktionen zwischen IFN und Polymeren in IFN-Polymer-Konjugaten. Dazu synthetisierten wir IFN-Konjugate mit verschiedenen Polymeren unterschiedlicher Hydrophilie (POX weniger hydrophil als PEG; LPG am hydrophilsten) und begannen mit drei Varianten ortsspezifisch konjugierter 14N-IFN-DBCO-Polymer-Konjugate zur strukturellen Analyse mittels trypsinbasierter LiP-MS (Abbildung 3A). Der interne Standard 15N-IFN-WT wurde nun von vornherein zum 14N-IFN-Polymer-Konjugat in einem molaren 1 : 1-Verhältnis hinzugegeben (gemeinsamer Verdau des 15N-IFN-WT und 14N-IFN-Polymer-Konjugates) und erste Proben wurden nach 10 min gezogen, die letzten Proben nach 150 min (Abbildung 3B). Zusätzlich wurde die Mutante 14N-IFN-K31N3 nach 60 min und 150 min analysiert. Die mittlere Anzahl identifizierter Peptide, das Ionenchromatogramm (XIC) der 14N- und 15N-Peptide, sowie die Intensitäten der Peptide mit ungeschnittener Trypsin-Schnittstelle sind für jedes DBCO-Konjugat in den Hintergrundinformationen dargestellt (Abbildung S8–S13).
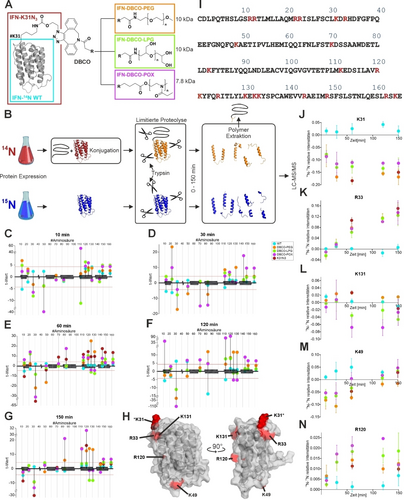
(A) Schematische Abbildung der 14N-IFN-DBCO-Polymer-Konjugate. (B) Darstellung des experimentellen Vorgehens bei der quantitativen LiP-MS-Analyse für 14N-IFN-Polymer-Konjugate unter Verwendung des Trypsin. t-Statistik von gepaarten t-Tests für ausgewählte Schnittstellen von IFN für verschiedene 14N-IFN-DBCO-Polymer-Konjugate nach (C) 10, (D) 30, (E) 60, (F) 120 und (G) 150 min des Verdaus. (H) Lokalisation von K31, R33, K131, K49 und R120 (rot gefärbt) des IFN (PDB: 1ITF). (I) Aminosäuresequenz des IFN im Buchstabencode. Trypsin-Schnittstellen (K und R) sind rot markiert. Differenzen zwischen den relativen Intensitäten zwischen 14N- und 15N-Peptiden über die Zeit sind für verschiedene Positionen gezeigt: (J) K31, (K) R33, (L) K131, (M) K49 und (N) R120. Resultate sind als Mittelwerte mit Standardabweichungen dargestellt (n=3).
Die Signifikanzen der quantifizierten Schnittstellen innerhalb der Sequenz jedes 14N-IFN-Polymer-Konjugats sind in Abbildung 3C–G als t-Statistik dargestellt (4.303; Signifikanzlevel α=0.05). Die p-Werte für die Schnittstellen sind in Abbildung S14 gezeigt. Wir wählten fünf signifikante Schnittstellen aus, um Ähnlichkeiten und Differenzen zwischen den Konjugaten über die Zeit hinweg zu illustrieren (Abbildung 3J–N). Zunächst analysierten wir die Konjugationsstelle K31, an der PEG, LPG, und POX an IFN gekoppelt wurde (Abbildung 3J). Wie erwartet war K31 nicht mehr für den Trypsinverdau der 14N-IFN-Polymer-Konjugate verfügbar, wohl aber im Falle des unmodifizierten 14N-IFN-WT. Durch den Austausch von K31 mit der nichtproteinogenen Aminosäure NAEK erhöhte sich der Trypsinverdau an Position R33. Dies kann dadurch begründet werden, dass der Verdau an dieser Schnittstelle nicht messbar war, wenn bereits eine Proteolyse an Position K31 erfolgt war. Die Modifikation von 14N-IFN-K31N3 mittels Polymerkonjugation hatte keinen Effekt auf den Trypsinverdau in der benachbarten Umgebung der Schleife (R33; Abbildung 3K). Diese Beobachtung, zeigt, dass die kovalent gebundenen Polymere hochflexibel sind und dadurch das Protein nicht einmal im Bereich der Konjugation vor dem proteolytischen Verdau schützen können.
Im Folgenden untersuchten wir den Verdau innerhalb verschiedener Umgebungen auf der Proteinoberfläche: K131 (Helix) und K49 (Schleife) (Abbildung 3L, Abbildung 3M).
Beide Lysine sind durch Wasserstoffbrückenbindungen stabilisiert.27 Die Quantifizierung des Verdaus an der Schnittstelle K131 (Helix) zeigte eine Reduktion der Intensität ab 30 min für 14N-IFN-DBCO-LPG und 14N-IFN-DBCO-POX, nicht jedoch für 14N-IFN-DBCO-PEG.
Ein deutlicher Rückgang des Trypsinverdaus an der Schnittstelle K49 (Schleife) wurde in allen untersuchten DBCO-Konjugaten ab 60 min gefunden (Abbildung 3M). Der Effekt war am deutlichsten beim 14N-IFN-DBCO-PEG, was mit der bevorzugten Interaktion von PEG mit solvenszugänglichen Lysinen und Argininen aus vorherigen Modellierungen übereinstimmte.8 Schließlich untersuchten wir den Verdau an Stelle R120 (Helix). Der Verdau war erhöht für alle 14N-IFN-DBCO-Polymer-Konjugate, ersichtlich aus den 14N-15N-Intensitäten über die Zeit (Abbildung 3N). Der Effekt war am stärksten im Falle von 14N-IFN-DBCO-POX und weniger intensiv für 14N-IFN-DBCO-PEG, mit signifikanten Unterschieden für alle IFN-Polymer-Konjugate nach 120 min. Dies deutet auf einen destabilisierenden Effekt der Polymer-Konjugation für all DBCO-Varianten in dieser Schleifenregion (K49) hin, in der absteigenden Reihenfolge POX, LPG und PEG.
Um globale Effekte der Polymer-Konjugation zu charakterisieren, wurde das Verhältnis tryptischer Peptide in Helices und Schleifen für alle 14N-IFN-DBCO-Polymer-Konjugate und 14N-IFN-K31N3 und 15N-IFN-WT zeitabhängig quantifiziert (Abbildung S15, S16). Im Gegensatz zur Proteolyse von 15N-IFN-WT führte der Verdau aller 14N-IFN-DBCO Polymer-Konjugate sowie der Mutante 14N-IFN-K31N3 zu einer vermehrten Anzahl tryptischer Peptide in helikalen Bereichen zu frühen Messzeitpunkten (10, 30 min) und zu solchen aus unstrukturierten Schleifen zu späteren Zeitpunkten (120, 150 min).
DBCO wird in der Click-Chemie für kupferfreie Azid-Alkin-Cycloreaktionen genutzt. Der Linker erhöht die Lipophilie und hat den Nachteil, dass regioisomere Addukte auftreten.29 Um den Effekt zu beurteilen, wiederholten wir die LiP-MS-Analyse mit Biokonjugaten, welche anstelle des DBCO-Linkers mit einem weniger hydrophoben BCN-Linker synthetisiert wurden, welcher zudem aufgrund seiner planaren Symmetrie keine Regioisomere ausbildet (Abbildung 4A). Die Intensität der identifizierten Peptide und das Ionenchromatogramm (XIC) der 14N- und 15N-Peptide sind für alle BCN-Konjugate in den Hintergrundinformationen aufgelistet (Abbildung S17–S19). Jede Trypsin-Schnittstelle wurde zunächst auf signifikante Unterschiede zwischen Biokonjugat und internem Standard für die Zeitpunkte 60 und 150 min überprüft und die zugehörigen t-Werte bestimmt (4.303; Signifikanzlevel α=0.05; Abbildungen 4B, C; p-Werte in Abbildung S20). Der prozentuale Anteil des Trypsinverdaus in Schleifen und Helices ist in Abbildung S21 dargestellt. Wie auch bei den DBCO-Konjugaten, stand die modifizierte Konjugationsstelle K31 nicht mehr für einen Verdau zur Verfügung, da an dieser Stelle die unnatürliche Aminosäure das Lysin ersetzt (Abbildung 4D). In der Folge konnte ebenso wie beim DBCO-Linker nun auch für den BCN-Linker beobachtet werden, dass die Proteolyse an der benachbarten Position R33 in allen BCN-Polymer-Konjugaten und der Mutante 14N-IFN-K31N3 im Vergleich zum WT erhöht war (Abbildung 4B, C, E). Unterschiede zwischen den DBCO- und BCN-Konjugaten wurden an K49 and R120 nach Trypsinverdau gefunden, nicht aber zwischen den verschiedenen Polymeren, die zur Herstellung der BCN-Konjugate verwendet wurden. Das lässt vermuten, dass für alle Polymere ähnliche Dynamiken der Interaktion mit der Proteinoberfläche in dieser Region vorliegen könnten. Wir vermuten, dass die Konformationen der 14N-IFN-BCN-Polymer-Konjugate einer homogeneren Verteilung als im Falle der DBCO-Konjugate folgen.
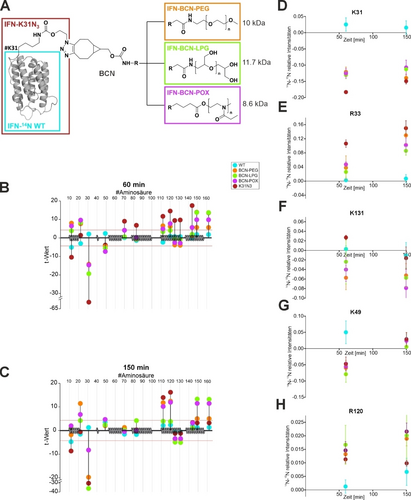
(A) Schematische Abbildung von 14N-IFN-BCN-Polymer-Konjugaten. t-Statistiken von gepaarten t-Tests ausgewählter Schnittstellen von IFN (Sekundärstruktur auf der x-Achse) für verschiedene 14N-IFN-BCN-Polymer-Konjugate nach (B) 60 und (C) 150 min des Trypsinverdaus. Differenzen der relativen Intensitäten über die Zeit hinweg zwischen 14N- und 15N-Peptiden in Bezug auf ausgewählte Positionen: (D) K31, (E) R33, (F) K131, (G) K49 und (H) R120. Resultate sind als Mittelwerte mit Standardabweichungen dargestellt (n=3).
In vorhergehenden Studien beobachteten wir geringe Unterschiede in der Schmelztemperatur Tm der DBCO/BCN-Konjugate mit POX im Vergleich zu PEG/LPG (Δ 2.7 °C), jedoch nicht bzgl. deren Aktivität.8 Im hier verwendeten LiP-MS-Ansatz konnten wir diese Beobachtungen nicht bestätigen, da dieser neue Datensatz keine strukturellen Unterschiede zwischen den IFN-Polymer-Konjugaten vermuten ließ, die die in vorhergehenden Studien gefundenen Unterschiede der Tm erklären könnten. Betrachtet man diese Datensätze nun gemeinsam, kann vermutet werden, dass die unterschiedlichen Tm als Folge rascher und lediglich vorübergehend vorliegender aber eben auch destabilisierend wirkender Polymer-IFN-Interaktionen aufzufassen sind und nicht auf spezifische, anhaltende Bindungen zwischen dem IFN und den Polymeren zurückzuführen sind, die mittels LiP-MS gefunden worden wären (z. B. Schnittstelle R120; 14N-IFN-DBCO-POX, see above).
Im Folgenden untersuchten wir, ob zwischen den verwendeten Polymeren und Linkerstrukturen signifikante Unterschiede bestehen (Abbildung 5A, B). Auffallend verhielten sich die POX-Konjugate, bei denen die meisten Schnittstellen mit Trypsin im Vergleich zu LPG- oder PEG-Konjugaten gefunden wurden. Das lässt vermuten, dass die hydrophilen Polymere PEG und LPG aber nicht POX ein ähnliches Wechselwirkungsmuster mit der IFN-Oberfläche haben. Diese Ergebnisse aus den LiP-MS-Experimenten wurden weiterhin mit atomistischen Molekulardynamiksimulationen der IFN-DBCO-Polymer-Konjugate untersucht (Abbildung 5C).8 Wir orientierten uns dabei an Studien anderer, die die maximale Aufenthaltszeit von PEG mit einzelnen Aminosäuren auf der Oberfläche des Albumins untersuchten.30 Insgesamt lassen diese Modelle vermuten, dass die Polymer-IFN Interaktionen lediglich vorübergehend sind und auch von kurzer Dauer, da diese zumeist nicht länger als 50 ns andauern. Auch hier fällt das POX-Konjugat im Vergleich zu den hydrophileren Polymeren aus der Reihe, mit insgesamt kürzeren Aufenthaltszeiten an der Proteinoberfläche. Die Ergebnisse dieser in silico Modellierung sind im Einklang mit der oben berichteten Beobachtung aus den LiP-MS Messungen, die ebenfalls eine bessere Umsetzung der POX-Konjugate durch Trypsin vermuten lassen (Abbildung 5A). Außerdem konnten wir feststellen, dass die Polymere mit unterschiedlichen Aminosäuren auf der Proteinoberfläche bevorzugt interagierten. Während PEG Lysine und Arginine bevorzugte, waren dies Aspartat und Glutamat im Falle des LPG, wahrscheinlich zurückzuführen auf die Wasserstoffbrücken, die sich zwischen den Hydroxylgruppen von LPG zu Aspartaten und Glutamaten ausbilden können. Die Carbonylgruppen des POX interagierten mit Aminosäuren, die als Donor von Wasserstoffbrücken fungieren, insbesondere das hydrophobe Tryptophan (Abbildung 5C). Auch die Protein-Polymer-Interaktionsenergien könnten auf eine stärkere Wechselwirkung der hydrophilen Polymere LPG und PEG mit dem IFN hinweisen, als das beim POX der Fall ist (Abbildung 5D). Insgesamt aber bedeckt PEG die größte Proteinoberfläche (Abbildung 5E).
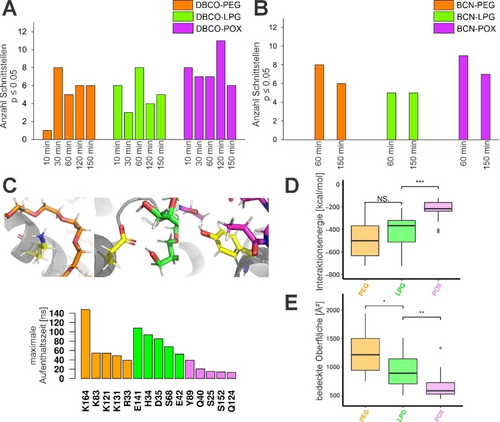
Anzahl an Trypsin-Schnittstellen mit unterschiedlichen Intensitäten (p≤0.05), dargestellt für (A) 14N-IFN-DBCO-Polymer-Konjugate und (B) 14N-IFN-BCN-Polymer-Konjugate im Vergleich zu 15N-IFN-WT. (C) Momentaufnahmen der zuvor durchgeführten Molekulardynamiksimulationen von IFN-DBCO-Polymer-Konjugaten (10 Replika für jeweils 150 ns). Charakteristische Protein-Polymer-Wechselwirkungen mit verschiedenen Aminosäuren sind dargestellt. Unter der Darstellung sind die 5 Aminosäuren gezeigt, an denen die längste maximale Aufenthaltszeiten für die Polymere berechnet wurde, basierend auf der Bestimmung konsekutiver Momentaufnahmen mit Polymeratomen innerhalb eines 5 Å-Abstands zur jeweiligen Aminosäure. (D) Totale Interaktionsenergie zwischen dem Polymer und dem Protein (ausgenommen K31 mit Linker), berechnet mittels lie-Funktion von AmberTools. (E) Totale Connolly-Proteinoberfläche, die durch das jeweilige Polymer bedeckt ist, berechnet mittels molsurf-Funktion von AmberTools. Die Signifikanzlevel in (D) und (E) wurden mit einem Wilcoxon-Test berechnet (p-Werte: ***=<0.001, **=<0.01, *=<0.05, NS.=nicht signifikant).
Abschließend versuchten wir, die unterschiedlichen Polymerinteraktionen vor allem des POX im Vergleich zum PEG oder LPG mit Bindungsdaten des IFN-IFNAR2 Rezeptors in Bezug zu setzen. Die SPR-Sensorgramme bestätigten die hohe Affinität des IFN zum IFNAR2 Rezeptor, genauer gesagt dessen Ektodomäne, die für diese Versuche verwendet wurde und im nanomolaren Bereich bindet (KD von ca. 7–8 nM; Abbildung S22, Tabelle S3). Die Ligand-Rezeptor-Ektodomänen-Interaktion folgte einer raschen Assoziations-Dissoziations-Kinetik, mit einer Assoziationsgeschwindigkeitskonstanten kon von (2.5±0.5)×106 M−1 s−1 und einer Dissoziationsgeschwindigkeitskonstanten koff von (1.8±0.1)×10−2 s−1. Die Dissoziationsgleichgewichtskonstante (KD=koff/kon), wurde durch eine Analyse des konzentrationsabhängigen Bindungssignals (Rmax-Werte) bestätigt – hierbei wurden ähnliche Bindungsaffinitäten von 7–9 nM erhalten. Die Polymerkonjugate des IFN hatten grundsätzlich bis zu 2-fach reduzierte Bindungsaffinitäten. Diese Reduktion war Folge einer reduzierten Assoziationskinetik; die Dissoziationskinetik war bei den Polymerkonjugaten im Vergleich zum unkonjugierten Wildtyp 1.5-fach erhöht. Die langsamere Assoziation der Polymer-IFN-Konjugate im Vergleich zum unmodifizierten Wildtyp ist im vorliegenden Falle vermutlich ebenfalls die Folge eines größeren hydrodynamischen Radius, wie an anderer Stelle berichtet.15 Dieser größere hydrodynamische Radius sollte die Diffusion des Polymer-IFN Konjugats verlangsamen. Wir interpretieren diese Daten in der Zusammenschau so, dass die Veränderungen der Bindungskinetik der Konjugate im Vergleich zum unmodifizierten Wildtyp mit dem Rezeptor vermutlich Folge der veränderten Moleküldimensionen sind, nicht aber ein verändertes Interaktionsverhalten, beispielsweise durch ein Abdecken der Interaktionsfläche des IFN durch ein Polymer, widerspiegeln.
Im Ergebnis ermöglichen derartige LiP-MS-Untersuchungen einen spannenden Einblick in das dynamische Wechselspiel von Protein-Polymer-Biokonjugaten. Der Ansatz erlaubt es, globale Effekte der Polymer-Protein-Interaktionen in helikalen und unstrukturierten Schleifenbereichen abzugrenzen und spezifische Effekte einzelner Polymerklassen, hier untersucht für PEG/LPG/POX, zu verstehen. Allgemein sind diese Interaktionen der Polymere mit der Proteinoberfläche kurzlebig und schwach und kontrastieren dahingehend die spezifischen Protein-Protein-Interaktionen, wie wir sie für den IFN/IFNAR2-Komplex gefunden haben (siehe oben).
Zukünftige Studien sollten einige Limitierungen unserer Experimente aufgreifen. So nutzen wir beispielsweise lediglich Trypsin für die LiP-MS-Untersuchungen. Es gibt aber keinen Grund, wieso man diese Protokolle nicht auf andere spezifische Proteasen ausweiten sollte, beispielsweise in der Absicht, vor allem die Interaktionen der hydrophoberen Proteinoberflächen mit Polymeren zu verstehen. Solche erweiterten „Protease-Bibliotheken“ könnten besonders für hydrophobere Polymere hilfreich sein.
Zusammenfassung
Diese Arbeit beschreibt eine Methode zur Bestimmung von Interaktionen zwischen kovalent verbundenen Polymeren und Proteinwirkstoffen. Als Beispiel dienten die Polymere, PEG, LPG und POX, sowie das Protein IFN-α2a. Diese Methode, LiP-MS, liefert quantitative und kinetische Informationen über die Interaktionen zwischen Polymer und Proteinwirkstoff innerhalb eines Biokonjugates. Dies gelang durch Verwendung von 15N-markiertem IFN-Wildtyp (IFN-WT) als internen Standard, welches in Gegenwart von Trypsin ebenso abgebaut wird, wie die IFN-Polymer Biokonjugate – allerdings deckt das Polymer Teile des IFN ab, so dass der Trypsinabbau in diesen Interaktionsbereichen des IFN und des Polymers langsamer oder gar nicht stattfindet. Durch zeitaufgelösten Vergleich der Trypsin-katalysiert entstehenden Peptidfragmente des 15N-markiertern IFN-Wildtyps mit den Peptdifragmenten aus den IFN-Polymer-Biokonjugaten (14N) kann mittels LC–MS/MS nun über die Zeit die Interaktion der Polymere mit dem Proteinwirkstoff beschrieben werden. Der experimentelle Ansatz wurde zunächst an Komplexen des IFN mit seinem Rezeptor überprüft, Trypsin konnte genau in den Bereichen deutlich weniger Fragmente erzeugen, von denen bekannt ist, dass hier die Protein-Protein-Interaktionen im IFN-IFNAR2-Komplex vorliegt. Die Methode wurde dann erweitert, um Polymer-Protein Wechselwirkungen in IFN-Biokonjugaten zu charakterisieren. Dazu wurden verschiedene Polymerfamilien und chemische Linkerstrukturen untersucht, insgesamt 6 Polymer-Protein Biokonjugate und mit der Absicht, die molekularen Interaktionen zwischen Polymer und Proteinwirkstoff exakt zu beschreiben. Hydrophiles PEG und LPG zeigten ähnliche Interaktionsprofile mit IFN-α2a im Biokonjugat, das hydrophobere POX wich deutlich ab, und zwar sowohl was die Anzahl der Interaktionen angeht, als auch den Ort auf der Proteinoberfläche. Die gefundenen Interaktionsprofile wurden mit Molekulardynamiksimulationen untermauert. Zusammengefasst liegt mit dem LiP-MS Aufbau nun eine quantitative Methode vor, mit der zeitaufgelöste Wechselwirkungsprofile zwischen Polymer und Proteinwirkstoff in einem Biokonjugat kartiert werden können. Die Methode ist in entsprechend ausgestatteten Laboren rasch etablierbar, so dass die weitestgehend empirische Optimierung von Biokonjugaten, wie sie derzeit noch größtenteils verbreitet ist, zukünftig durch ein rationales und Hypothesen-getriebenes Vorgehen ergänzt werden könnte.
Hintergrundinformationen
Zusätzliche Literatur findet sich in den Hintergrundinformationen.31
Danksagung
Wir danken Dr. Andreas Schlosser, Stephanie Lamer, Prof. Dr. Bettina Warscheid, Dr. Julian Bender und Johannes Zimmermann (Universität Würzburg) für ihre Unterstützung bei der Massenspektrometrie. Diese Studie wurde finanziert durch das Bundesministerium für Bildung und Forschung (BMBF) innerhalb der “BICRA”- (#16GW0334 K) und „Next-PEG“-Verbundprojekte zwischen der Freien Universität Berlin (FKZ: 13XP5049 A), der Julius-Maximillians-Universität Würzburg (FKZ: 13XP5049B) und der Friedrich-Schiller-Universität Jena (FKZ: 13XP5049 C). Open Access Veröffentlichung ermöglicht und organisiert durch Projekt DEAL.
Interessenkonflikt
Die Autoren erklären, dass keine Interessenkonflikte vorliegen.
Open Research
Data Availability Statement
Die Daten, die die Ergebnisse dieser Studie unterstützen, sind auf begründete Anfrage beim Autor erhältlich.