Untersuchung der Adressierbarkeit des Cysteinoms der Proteinkinasen durch Kovalente Fragmente
Abstract
Proteinkinasen sind wichtige Zielstrukturen für Arzneistoffe, jedoch wurden bisher nur für einen Bruchteil der mehr als 500 menschlichen Kinasen spezifische Hemmstoffe entwickelt. Eine große Herausforderung bei der Entwicklung von Inhibitoren für eng verwandte Kinasen ist die Selektivität. Im Gegensatz zu ihren nicht kovalenten Gegenstücken bieten kovalente Inhibitoren hierbei den Vorteil, dass sie selektiv auf strukturell ähnliche Kinasen abzielen können, indem sie spezifische Proteinseitenketten, insbesondere nicht konservierte Cysteine, modifizieren. Bisherige kovalente Fragment-Screens ergaben potente und selektive Verbindungen für einzelne Kinasen wie ERK1/2, jedoch wurde dieser Ansatz noch nicht auf einen breiteren Teil des Kinoms angewendet. Darüber hinaus wurden viele der potenziell zugänglichen Cysteinpositionen bisher noch nicht adressiert. Mit dieser Arbeit stellen wir einen allgemein anwendbaren Ansatz vor, um die Adressierbarkeit von Cysteinen in der Nähe der ATP-Bindetasche mit fragmentartigen kovalenten Inhibitoren zu untersuchen. Wir präsentieren die Entwicklung einer auf Kinasen fokussierten Fragmentbibliothek und ihr systematisches Screening gegen eine kuratierte Auswahl von 47 Kinasen mit 60 Cysteinen in der Nähe der ATP-Bindestellte. Dazu wurden LC/MS- und Differential Scanning Fluorimetry (DSF)-Assays verwendet, gefolgt von einer Hit-Validierung durch verschiedene ergänzende Techniken. Unsere Ergebnisse erweitern das Repertoire an adressierbaren Cysteinen in der Enzymfamilie der Proteinkinasen, geben Einblick in einzigartige Bindungsmodi, die anhand von Kristallstrukturen identifiziert wurden und liefern isoformspezifische Hit-Moleküle mit vielversprechenden Profilen als Ausgangspunkte für die Entwicklung hochwirksamer und selektiver kovalenter Hemmstoffe.
Einleitung
Proteinkinasen spielen eine wesentliche Rolle bei der Regulierung praktisch aller zellulärer Prozesse, einschließlich der Zellproliferation, Apoptose, subzellulärer Translokation und des Stoffwechsels. Eine fehlregulierte Kinasefunktion ist eine der Hauptursachen für Krankheiten.1 Aus diesem Grund gehören Proteinkinasen zu den erfolgreichsten Zielstrukturen für Arzneimittel. Über 80 niedermolekulare Kinaseinhibitoren (small molecule kinase inhibitors; SMKI) wurden von der US-amerikanischen Food and Drug Administration (FDA) sowie von europäischen und asiatischen Behörden bisher zugelassen.2, 3 Trotz der Schlüsselfunktionen von Proteinkinasen in der gesunden Physiologie wie auch bei Krankheit ist die Mehrzahl dieser Enzyme noch wenig erforscht. So decken zugelassene Kinaseinhibitoren und SMKI, die klinisch untersucht werden, nur etwa 30 % des menschlichen „Kinoms” ab.2 Gleichzeitig bleibt ein erheblicher Teil des Kinoms noch völlig unerschlossen.4
Hochselektive Inhibitoren, die als chemische Sonden (sog. „Chemical Probes“) eingesetzt werden, haben enorm zur Identifizierung und Validierung neuer Kinase-Targets beigetragen. Ein großes Hindernis für ihre Entwicklung ist jedoch der Mangel an effizienten Strategien zur Erzielung von Selektivität, insbesondere gegenüber eng verwandten Kinasen. Ein sehr erfolgreicher Ansatz zur Erhöhung der Selektivität ist die Kombination eines geeigneten reversiblen Bindungselements mit einer reaktiven Gruppe, typischerweise einem schwachen Elektrophil, das als „Warhead“ bezeichnet wird, um eine wenig konservierte nukleophile Aminosäure wie Cystein kovalent zu adressieren.5, 6 Die Bildung der kovalenten Bindung erhöht die Wirksamkeit und Selektivität selbst zwischen hoch konservierten Kinasen erheblich, wenn der adressierte Cysteinrest nur in einem Familienmitglied vorkommt. So wurden beispielsweise selektive JAK3-Inhibitoren auf der Grundlage eines pan-JAK-Inhibitors entwickelt, indem ein Cysteinrest adressiert wurde, der nur in der JAK3-Isoform vorkommt.7
Trotz der jüngsten Fortschritte bei der Entwicklung kovalenter Kinaseinhibitoren, die auf das „katalytische Lysin“ abzielen,8 sind Cysteine aufgrund ihrer einzigartigen thiolhaltigen Seitenkette, welche die höchste Nukleophilie aller proteinogenen Aminosäuren aufweist, nach wie vor das häufigste Ziel für kovalente Inhibitoren.5, 6 Aufgrund dieser Eigenschaft können Cysteine mit relativ schwachen Elektrophilen adressiert werden, was die Wahrscheinlichkeit einer unspezifischen Reaktivität der Inhibitoren verringert. Im Gegensatz zu Serin und Threonin machen die höhere intrinsische Nukleophilie und der niedrigere pKs-Wert der Cystein-Thiolgruppe eine starke Aktivierung der Seitenkette durch die Proteinmikroumgebung, z. B. in katalytischen Di- oder Triaden, für die kovalente Adressierung nicht notwendig. Ein weiterer Vorteil ist, dass Cystein eine relativ seltene Aminosäure darstellt und für die Katalyse in Proteinkinasen nicht erforderlich ist. Daher ist die Ausrichtung auf das „Cysteinom“ der Proteinkinasen eine gut geeignete Strategie für die Entwicklung selektiver kovalenter SMKIs.9 Ungefähr 215 der über 518 Proteinkinasen weisen potenziell zugängliche Cysteine in oder nahe der konservierten ATP-Bindungstasche auf. Nach bisherigem Kenntnistand sind diese Cysteine auf mindestens 18 verschiedene räumliche Positionen verteilt.10 Bislang wurden jedoch nur etwa 70 dieser Kinasen kovalent adressiert,11 wobei das Ziel zumeist einige wenige Cysteinpositionen waren. So wurde beispielsweise das der αD-Helix vorgelagerte Cystein der vorderen Tasche (F2), das in 11 Proteinkinasen (EGFR, HER2, HER4, BTK, ITK, BMX, TXK, TEC, BLK, JAK3 und MKK7) vorkommt, überproportional häufig adressiert. Zehn von elf der aktuell von der FDA zugelassenen kovalenten SMKIs greifen dieses Cystein an. Im Gegensatz dazu wurden einige andere Positionen noch überhaupt nicht adressiert, wie z. B. die P-Loop-Cysteine von CHEK2 (CHK2) und den CSNK1G-Isoformen. Die Adressierbarkeit von Cysteinresten kann durch limitierte sterische Zugänglichkeit und eine Proteinumgebung eingeschränkt werden, in der die Thiolatform der Seitenkette ungünstig ist (d. h. durch Erhöhung des Thiol pKs-Werts). Andererseits können sich zugängliche Cysteine mit niedrigem pKs-Wert „hyperreaktiv“ verhalten.12 Zwar wurden in silico-Methoden entwickelt, um den pKs-Wert von Cysteinen als Parameter für die Reaktivität abzuschätzen,13 doch sind solche Berechnungen fehleranfällig, und experimentelles Vorwissen über die Zugänglichkeit und Adressierbarkeit von Cysteinen ist zum Start eines Entwicklungsprogramms für kovalente Inhibitoren äußerst wertvoll. Während so genannte „electrophile first“-Ansätze, bei denen typischerweise sehr kleine und hochreaktive Fragmente verwendet werden, zur Bewertung der allgemeinen Adressierbarkeit von Cysteinen und anderen Aminosäuren verwendet werden können,14 sind die erhaltenen Treffer oft schwer zu optimieren. Neben den Schwierigkeiten beim Ausbau des Fragment-Hits ist auch der Wechsel zu weniger reaktiven Elektrophilen oft schwer zu bewerkstelligen. Umgekehrt gehen „ligand first“-Ansätze in der Regel von elaborierten (und damit recht selektiven) reversiblen Bindungselementen aus, was den Abdeckungsbereich und ggf. die synthetische Zugänglichkeit einschränkt. In jüngster Zeit hat sich das Screening von Substanzbibliotheken mit kleinen, reaktiven Molekülen als sehr vielversprechend für die Entdeckung kovalenter Kinaseinhibitoren erwiesen. So haben St. Denis et al. eine elektrophile Fragmentbibliothek für ein röntgenkristallographisches Screening beschrieben, die die Entwicklung eines kovalenten ERK1/2-Inhibitors ermöglichte.15 Kuki et al. nutzten ein fragmentbasiertes Screening, um den ersten publizierten kovalenten EGFR-Inhibitor zu entwickeln, der auf das vergrabene C775 abzielt.16 Heitmann et al. identifizierten neue Hemmstoffe auf Chinazolinon-Basis für IP6K17 und die Gray-Gruppe entwickelte eine Strategie zur Nutzung einer einzelnen promiskuitiven Verbindung, mit der die Zugänglichkeit bestimmter Cysteine in Kinasen sondiert werden kann.10
Hier verwenden wir eine kleine Bibliothek von Acrylamid-tragenden, fragmentartigen Verbindungen, die eine Vielzahl von kanonischen „Hinge-Bindungselementen“ mit verschiedenen Linker-Geometrien kombinieren, um die Kinomabdeckung zu maximieren. Wir untersuchten 47 Kinasen mit 60 Cysteinen in der Nähe der ATP-Bindungstasche mit Hilfe von Massenspektrometrie und Thermal Shift Assays, gefolgt von einer weiteren Validierung der interessantesten Hits durch ergänzende Tests, Kristallstrukturen und kinetische Analysen der Bindungsbildung. Wir identifizierten 18 Kinasen mit Cysteinen an 19 verschiedenen Positionen, die zum ersten Mal kovalent adressiert wurden, wodurch wir die Zahl an nachweislich adressierbaren Cysteinen deutlich erhöhen konnten. Darüber hinaus wurden in unseren Kristallstrukturen mehrere einzigartige Bindungsmodi identifiziert, was eine Basis für die breitere Erforschung des adressierbaren Cysteinoms und die künftige Entwicklung kovalenter Hemmstoffe bildet.
Ergebnisse und Diskussion
Design und Synthese der Kinase-Fokussierten Kovalenten Fragmentbibliothek
Trotz der anhaltenden Entwicklung neuer kovalenter Kinaseinhibitoren wurden viele der Cysteinpositionen der Kinasen noch nicht kovalent adressiert.9 Unser Ziel war es, eine Kinase-fokussierte Bibliothek fragmentartiger reaktiver Moleküle zu entwerfen, welche die Identifizierung von Hits für Kinasen, die bisher nicht kovalent adressiert wurden, ermöglicht. Dazu haben wir verschiedene Hinge-Bindungsmotive, die die Affinität für Untergruppen des Kinoms definieren (Abb. 1), mit Linkern verschiedener Geometrie kombiniert, die wiederum das reaktive Acrylamid zu verschiedenen Cysteinpositionen ausrichten. Nach diesem Konzept wurden 47 Verbindungen generiert. Obwohl die Verbindungen an der oberen Grenze des chemischen Raums, der für „Fragmente“ definiert wurde angesiedelt sind, entsprechen die meisten von ihnen der sog. „rule of three“ (RO3).18 Dementsprechend werden sie im gesamten Manuskript der Einfachheit halber als „kovalente Fragmente“ bezeichnet. Zuvor lieferte ein ähnlicher Ansatz, jedoch unter Verwendung einer kleineren Bibliothek aus 10 Verbindungen mit einem reversibel-kovalenten Cyanoacrylamid-Warhead, den Ausgangspunkt für den ersten publizierten Inhibitor der C-terminalen Kinasedomäne der MSK1 (CTD; hier bezeichnet als MSK1~2).19 Das Hinge-Bindungsmotiv und sein Wasserstoffbrückenbindungsmuster sind nicht nur ausschlaggebend für die Ausrichtung und Rigidität der angehängten Teile innerhalb der Bindungstasche, sondern können auch als Selektivitätsfilter fungieren, da Kinasen unterschiedliche Präferenzen hinsichtlich ihrer Hinge-Interaktionen zeigen.20 Wir wählten sechs verschiedene N-Heterocyclen als ATP-Mimetika mit unterschiedlichen Linker-Anknüpfungspunkten und Substitutionsmustern, was insgesamt 14 verschiedene Ausgangsvektoren ergab. Zudem haben wir uns für die Anbringung von neun verschiedenen Linkern auf Arylbasis mit verschiedenen Topologien entschieden, um den elektrophilen Warhead anzubringen. Vielversprechende Kombinationen wurden mit dem Ziel ausgewählt, eine hohe topologische Vielfalt zu erreichen, um ein effektives Scannen der Oberfläche der ATP-Bindungstasche zu ermöglichen. Um Spezifität zu erzielen, setzten wir schwach reaktive Acrylamid-Elektrophile ein, welche eine initiale reversible Bindung erfordern, um die Warheads auzurichten und so die effiziente Bildung der kovalenten Bindung zu ermöglichen. Aufgrund ihrer relativ geringen intrinsischen Reaktivität und der damit verbundenen geringen Promiskuität sind Acrylamide gut etablierte reaktive Gruppen für die Adressierung von Cysteinen, wie die Mehrzahl der von der FDA zugelassenen kovalenten Kinaseinhibitoren zeigt.21
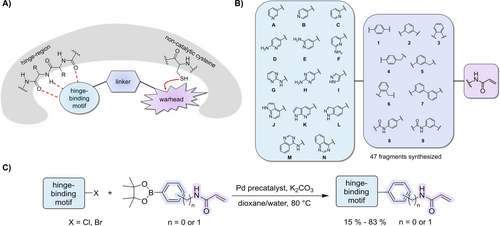
Design und Synthese der kovalenten Fragmentbibliothek. A. Designhypothese für die kovalenten Fragmente. B. Die Hinge-Bindungsmotive A–N wurden über Aryl-Linker 1–9 mit einem elektrophilen Acrylamid verbunden, um eine aus 47 Molekülen bestehende Bibliothek zu erzeugen. C. Synthese der meisten Fragmente durch Suzuki-Kreuzkupplung des halogenierten Hinge-Bindungsmotivs und Phenyl- oder Benzylacrylamid-Boronsäurepinakolester.
Die Synthese der meisten Fragmente erfolgte durch Suzuki-Kupplungen des Hinge-bindenden Heteroarylhalogenids mit einem zuvor synthetisierten Phenyl- oder Benzylacrylamid-boronsäurepinakolester. Diese Route ermöglichte eine schnelle und einfache Synthese von 37 Fragmenten (Abb. 1C). Für die beiden Verbindungen mit Pyrazol als Hinge-Bindemotiv (I1, I2) musste die Synthese unter Verwendung des Pyrazolboronsäurepinakolesters und des Linkerhalogenids invertiert werden, um angemessene Ausbeuten zu erzielen. Die Ausbeuten der Suzuki-Kupplungsreaktion von Pyrazol- und Indazolderivaten wurden durch Boc-Schützung des Azol-NH’s verbessert. Die Strukturen A8, A9, B8 und B9 wurden durch Amidkupplung der entsprechenden Pyridincarbonylchloride mit zuvor hergestellten N-(Aminophenyl)acrylamiden synthetisiert. N-verknüpfte Verbindungen mit dem Chinazolin-Hinge-Bindemotiv M wurden über nukleophile aromatische Substitutionen von N-(Aminophenyl)acrylamiden oder N-(Aminobenzyl)acrylamiden und 4-Chlorchinazolin hergestellt.
Biologische Analyse der Fragmentbibliothek
Um das Spektrum der kovalent adressierbaren Kinasen zu erweitern und um Startpunkte für künftige medizinalchemische Projekte zu erhalten, wurden 47 Proteinkinasen mit insgesamt 60 Cysteinresten in der Nähe des aktiven Zentrums ausgewählt. Diese Kinasen, von denen viele zum wenig untersuchten Teil des Kinoms gehören, weisen Cysteine in der P-Schleife („P-Loop“), der Dach-Region („Roof“), der Aktivierungs- oder A-Schleife, („Activation Loop“), der hinteren Tasche („Back Pocket“), der „Hinge-Region“, der vorderen Tasche („Front Pocket“) und der DFG-Region auf (Abb. 2A, B). Ein erstes Screening der Fragmentbibliothek erfolgte durch Flüssigchromatographie-gekoppelte Massenspektrometrie (LC/MS) der intakten Proteine, bei der die kovalente Modifikation quantifiziert wurde, sowie durch ergänzende DSF-Assays („Differential Scanning Fluorimetry“), die eine Abschätzung der Bindungsaffinitäten der Fragmente ermöglichten. Bei Letzteren deuten Tm-Verschiebungen unter 2 K auf eine schwache oder keine spezifische Interaktion hin, Verschiebungen zwischen 2 K und 5 K auf eine moderate Bindungswechselwirkung, während Verschiebungen>5 K typischerweise auf eine starke Bindung hinweisen. Die interessantesten Treffer wurden durch Mutagenese-Studien, Co-Kristallisation und Kinase-Enzym-Assays, MS-basierte kinact/KI-Bestimmung, tryptischen Verdau und zelluläre NanoBRET-Assays weiter analysiert. Unser Ansatz führte zu Treffern für mehrere Kinasen und Cysteinpositionen, für die bisher keine kovalenten Liganden bekannt waren. Die Ergebnisse der MS- und DSF-Tests sind in Abb. 2C zusammengefasst. Aufgrund der großen Anzahl der erzielten Hits werden im Hauptmanuskript nur einige interessante Beispiele hervorgehoben. Die vollständigen Screening-Daten sowie die Fragmentstrukturen und Cysteinpositionen sind im Anhang verfügbar.
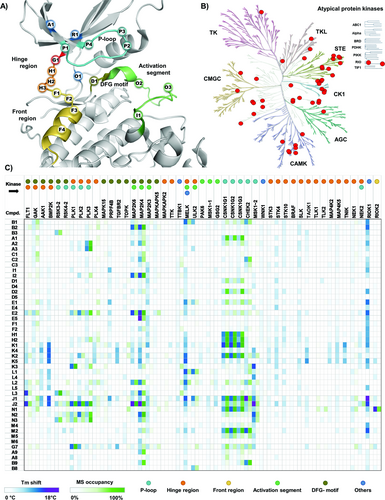
A. Hier ist das Cysteinom der Proteinkinasen mit potenziell adressierbaren Cysteinen um das aktive Zentrum der Kinasedomäne hervorgehoben. Die Abbildung wurde auf der Grundlage der Analyse von Chaikuad et al9 erstellt. B. Repräsentativer Satz von Kinase-Targets mit Cysteinen an nicht konservierten Positionen, gegen welche die Bibliothek kovalenter Fragmente getestet wurde. C. Heatmap, die die Wechselwirkungen zwischen den kovalenten Fragmenten und den untersuchten Proteinkinasen zeigt. DSF-Assays sind mit blauen Farbgradienten hervorgehoben, und LC/MS-Experimente der intakten Proteine nach 2 Stunden Inkubation mit zweifachem Substanzüberschuss sind mit einem grünen Gradienten dargestellt (siehe Bildunterschrift).
Im Allgemeinen sind Kinasen mit Cysteinen in der P-Schleife, der vorderen Region, der DFG-Region und der A-Schleife mit unserer kovalenten Fragmentbibliothek besser zugänglich, während Kinasen mit Cysteinen in der Hinge-Region und vergrabenen Cysteinen in der Dach-Region und der hinteren Tasche schwerer kovalent zu adressieren sind. Es ist anzumerken, dass bei Kinasen mit einem Cystein in der Hinge-Region das Vorhandensein des Warheads die Hinge-Bindung aufgrund von sterischer Abstoßung oder Inkompatibilität des kovalenten Bindungsmodus mit der Hinge-Interaktion negativ beeinflussen kann. Dies ist wahrscheinlich der Grund für die allgemein niedrigen Trefferquoten unserer Bibliothek in Bezug auf Hinge-Cysteine.
Die am häufigsten adressierten Kinasen waren die Caseinkinase 1 (CK1) gamma-Isoformen CSNK1G1/2/3, die eine hohe Sequenzidentität innerhalb ihrer Kinasedomäne aufweisen und einen Cysteinrest in der P-Schleife (Position P1; siehe Abb. 2A) teilen. Den eng verwandten CK1-Familienmitgliedern CSNK1A1, CSNK1D und CSNK1E fehlt dieses Cystein, so dass ein kovalenter Ansatz das Repertoire an selektiven Hemmstoffen für die CSNK1G-Isoformen erweitern könnte.22 Obwohl unseres Wissens nach noch keine kovalenten Inhibitoren von CK1-Kinasen bekannt sind,11 wurden bei unserem Screening mehrere kovalente Treffer mit vielversprechender Affinität und/oder Selektivität identifiziert. Es wurde beschrieben, dass CSNK1G-Isoformen eine wichtige Rolle bei der Regulierung verschiedener relevanter Signalwege spielen, jedoch wurden bisher nur wenige Inhibitoren veröffentlicht.23 Insgesamt wurden sieben Fragmente identifiziert, die kovalent mit einem ΔTm-Wert von über 4 K binden. Unter diesen zeigte das Azaindolderivat K1 die höchste thermische Stabilisierung der CSNK1G-Isoformen mit ΔTm-Werten von 8,7 K, 9,4 K und 7,2 K für CSNK1G1, CSNK1G2 bzw. CSNK1G3. Die Massenspektrometrie zeigte eine 100 %ige kovalente Modifikation aller CSNK1G-Isoformen nach 2 Stunden Inkubation. Exemplarisch für die CK1-gamma-Familie testeten wir die in vitro-Potenz von K1 für CSNK1G2 in einem enzymatischen Assay (ReactionBiology HotSpot™ Assay, siehe Anhang Tabelle S1). Die Verbindung zeigte einen IC50-Wert von 0,57 μM, was dieses Fragment als vielversprechenden Ausgangspunkt für die Entwicklung kovalenter CSNK1G-Inhibitoren hervorhebt. Darüber hinaus zeigten K1, H1 und H2 eine vielversprechende Selektivität gegenüber anderen Kinasen in unserem Panel. Wie erwartet diskriminierten die Hits jedoch nicht zwischen den einzelnen CSNK1G-Isoformen.
Die dual-spezifischen mitogen-aktivierten Proteinkinase-Kinasen (MAP2K1–MAP2K7) gehören zur STE7-Familie und haben einen Cysteinrest an der D1-Position, der dem konservierten DFG-Motiv vorausgeht. Diese Kinasen regulieren wichtige zelluläre Funktionen wie die Proliferation, Differenzierung, Motilität und das Überleben von Zellen und werden mit entzündlichen Erkrankungen sowie Krebs in Verbindung gebracht.24 Obwohl sich viele Studien auf die Entwicklung von MAP2K (MEK)-Inhibitoren konzentrierten,25 wurden bisher nur wenige selektive kovalente Inhibitoren für diese Familie beschrieben. Exemplarisch für die STE7-Familie haben wir weniger erforschte Targets wie MAP2K3 (MKK3), MAP2K4 (MKK4) und MAP2K6 (MKK6) in unser Screening-Panel aufgenommen. Die strukturell nah verwandten Isoformen MAP2K4 und MAP2K6 wurden von einer Reihe von Verbindungen wie B2, E2, L2 und J2 adressiert, jedoch mit geringer Spezifität. Das Azaindolfragment J2, das in unserem Screening insgesamt die niedrigste Selektivität aufwies, zeigte die höchste thermische Stabilisierung und kovalente Modifikation von MAP2K6 (ΔTm=11,6 K, MS 100 % kovalente Bindungsbildung nach 2 h). Ähnliche Werte wurden für MAP2K4 (ΔTm=11,9 K, MS 100 %ige Bindungsbildung) und für mehrere Kinasen, die ein Cystein an der P4-Position der P-Schleife tragen, wie RSK4~2 (d. h. die C-terminale der beiden RSK-Kinasedomänen), PLK1/2/3, MSK1~2 und NEK2, ermittelt. Eine nicht-kovalente Bindung an BMP2K, MELK und RIOK1 wurde ebenfalls beobachtet.
Es ist wichtig zu beachten, dass das erreichbare ΔTm-Werte zwischen den verschiedenen Kinasen variiert und nicht unbedingt mit der Hemmaktivität korreliert. Daher haben wir J2 in enzymatischen und zellulären Assays weiter auf die Bindung gegen PLK1,2,3 und FLT1 untersucht (Anhang Tabelle S1). Im enzymatischen Assay zeigte Azaindol J2 eine niedrig mikromolare Aktivität gegenüber FLT1, PLK1 und PLK2 (3,2, 3,4 bzw. 1,1 μM), wobei auch in einem zellulären NanoBRET-Assay eine ähnliche Wirksamkeit gegenüber FLT1 (EC50=2,95 μM) bestätigt werden konnte.
Darüber hinaus synthetisierten wir ein nicht reaktives Propionamid-Äquivalent von J2, genannt J2i, das im NanoBRET-Assay etwa viermal weniger aktiv war (EC50=11,9 μM). Dies deutet darauf hin, dass die kovalente Bindungsbildung auch in lebenden Zellen stattfindet. Im NanoBRET-Assay wurde zudem eine starke Bindung von J2 an MAP2K6 (EC50=0,27 μM) beobachtet. Die Fragmente B2 und I2 zielten den DSF-Daten zufolge selektiv auf MAP2K4 und MAP2K6 ab, und die kovalente Bindung wurde durch LC/MS bestätigt. Interessanterweise hatten alle Fragmente, die mehr als 50 % kovalente Bindung an MAP2K6 zeigten, denselben Linker (meta-Phenyl-Linker 2) zwischen Warhead und Hinge-Bindungsmotiv. Dies deutet darauf hin, dass dieser Linker ein bevorzugtes Motiv für die kovalente Bindung an das D1-Cystein von MAP2K6 ist. Bemerkenswerterweise zeigte das Aminopyridin E5, ein Analogon von E2, Affinität für MAP2K4 (ΔTm=13 K, MS 70 % Bindungsbildung) und traf keine andere Kinase aus der Gruppe, auch nicht die eng verwandte MAP2K6. Somit könnte diese Verbindung ein potenzieller Ausgangspunkt für die Entwicklung selektiver kovalenter Hemmstoffe von MAP2K4 sein, einem interessanten Zielprotein für die Leberregeneration.26 Diese herausragende Selektivität scheint auf die Methylengruppe zwischen Warhead und Phenyl-Linker zurückzuführen zu sein, welche die Flexibilität erhöht und gleichzeitig die Reaktivität des Warheads deutlich verringert. Diese Verringerung ergibt sich aus der Verlagerung des Amidstickstoffs in die benzylische Position, wodurch der elektronenziehende mesomere Effekt des aromatischen Rings auf den Amidstickstoff entfällt. Hierdurch wird die desaktivierende Wirkung des Stickstoffes auf das Michael-System verstärkt. Andere Verbindungen mit verwandten Methylen-Linkern zeigten im Screening keine Bindung an eine Kinase, unabhängig von dem angehängten Hinge-Bindungsmotiv, was ebenfalls mit den oben genannten Merkmalen zusammenhängen könnte. Eine isoformselektive Adressierung von MAP2K4 wurde auch für Chinazolin N2 (ΔTm=6,8 K, MS 100 % Modifikation) beobachtet. N2 zeigte jedoch auch Affinität für andere Kinasen im Screening wie die PLK-Isoformen und MSK1~2.
Die bereits erwähnte Rezeptor-Tyrosinkinase FLT1, die ein Cystein in der DFG-Region (D1) und der Hinge-Region (H2) aufweist, wurde nicht nur von Azaindol J2, sondern auch von Indazol L3 adressiert. Während J2 (ΔTm=9,8 K, MS 70 % Modifikation) eine stärkere Bindung zeigte, wies L3 (ΔTm=9,5 K, MS 35 % Modifikation) eine erhöhte Selektivität auf und hatte RSK3~2 (ΔTm=3,5 K, MS 100 % Modifikation) und MAP2K4 (ΔTm=6,8 K, MS 75 % Modifikation) als prominente zusätzliche Targets. Die Aktivität auf RSK3~2 war jedoch in enzymatischen und zellulären NanoBRET-Tests gering (IC50>25 μM, EC50>50 μM). Pyridin B3 wurde als Hit für RSK3~2 mit einer relativ geringen thermischen Stabilisierung (ΔTm=2,9 K, MS 70 % Modifikation), aber hoher Selektivität innerhalb des Panels identifiziert, während Chinazolin N3 (ΔTm=3,6 K, MS 75 % Modifikation) weniger selektiv war. Diese Verbindungen, die einen ortho-substituierten Linker gemeinsam haben, zeigen interessante frühe SARs für die Entwicklung von kovalenten RSK3~2-Inhibitoren auf.
Die Kinase MELK gehört zur CAMK-Familie und hat vier Cysteinreste in der Umgebung des aktiven Zentrums, und zwar an Position O1 unterhalb des Gatekeeper-Rests, O2 und O3 in der Aktivierungsschleife und H2 in der Hinge-Region. Die Aufklärung der Rolle von MELK bei Krebs wird dadurch erschwert, dass es keine gut charakterisierten, selektiven Inhibitoren gibt und eine erfolgreiche kovalente Adressierung bisher nicht beschrieben wurde. Wir haben vier Fragmente mit einem para-(J1, B1, L1) oder meta-substituierten Phenyl-Linker (L2) identifiziert, die MELK mit ΔTm zwischen 6,2 K und 7,7 K stabilisierten, und alle Verbindungen - außer Azaindol J1 – zeigten≥50 % kovalente Modifikation. Interessanterweise zeigte diese Kinase auch viele Treffer im DSF-Screening, die keine kovalente Bindung ausbildeten. Der selektivste kovalente MELK-Ligand, Indazol L1 (ΔTm=7,7 K, MS 50 % Modifikation, IC50=2,85 μM), richtete sich auch gegen ULK2 (ΔTm=6,0 K und MS 60 % Modifikation), welche ein H2 und O3 Cystein aufweist, und ist somit auch ein interessanter Ausgangspunkt für die Entwicklung neuartiger, kovalenter ULK2-Inhibitoren. Solche Verbindungen wären für die Untersuchung der Autophagie-Stoffwechselwege sehr wertvoll. Die größte thermische Stabilisierung unter allen Verbindungen und Kinasen wurde für CHEK2, welche ein Cystein in der P-Schleife (P3) enthält, und Azaindol J1 (ΔTm=19,5 K, MS 80 % Modifikation) festgestellt. Die Hemmung wurde in einem enzymatischen Assay bestätigt (IC50=0,66 μM). Signifikante Tm-Verschiebungen wurden auch für RIOK1, MELK und BMP2K beobachtet, die jedoch nicht kovalent modifiziert wurden, sowie für alle CSNK1G-Isoformen, bei denen eine 100 %ige Bindungsbildung durch MS nachgewiesen wurde. Eine schwächere Stabilisierung von CHEK2 wurde für Aminochinazolin M2 (ΔTm=7,9 K, MS 95 % Bindungsbildung) und Pyridin B9 (ΔTm=5,4 K, MS 95 % Bindungsbildung) festgestellt. Das letztgenannte Fragment verwendet ein zusätzliches Amidmotiv zur Verknüpfung des Phenyl-Linkers. Solche verlängerten Verbindungen (siehe Linker 8 und 9) zeigten im Allgemeinen eine hohe Selektivität innerhalb unseres Kinasepanels. Interessante Treffer wurden auch für die RIO-Kinasen RIOK1 und RIOK2 identifiziert, zwei kaum untersuchte Kinasen mit ATPase-Funktion, die eine wichtige Rolle bei der Regulierung der Transkription zu spielen scheinen.27 Während für RIOK1, das ein A1-Cystein aufweist, zahlreiche nicht-kovalente Hits gefunden wurden, identifizierten wir Chinazolin N1 als den einzigen kovalenten Liganden für RIOK2, welche ein Cystein in der vorderen Tasche aufweist. Die Bindung dieser Verbindung wurde außerdem durch NanoBRET (EC50=4,10 μM) und kinetische Experimente bestätigt (siehe unten).
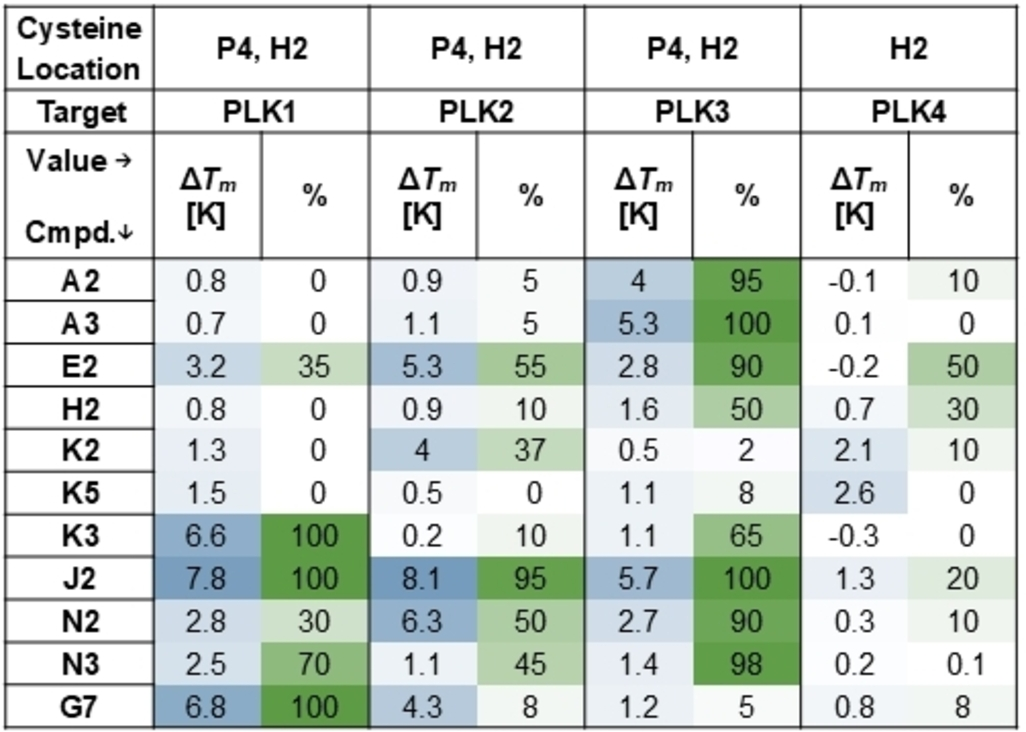
Ein interessantes Bindungsmuster wurde für die Mitglieder der Polo-like-Kinase-Familie PLK1–4 ermittelt (Tabelle 1). PLKs haben eine Sequenzidentität von 38 % bis 70 % innerhalb ihrer Kinasedomänen, und eine isoformselektive Hemmung wurde bisher noch nicht erreicht.28 Da PLK-Mitglieder bei zahlreichen Krankheiten, insbesondere Krebs, eine wichtige Rolle spielen, wurden bereits mehrere PLK-Inhibitoren in klinische Studien gebracht, und es besteht großes Interesse an der Entwicklung isoformselektiver Inhibitoren.29 Diese Kinasen haben alle ein schwer zugängliches Cystein in der Hinge-Region (H2). PLK1, PLK2 und PLK3 besitzen zusätzlich ein Cystein in der P-Schleifen (P4), welches bisher noch nicht kovalent adressiert wurde. Wir konnten mehrere vielversprechende Hits für PLK1/2/3 identifizieren, während für PLK4, das Familienmitglied mit der geringsten Sequenzidentität und ohne das P4-Cystein, nur schwache Tm-Verschiebungen und kovalente Modifikationen gefunden wurden. Interessanterweise wurden, anders als in der CSNK1G-Familie, für (Amino)pyri(mi)din- und Azaindol-Verbindungen nennenswerte Isoform-Selektivitätsmuster sowohl hinsichtlich der thermischen Stabilisierung als auch der kovalenten Bindung gefunden, wobei einige Treffer (z. B. Pyridin A3 für PLK3 und Aminopyrimidin G7 für PLK1) eine vollständige kovalente Modifikation und eine vielversprechende Gesamtselektivität zeigten. Obwohl diese Trends in komplementären enzymatischen und zellulären NanoBRET-Tests nicht eindeutig bestätigt werden konnten, deuten die Daten darauf hin, dass eine isoformselektive kovalente Hemmung von PLKs trotz der äquivalenten Cystein-Positionierung möglich sein könnte. Diese überraschende Selektivität könnte auf kleine Sequenzunterschiede in der Hinge-Region der verschiedenen PLK-Isoformen zurückzuführen sein, die zu einer leicht unterschiedlichen Positionierung der Fragmente führen und dadurch die Affinität und die Reaktionstrajektorie verändern können.
Kinetische Charakterisierung von Schlüssel-Verbindungen
Wir untersuchten außerdem die Korrelation zwischen den DSF ΔTm-Werten und der durch LC/MS gemessenen kovalenten Modifikation (Abb. 3A). Interessanterweise wiesen diese Parameter keine ausgeprägte Korrelation auf, und selbst manche Fragmente mit relativ geringer thermischer Stabilisierung zeigten eine signifikante kovalente Bindung. So erreichten beispielsweise Hits für PLK3 mit ΔTm-Werten zwischen 1,4 K und 5,7 K eine kovalente Modifikation von über 90 %. Wichtig ist, dass die irreversible kovalente Bindung ein zeitabhängiger Prozess ist, der durch die nicht-kovalente Affinität (definiert durch die Dissoziationskonstante Ki oder die verwandte Konstante KI) oder die chemische Reaktion, welche die kovalenten Bindung ausbildet (definiert durch die Geschwindigkeitskonstante erster Ordnung kinact) angetrieben werden kann. Somit werden irreversible kovalente Inhibitoren am besten durch den zeitunabhängigen kinact/KI-Wert charakterisiert, eine Geschwindigkeitskonstante zweiter Ordnung, die die Gesamteffizienz des kovalenten Bindungsprozesses definiert.30 Die oben erwähnte fehlende Korrelation zwischen ΔTm und kovalenter Bindung steht im Einklang mit der Tatsache, dass schwache nicht-kovalente Liganden zu potenten kovalenten Inhibitoren gemacht werden können, wenn eine ausreichende Reaktionsrate am Zielprotein (d. h. kinact) erreicht werden kann, wobei dies idealerweise durch eine geeignete Ausrichtung in der Bindetasche ohne Erhöhung der intrinsischen Reaktivität erfolgen sollte.6 Um weitere Einblicke in die Bindungseigenschaften interessanter Hits zu erhalten, haben wir die Kinetik der kovalenten Bindung durch zeit- und konzentrationsabhängige MS-Experimente an intakten Proteinen ermittelt (siehe die Beispiele in Abb. 3C–F und SI).29 Die vollständigen kinetischen Daten sowie die IC50-Werte aus enzymatischen Assays und die EC50-Werte, die durch NanoBRET in HEK293T-Zellen bestimmt wurden, sind in SI-Tabelle S1 aufgeführt.
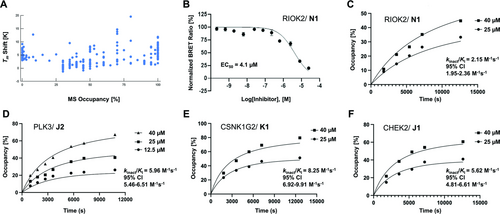
A. Korrelation der Ergebnisse aus DSF-Assays mit Protein LC/MS-Daten. Dargestellt sind ausgewählte Hits mit einer kovalenten Modifikation (LC/MS)>30 % und/oder DSF ΔTm>3 K. Die kovalente Modifikation ist in 5 %-Schritten gerundet. B. Beispiel eines zellulären NanoBRET-Experiments in HEK293T-Zellen, hier die Bindung von N1 an RIOK2. C. Kinetik der kovalenten Bindung (kinact/KI) von Verbindung N1 und RIOK2. D. Bindung der Verbindung J2 an PLK3. E. Bindung der Verbindung K1 an CSNK1G2. F. Bindung der Verbindung J1 an CHEK2. Die kinact/KI-Werte wurden durch die zeit- und konzentrationsabhängige Messung der kovalenten Bindungsbildung mittels ESI-TOF-MS am intakten Protein bestimmt.
Die kinetische Analyse von Pyridin A3 auf PLK3 ergab einen kinact/KI-Wert von 3,79 M−1 s−1, während die Inaktivierungseffizienzen von N1 auf RIOK2 und G7 auf PLK1 mit kinact/KI-Werten von 2,15 M−1 s−1 bzw. 2,37 M−1 s−1 etwas geringer waren. Die Inaktivierungseffizienzen von J2 an PLK3, J1 an CHEK2 und K1 an CSNK1G2 waren nur geringfügig höher (kinact/KI=5,96 M−1 s−1, 5,62 M−1 s−1 bzw. 8,25 M−1 s−1). Die Inaktivierungskonstanten dieser nicht optimierten Fragmente liegen zwar um mehrere Größenordnungen unter denen zugelassener kovalenter Kinaseinhibitoren (typischerweise im Bereich von 105–107 M−1 s−1),31 dennoch qualifizieren sie die identifizierten Treffer als vielversprechende Ausgangspunkte für weitere Optimierungen.
Reaktivität der Fragmente gegenüber Thiolen
Ein kritischer Parameter bei der Entwicklung kovalenter Inhibitoren ist die intrinsische Reaktivität des kovalenten Warheads, die auf ein notwendiges Minimum beschränkt werden sollte, um promiskuitive Reaktionen mit Thiolen (z. B. Cystein in Off-Target-Proteinen) oder anderen Nukleophilen zu vermeiden. Wir testeten die Stabilität ausgewählter Verbindungen bei physiologischem pH-Wert (pH 7,4, 40 °C, HEPES-Puffer/ACN 1 : 1) in Gegenwart von Glutathion (GSH; 5 mM) als reichlich vorhandenem und repräsentativem physiologischem Nukleophil (Tabelle 2).32 Die Daten zeigten, dass die Halbwertszeit der ausgewählten Verbindungen deutlich höher war als die des von der FDA zugelassenen Arzneistoffes Afatinib, was eine verhältnismäßig geringe Reaktivität für einen fragmentbasierten Ansatz bedeutet. Zudem ist hervorzuheben, dass wir nur eine geringe Korrelation zwischen der Reaktivität des Warheads und der Trefferquote am Zielprotein feststellen konnten. Dies deutet darauf hin, dass unser Ansatz Hits liefert, bei denen die kovalent Bindung nicht auf hohe intrinsische Reaktivität des Warheads sondern eine korrekte Ausrichtung durch initiale reversible Bindung zurückzuführen ist.
Fragment |
GSH t1/2 [h] |
Anzahl an Hits* |
|---|---|---|
N1 |
21.4 |
4 |
B9 |
39.6 |
2 |
A3 |
39.8 |
1 |
E2 |
53.3 |
5 |
J1 |
54.6 |
5 |
L3 |
55.0 |
2 |
J3 |
56.8 |
12 |
K1 |
76.2 |
3 |
G7 |
117.5 |
2 |
Afatinib |
3.2 |
n.d. |
Nachweis der Bildung einer Kovalenten Bindung mit den Angenommenen Cysteinpositionen
Um zu überprüfen, ob die identifizierten kovalenten Fragmente an den vorab angenommenen Cysteinrest binden, bestätigten wir die Bindung entweder durch Proteinkristallographie, zielgerichtete Mutagenese oder MS-basierte Analyse nach tryptischem Verdau (zusammengefasst in Tabelle 3). Die Bildung kovalenter Bindungen mit Cysteinen wurde durch Kristallstrukturen von MAP2K6 (DFG-Region) im Komplex mit J2 und E2, RIOK2 (vordere Tasche) mit N1 sowie MELK und ULK1 (Aktivierungsschleife) im Komplex mit L1 bestätigt (Abb. 4).
Kinase |
Methode |
Adressiertes cysteine |
Kinase-variante |
Bestätigtes Fragment |
|---|---|---|---|---|
FLT1 |
Mutagenese |
D1 |
FLT1 (C1039S) |
J2, L3 |
RSK3~2 |
Mutagenese |
P4 |
RSK3 (C429A) |
B3, N3, L3 |
RSK4~2 |
Mutagenese |
P4 |
RSK4 (C440A) |
J2, L3 |
RSK4~2 |
Mutagenese |
D1 |
RSK4 (C564A) |
N2 |
PLK3 |
Mutagenese |
P4 |
PLK3 (C76S) |
A2, A3, J2, I2, E2, H2, K3, N2, N3 |
TTBK1 |
Röntgenkristallstruktur |
O1 |
TTBK1 (wt) |
K3 |
MELK |
Mutagenese+Röntgenkristallstruktur |
O2 |
MELK (C154+wt) |
B1, L1, L2 |
ULK1 |
Röntgenkristallstruktur |
O3 |
ULK1 (wt) |
L1 |
ULK2 |
Mutagenese |
O3 |
ULK2 (C175S) |
J1, L1, B8 |
CSNK1G2 |
Mutagenese |
P1 |
CSNK1G2 (C54A+wt) |
K1, D2, A1, J1, H2, N1, K2, M2, M5 |
CHEK2 |
Mutagenese |
P3 |
CHEK2 (C231A) |
M2 |
MSK1~2 |
Mutagenese |
P4 |
MSK1 (C440A) |
J2 |
MAP2K6 |
Röntgenkristallstruktur |
D1 |
MAP2K6 (wt) |
J2, E2 |
RIOK2 |
Röntgenkristallstruktur |
F3 |
RIOK2 (wt) |
N1 |
NEK1 |
Tryptischer Verdau |
H2 |
NEK1 (wt) |
N1 |
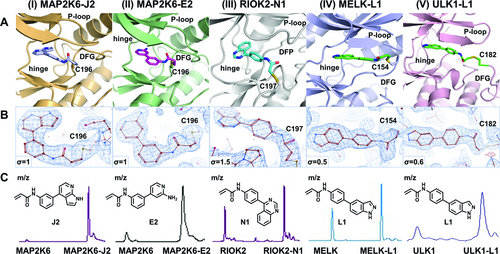
Co-Kristallstrukturen und Proteinmassenspektren, die die Bildung kovalenter Bindungen bestätigen. A. (I) MAP2K6, kristallisiert mit J2 bei einer Auflösung von 2,3 Å, gebunden an DFG–1 (D1) Cys196 (PDB: 8P7J). (II) MAP2K6, kristallisiert mit E2 bei einer Auflösung von 2 Å, gebunden an DFG–1 (D1) Cys196 (PDB: 8PM3). (III) RIOK2, kristallisiert mit N1 bei einer Auflösung von 3 Å, gebunden an die vordere Tasche (αD+2/F3) Cys197 (PDB: 9F81). (IV) MELK, kristallisiert mit L1 bei einer Auflösung von 2 Å, gebunden an die A-Schleife (DFG+2/O2) Cys154 (PDB: 9F31). (V) ULK1 kristallisiert mit L1 bei einer Auflösung von 2,1 Å, gebunden an die A-Schleife (O3) Cys182 (PDB: 9F32). Alle Strukturen wurden zusätzlich einem kovalenten Re-Docking unterzogen, das sehr ähnliche Posen ergab (siehe Hintergrundinformationen Abbildung S6). B. Gewichtete 2m-FO-DFc-Elektronendichtekarten, Kontur bei den σ-Werten, die in den Abbildungen in Feld B angegeben sind. C) Massenverschiebung durch kovalente Modifikation der Proteine mit der jeweiligen Verbindung, nachgewiesen durch LC/MS nach 2-stündiger Inkubation mit 2-fachem molaren Überschuss der Verbindungen.
In den Kristallstrukturen von MAP2K6 zeigten die beiden Fragmente E2 und J2 die erwartete Bindung an Cys196 vor dem DFG-Motiv, wobei das Aminopyridin und das Azaindol Wasserstoffbrückenbindungen zur Hinge-Region bildeten. Die kovalente Bindung mit dem DFG-1-Cystein verzerrte die Aktivierungsschleife und führte zu einer nach innen gerichteten Drehung des DFG-Phenylalanins (Phe198). Die P-Schleife nahm eine seltene gefaltete Konformation an. Es wurde bereits berichtet, dass dieser Bindungsmodus die Selektivität von Inhibitoren erhöht,33 z. B. für die mitogen-aktivierten Proteinkinasen MAPK14 und MAP4K4.34 Die Faltung der P-Schleife könnte aus der Wechselwirkung mit dem DFG-Phenylalanin resultieren, welches das Tyrosin (Tyr64) der P-Schleife in die Bindungstasche drückt und so eine π-Amid-Stacking-Wechselwirkung mit dem mitkristallisierten Fragment ermöglicht. Diese Wechselwirkung wurde sowohl in den Kristallstrukturen von E2 als auch von J2 beobachtet, war jedoch im E2/MAP2K6-Komplex deutlicher zu erkennen, da das Azaindol und der zentrale Phenyl-Linker eine orthogonale Ausrichtung zueinander einnahmen. Die kovalente Bindung des Fragments N1 an Cys197 in der lösungsmittelexponierten Frontregion der atypischen Kinase RIOK2 wurde ebenfalls durch eine Kristallstruktur bestätigt, was den ersten Nachweis einer Adressierung des F3-Cysteins in RIOK2 darstellt. Hier bildet der Chinazolin-Anteil eine Wasserstoffbrückenbindung, die auf die Hinge-Region gerichtet ist. Der Phenyl-Linker baut hydrophobe CH-π-Wechselwirkungen mit Pro195 in der vorderen Region auf, die zur Bindung beitragen. Das Fragment L1 wurde sowohl mit MELK als auch mit ULK1 kristallisiert. Wir wählten ULK1 anstelle der im Screening verwendeten ULK2, da ULK1 in der Regel zu höher aufgelösten Beugungsdaten führt.35 In den Kristallstrukturen von MELK und ULK1 waren die Aktivierungsschleifen verzerrt und auf die ATP-Bindungstasche ausgerichtet. L1 bildete eine kovalente Bindung mit dem O2-Cystein (Cys154) der Aktivierungsschleife in MELK bzw. dem O3-Cystein (Cys182) in ULK1 aus. Soweit wir wissen, handelt es sich hier um die ersten kovalenten MELK und ULK1 Inhibitoren und um ein seltenes Beispiel dafür, dass Cysteine in der Aktivierungsschleife kovalent adressiert werden. Wie erwartet, bildete die Indazoleinheit Wasserstoffbrücken mit dem Rückgrat in der Hinge-Region und hydrophobe Wechselwirkungen in der ATP-Bindungstasche aus. Eine zusätzliche Wasserstoffbrücke wurde im ULK1/L1-Komplex zwischen dem Stickstoff des Amids und dem Carbonyl des Peptidrückgrats von Histidin (His24) in der P-Schleife beobachtet. Darüber hinaus bildete das Carbonyl des Warheads eine Wasserstoffbrücke mit dem NH des kovalent modifizierten Cysteins (Cys182) aus, eine Wechselwirkung, die die Reaktion erleichtern könnte. TTBK1 wurde als zusätzliche Kinase im Komplex mit K3 kristallisiert, allerdings konnte die Struktur nur mit einer Auflösung von 3 Å gelöst werden, und die Elektronendichte für den Liganden war nicht von ausreichender Qualität, um K3 eindeutig darzustellen. Wir beobachteten jedoch zusätzliche Elektronendichte neben der Seitenkette von Cys91 (O1-Position), das sich in der hinteren Tasche befindet, und Cys96 an der Proteinoberfläche (ergänzende Abb. S3), was darauf hindeutet, dass dieses sterisch gehinderte Cystein der hinteren Tasche zugänglich ist. Nur etwa 5 Kinasen enthalten Cysteine an dieser Position, was eine hervorragende Möglichkeit für die Entwicklung selektiver Hemmstoffe bieten könnte.
Um die kovalenten Bindungsstellen der identifizierten Verbindungen weiter zu validieren, führten wir Mutagenesestudien durch, indem wir Cysteine entweder gegen Serin oder Alanin austauschten, gefolgt von LC/MS Analyse am intakten Protein (Abb. S2, Tabelle S2). Für die PLK-Familie bestätigten wir die kovalente Bindung von J2 an PLK3-Cys76, welches sich in der P-Schleife (P4-Position) befindet. Da J2 auch auf die anderen PLK-Isoformen (PLK1/2) abzielt, kann davon ausgegangen werden, dass die P4-Cysteine von PLK1/2 analog gebunden wurden. Durch Mutation des P1-Cysteins von CSNK1G2 und des D1-Cysteins von MAP2K6 bestätigten wir deren Adressierung durch K1 und N1 an CSNKGs und von E2 und J2 an MAP2K4/MAP2K6.
Darüber hinaus haben wir durch tryptischen Verdau und anschließende LC/MS-Analyse die kovalente Bindung von N1 an Cys83 in NEK1 bestätigt, das sich an der H2 Position in der Hinge-Region befindet, die im Allgemeinen schwer zu erreichen ist. Interessanterweise zeigte diese Verbindung eine beträchtliche kovalente Bindung (70 % Modifikation) und ist damit unseres Wissens der erste kovalente Ligand, bei dem eine kovalente Bindung and diese Cysteinposition nachgewiesen wurde. Da viele Kinasen ein H2-Cystein enthalten, ermutigen unsere Ergebnisse dazu, auch dieses Cystein zukünftig ins Visier zu nehmen. Analoge Analyse von PLK4-L2 ergab, dass Cys172 in der Nähe der A-Schleife adressiert wurde (Tabelle S3).
Zusammenfassend lässt sich sagen, dass wir mit unserem Ansatz eine Reihe von fragmentartigen Verbindungen identifizieren konnten, die kovalent auf verschiedene Cysteine in der Umgebung der ATP-Tasche abzielen. Das geringe Molekulargewicht und die begrenzte Komplexität der Verbindungen sowie die große Zahl an Ausgangsvektoren machen sie zu guten Startpunkten für die Entwicklung neuer Inhibitoren, insbesondere für Kinasen, die bisher nicht bzw. nicht gezielt kovalent adressiert wurden, wie z. B. PLK1/2/3, MELK, TTBK1, CSNK1G1/2/3, FLT1, NEK1, MAP2K6, RSK3, RSK4, MSK1, RIOK2, ULK1/2 und CHEK2.
Zusammenfassung und Schlussfolgerungen
Proteinkinasen enthalten nach bisherigem Stand der Literatur Cysteine an mindestens 18 Positionen in der Nähe der ATP-Bindungsstelle.9 Während über 200 Proteinkinasen ein Cystein an einer oder mehreren dieser Stellen aufweisen, wurden viele von ihnen noch nicht kovalent adressiert, und die derzeitigen kovalenten Kinaseinhibitoren sind weitestgehend auf die F2-Cysteinposition in der vorderen Tasche ausgerichtet. Hier stellen wir einen modularen Ansatz für Kinase-fokussierte fragmentartige Verbindungen vor, die diese verschiedenen Cysteinpositionen adressieren. Zur Validierung des Konzepts haben wir eine Bibliothek von 47 Verbindungen synthetisiert und an 47 Kinasen mit 60 potenziell erreichbaren Cysteinen mittels Massenspektrometrie und DSF-Assays getestet. So konnten wir Einblicke in die Adressierbarkeit einzelner Cysteinpositionen erhalten. Letztere kann aufgrund unterschiedlicher Proteinumgebungen, die die Zugänglichkeit und den pKs-Wert und somit die Nukleophilie der Cysteine bestimmen, stark variieren.36 Um eine breite Abdeckung an Cysteinpositionen zu erreichen, wählten wir Kinasen mit Cysteinen in der P-Schleife, der Dach-Region, der Hinge-Region, der hinteren Tasche, der vorderen Tasche, der DFG-Region oder der Aktivierungsschleife aus und konzentrierten uns dabei hauptsächlich auf den weniger untersuchten Teil des menschlichen Kinoms. Wir entdeckten kovalente Inhibitoren für Kinasen und Cysteinpositionen, die bisher noch nicht bzw. nicht gezielt adressiert wurden, darunter CSNK1G1/2/3, CHEK2, PLK1/2/3, RSK3/4 mit P-Loop-Cysteinen, MELK und ULK1/2 mit Cysteinen in der A-Schleife, FLT1 und RSK4 mit Cysteinen in der DFG-Region und RIOK2 mit einem Cystein in der vorderen Tasche. Die einzelnen Zielcysteine konnten durch Mutagenesestudien, tryptischen Verdau und/oder Kristallstrukturen validiert werden. Wir konnten außerdem zeigen, dass ein Cystein an der H2-Position (GK+3), von dem man annahm, dass es aufgrund sterischer Hinderung und seiner Lage unterhalb der Hinge-Region für ein Elektrophil schwer zu erreichen ist, in NEK1 kovalent modifiziert werden kann. Obwohl wir nicht in der Lage waren, den genauen Bindungsmodus des identifizierten Hits (d. h. Chinazolin N1) aufzuklären, bieten diese Ergebnisse eine Grundlage für eine systematischere Erforschung der Adressierbarkeit dieses Cysteins, welches das am stärksten konservierte Cystein des Kinoms ist und in über 80 Kinasen vorkommt. Während Cysteine in der hinteren Tasche und der Dach-Region weniger zugänglich zu sein scheinen, bestätigen unsere Strukturdaten von TTBK1 die generelle Adressierbarkeit der Cysteinposition hinter dem Gatekeeper-Rest (O1). Eventuell könnten lipophilere und/oder räumlich eingeschränktere Warheads besser geeignet sein, um solche Cysteine zu adressieren. Dies wurde in Studien gezeigt, in denen Alkinyl-substituierte Heteroaryl-Warheads eingesetzt wurden, um ein Cystein in der hinteren Tasche der NF-κB-induzierenden Kinase NIK zu adressieren.37 Bemerkenswert ist, dass unsere Verbindungen trotz ihrer begrenzten Komplexität im Allgemeinen eine geringe Promiskuität aufwiesen und wir keine Korrelation zwischen der intrinsischen Reaktivität und der Trefferquote in unserem Screen beobachten konnten. Dies stimmt mit einem kovalenten Mechanismus überein, der die Bildung eines nicht-kovalente Komplexes vor Ausbildung der kovalenten Bindung erfordert. Die vielversprechendsten Hits wurden in enzymatischen und zellulären NanoBRET-Assays, MS-basierten kinetischen Messungen (kinact/KI) sowie mittels GSH-Reaktivitätsdaten weiter charakterisiert, wodurch wir einen umfangreichen Datensatz zur Verfügung stellen, der künftige medizinalchemische Entwicklungen für diese und weitere Zielproteine ermöglicht.
Zusammenfassend lässt sich sagen, dass unsere Studie einen einfachen und rationalen Ansatz zur Untersuchung des Cysteinoms der Proteinkinasen aufzeigt und vielversprechende Startpunkte für die künftige Entwicklung wirksamer und selektiver kovalenter Kinaseinhibitoren für den weniger untersuchten Teil des Kinoms und darüber hinaus liefert.
Hintergrundinformationen
Beschreibungen der Experimente, NMR-Spektren, Bioassay- und Bindungskinetikdaten, GSH-Stabilitäten sowie die vollständigen Screening- und Peptid-Mapping-Daten sind als Anhang beigefügt. Die Autoren haben zusätzliche Referenzen im Anhang angegeben, die mit fortlaufender Nummerierung in das Manuskript aufgenommen wurden.
Danksagung
SR, YP, B-TB, AC und SK sind dankbar für die Unterstützung durch das Structural Genomics Consortium (SGC), eine eingetragene Wohltätigkeitsorganisation (Nr. 1097737), die Mittel von der Bayer AG, Boehringer Ingelheim, Bristol Myers Squibb, Genentech, Genome Canada über das Ontario Genomics Institute, EU/EFPIA/OICR/McGill/KTH/Diamond Innovative Medicines Initiative 2 Joint Undertaking [EUbOPEN grant 875510], Janssen, Pfizer und Takeda erhält. GW und SK bedanken sich für die Unterstützung durch das Deutsche Krebsforschungszentrum (DKTK). GW dankt außerdem für die Unterstützung durch das CSC-Stipendium (Nr. 202008080291). Die Studie wurde zudem von der Deutschen Krebshilfe unterstützt (TACTIC; Projektnummer 70115201, an SK, MG, SAL), wofür wir uns bedanken. XJL, LH, MF, SAL und MG bedanken sich bei der Deutschen Forschungsgemeinschaft (DFG) im Rahmen der Exzellenzstrategie EXC 2180–390900677 [„Image Guided and Functionally Instructed Tumor Therapies (iFIT)“] für die Förderung. MG dankt außerdem der Deutschen Forschungsgemeinschaft (DFG), Projektnummer 511101075, und dem Fonds der Chemischen Industrie (FCI) für die finanzielle Unterstützung. LH und MG bedanken sich für die Förderung durch das Eliteprogramm für Postdocs der Baden-Württemberg Stiftung. NS und MG danken Frederik Wilhelm Hacker für technische Unterstützung und Benedikt Masberg für die Messung von HRMS-Daten.
B.-T.B. ist Mitgründer und Geschäftsführer des Auftragsforschungsinstituts CELLinib GmbH (Frankfurt am Main, Deutschland). Die anderen Autoren erklären, dass keine Interessenkonflikte bestehen. Open Access Veröffentlichung ermöglicht und organisiert durch Projekt DEAL.
Interessenkonflikt
Die Autoren erklären, dass keine Interessenkonflikte vorliegen.
Open Research
Data Availability Statement
Ein Teil der Daten (Röntgenkristallstrukturen) wurde in der Protein Data Bank hinterlegt. Diese werden bei Veröffentlichung der Studie offengelegt. Weitere Daten sind in den Hintergrundinformationen inklusiver der angehängten Excel-Dateien zusammengestellt. Die vollständigen unprozessierten Datensätze können auf Anfrage von den Autoren erhalten werden.