Eine DNA-Polymerase-Variante erkennt den epigenetischen Marker 5-Methylcytosin durch erhöhten Fehleinbau
Abstract
Eine Dysregulation der DNA-Methylierung wird mit der Entstehung humaner Krankheiten, insbesondere Krebs, in Verbindung gebracht. Die Untersuchung abnormaler Methylierungsmuster ist daher vielversprechend für die klinische Diagnostik. DNA-Polymerasen unterscheiden jedoch nicht effektiv zwischen der Umsetzung von 5-Methylcytosin (5 mC) und unmethyliertem Cytosin, wodurch die Methylierungsinformation während der Amplifikation oder Sequenzierung verloren geht. Aktuelle Nachweismethoden erfordern daher mehrstufige DNA-Konversionsbehandlungen oder eine sorgfältige Analyse der Sequenzierungsdaten, um einzelne 5 mC-Basen zu entschlüsseln. Um diese Herausforderung zu überwinden, schlagen wir einen neuartigen DNA-Polymerase-vermittelten Ansatz zur Detektion von 5 mC vor. Hier beschreiben wir die Entwicklung einer thermostabilen DNA-Polymerase-Variante aus Thermus aquaticus, die eine veränderte Genauigkeit für 5 mC aufweist. Durch einen Screening-Ansatz konnten wir eine DNA-Polymerase identifizieren, die während der DNA-Synthese einen erhöhten Fehleinbau gegenüber 5 mC zeigt. Diese DNA-Polymerase erzeugt Mutationssignaturen an methylierten CpG-Stellen, die einen direkten Nachweis von 5 mC durch das Auslesen einer erhöhten Fehlerrate nach der Sequenzierung ohne vorherige Behandlung der Proben-DNA erlaubt.
Die häufigste epigenetische Modifikation im menschlichen Genom ist 5-Methylcytosin (5 mC), das 4 % aller Cytosin (C)-Nukleinbasen ausmacht.1 Die postreplikative enzymatische Addition einer Methylgruppe an das 5-Kohlenstoffatom von C wird als DNA-Methylierung bezeichnet2 und tritt hauptsächlich in einem symmetrischen Muster an 5′-Cytosin-Phosphat-Guanin-3′-Dinukleotiden (CpG)3 auf. Die DNA-Methylierung spielt eine wesentliche Rolle bei der Regulation der Genexpression4 und beeinflusst entscheidend Prozesse wie die X-Inaktivierung,5 die genomische Prägung,6 sowie die zelluläre Entwicklung und Differenzierung.7 Dysfunktionen und Veränderungen in Methylierungsmustern können mit dem Auftreten verschiedener humaner Krankheiten, insbesondere maligner Erkrankungen, in Verbindung gebracht werden.8 Daher gelten abnorme Hyper- und Hypomethylierungen als vielversprechende Biomarker für die Diagnose und Prognose von Krebserkrankungen.9
DNA-Polymerasen sind für die Replikation und Erhaltung der in der DNA kodierten genetischen Information unerlässlich. Zudem werden sie häufig in biotechnologischen Anwendungen zur Nukleinsäureanalyse durch Amplifikation oder Sequenzierung eingesetzt.10 Allerdings unterscheiden DNA-Polymerasen nicht zwischen der Umsetzung von 5 mC und unmodifiziertem C,11 was zu einem Verlust der Methylierungsinformation während der DNA-Synthese führt. Die Basenpaarungseigenschaften von 5 mC bleiben unverändert, da die Methylgruppe von 5 mC die Watson-Crick-Wasserstoffbrückenbindungen nicht beeinträchtigt12 und zur großen Furche der DNA-Doppelhelix ausgerichtet ist. In dieser Furche können DNA-Polymerasen sogar wesentlich sperrigere Modifikationen akzeptieren, die um mehrere Größenordnungen größer sind als das natürliche Substrat.13
Folglich benötigen Short-Read-Sequenzierungsmethoden, die üblicherweise zur Erstellung von Methylierungsprofilen mit Einzelbasenauflösung verwendet werden, eine chemische und/oder enzymatische Vorbehandlung der DNA-Probe, um 5 mC-Stellen von unmodifizierten Cs zu unterscheiden. Die Bisulfit-Sequenzierung ist der Goldstandard für die Detektion von 5 mC und basiert auf einer Bisulfit-Behandlung der Matrizen-DNA, bei der C in Uracil (U) umgewandelt wird, während 5 mC unverändert bleibt.14 Auf die Konversion folgen Amplifikation und Sequenzierung, wodurch 5 mC als unverändertes C gelesen wird, während unmethyliertes C als Thymin (T) gelesen wird.15 Obwohl die Bisulfit-Sequenzierung eine seit langem etablierte Methode ist, hat sie erhebliche Nachteile.16 Die Konversion von unmodifiziertem C in U reduziert die Komplexität der DNA-Sequenzen und beeinträchtigt nachfolgende Arbeitsschritte und die Analyse der Sequenzierungsdaten.17 Außerdem führen die harschen Reaktionsbedingungen zur Fragmentierung und zum Abbau von bis zu 99 % der Proben-DNA.18 Enzymatische, Bisulfit-freie Konversionsverfahren arbeiten mit deutlich milderen Reaktionsbedingungen und die Proben-DNA bleibt nach der Behandlung weitgehend intakt.19 Dennoch sind konversionsbasierte Methoden in der Regel durch mehrere Arbeitsschritte, Adenin(A)T-reiche Sequenzprodukte sowie unvollständige oder fehlerhafte Konversion beeinträchtigt.20 Neben der Sequencing-by-Synthesis-Methode stellt die Long-Read-Sequenzierung von Oxford Nanopore, die eine direkte Detektion von 5 mC ermöglicht, eine vielversprechende Alternative dar.21 Der erfolgreiche Nachweis von 5 mC erfordert jedoch große Mengen an unamplifizierter Templat-DNA und ein umfangreiches Algorithmentraining.22 Darüber hinaus führt die Variabilität der Signalveränderungen zu unterschiedlichen und begrenzten Genauigkeiten der Methylierungs-Calling-Tools.23
Um einen direkten Nachweis von 5 mC mit Einzelbasenauflösung zu ermöglichen, ohne die Probe vor der Analyse konvertieren zu müssen, berichten wir hier über ein gentechnisch verändertes thermostabiles Enzym, welches auf der Basis des großen Fragments der DNA-Polymerase von Thermus aquaticus (KlenTaq DNA-Polymerase,24 im Folgenden als KTq bezeichnet) entwickelt wurde und eine veränderte Genauigkeit gegenüber 5 mC aufweist. Im Vergleich zu ihrem unmodifizierten Gegenstück erkennt die DNA-Polymerase die modifizierte Nukleinbase durch einen erhöhten Fehleinbau während der Polymerase-Kettenreaktion (PCR). Die daraus resultierenden Mutationssignaturen an methylierten CpG-Stellen werden anschließend durch Auslesen erhöhter Fehlerraten (T-Base-Calls) im nativen Sequenzkontext mittels Next-Generation-Sequencing (NGS) nachgewiesen (Schema 1).
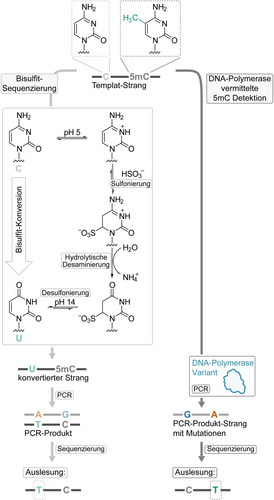
Vergleich der Bisulfit-Sequenzierung (links) und DNA-Polymerase-vermittelten 5 mC-Detektions (rechts) Strategie für den Nachweis von 5 mC.
Zunächst wurde ein Screening-basierter Entwicklungsansatz ausgearbeitet, um aus einer Mutantenbibliothek eine KTq-Variante mit erhöhtem Fehleinbau gegenüber methylierten Basen zu identifizieren. Die für das Screening verwendeten DNA-Polymerase-Varianten umfassten einzelne Aminosäuresubstitutionen mit einem breiten Spektrum an Mutationsstellen.25 Zusätzlich wurden funktionell vielversprechende Mutationsstellen rational kombiniert, um Doppelmutationsvarianten zu erzeugen, was zu insgesamt 970 zielgerichteten KTq-Varianten führte. Des Weiteren wurden über 2100 KTq-Varianten evaluiert, die Mehrfachmutationen enthielten. Diese Varianten wurden durch kombinatorisches Mischen aktiver Mutanten mit der RACHITT-Methode (Random Chimeragenesis on a Transient Template)26 und anschließender Vorselektion auf PCR-Aktivität generiert.25h
Die Bibliotheken wurden in Escherichia coli (E. coli) exprimiert und die DNA-Polymerase-Varianten direkt aus Zelllysaten in Primerverlängerungsreaktionen untersucht. Die Screeningreaktionen wurden parallel mit Oligonukleotiden der gleichen Sequenz durchgeführt, wobei entweder C oder 5 mC an der Stelle des ersten Einbaus als Templat verwendet wurde. Die 5′-Markierung der Primer mit zwei verschiedenen Fluoreszenzfarbstoffen, 6-Carboxyfluorescein (FAM) und Hexachlorfluorescein (HEX), sowie die unterschiedlichen 5′-Überhänge ermöglichten das Pooling von Primerverlängerungsreaktionen für die Multiplexanalyse mehrerer KTq-Varianten durch Kapillarelektrophorese (CE) (Abbildung 1a). Als Substrat für den Einbau einzelner Nukleotide wurde entweder das komplementäre 2′-Desoxyguanosin-5′-triphosphat (dGTP) (Basenpaarung) oder das nicht-komplementäre 2′-Desoxyadenosin-5′-triphosphat (dATP) (Fehlpaarung) verwendet. Wir wählten dATP, um KTq-Varianten mit erhöhter Fehleinbauaktivität zu identifizieren, da das KTq-Wildtyp-Enzym 2′-Desoxyadenosin-5′-monophosphat (dAMP) gegenüber C und 5 mC effizienter als die anderen nicht-komplementären Nukleotide einbaut (Abbildung S1).
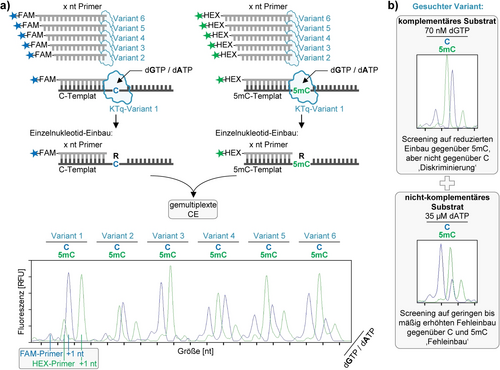
Screening nach KTq-Varianten mit erhöhtem Fehleinbau gegenüber 5 mC. a) Expressionslysate der DNA-Polymerase-Bibliothek wurden für den Einzelnukleotid-Einbau von dGMP oder dAMP gegenüber C und 5 mC verwendet. Die Verwendung von Primern unterschiedlicher Länge und 5′-Fluoreszenzmarkierung, FAM für Reaktionen mit C und HEX für Reaktionen mit 5 mC, ermöglichte die Multiplexanalyse von 12 Primerverlängerungsreaktionen in einer Kapillare durch CE. Die Primergröße korreliert mit der Migrationszeit der Oligonukleotide, und die Unterschiede zwischen FAM- und HEX-markierten Primern gleicher Länge resultieren aus der unterschiedlichen elektrophoretischen Mobilität der Fluorophoren. Die Rechtsverschiebung des Fluoreszenzsignals in den Elektropherogrammen entspricht dem Nukleotideinbau (R=Purinbase), und der Vergleich der Intensität des Verlängerungspeaks mit der Intensität des nicht verlängerten Primers erlaubt eine qualitative Bewertung der Effizienz der Primerverlängerungsreaktion. b) Gesuchte Eigenschaften vielversprechender KTq-Varianten: hohe Diskriminierung von 5 mC für den dGMP-Einbau und eine geringe bis mäßige Effizienz für dAMP-Fehleinbau gegenüber C und 5 mC. Die gezeigten Elektropherogramme wurden während der Screening-Experimente erhalten und sind beispielhaft für die zu erwartenden Ergebnisse dargestellt.
Um eine DNA-Polymerase mit einer erhöhten Fehlerrate gegenüber 5 mC zu identifizieren, suchten wir nach KTq-Varianten, die 5 mC durch einen verminderten Einbau von 2′-Desoxyguanosin-5′-monophosphat (dGMP) gegenüber der modifizierten Templatbase diskriminieren. Basierend auf früheren Studien zur Genauigkeit von DNA-Polymerasen27 vermuteten wir, dass die verminderte Effizienz dieser KTq-Varianten beim Einbau des komplementären Nukleotids gegenüber 5 mC den Anteil des Einbaus nicht-komplementärer Nukleotide erhöhen würde.Um diesen Fehleinbau zu fördern, wurden die KTq-Varianten zusätzlich auf einen gering bis mäßig erhöhten dAMP-Fehleinbau, jedoch ohne Diskriminierung von C und 5 mC, untersucht (Abbildung 1b). Dies sollte zu einer katalytisch aktiven DNA-Polymerase-Variante führen, die Mutationssignaturen gegenüber 5 mC erzeugt, aber kanonische Nukleotide ohne erhöhte Fehlerraten prozessiert. Wir wählten daher nur KTq-Varianten aus, die dAMP mit vergleichbarer Effizienz gegenüber C und 5 mC falsch einbauten und dabei nicht mehr als 50 % des Primers durch dAMP-Einbau verlängerten, was etwa 3 % der ursprünglich untersuchten KTq-Varianten entsprach. In einem weiteren Screeningschritt wurden nur diejenigen KTq-Varianten ausgewählt, die im Vergleich zum KTq-Wildtyp eine gleiche oder höhere Diskriminierungseffizienz gegenüber 5 mC aufwiesen. In dieser finalen Screening-Runde wurden 12 vielversprechende KTq-Varianten identifiziert, die 0,4 % der Mutantenbibliothek ausmachten (Abbildungen S2 und S3). Diese Varianten wurden aus der kombinatorischen RACHITT-Mutantenbibliothek gewonnen und entsprechend ihrer Position in der Bibliothek benannt (Tabelle S1). Nach Genexpression und Reinigung der 12 vielversprechendsten Enzyme wurden die untersuchten Eigenschaften in weiteren Experimenten mit Primerverlängerungsreaktionen verifiziert (Abbildung S4).
Des Weiteren führten wir Experimente zum Einbau mehrerer Nukleotide durch, um die Elongationskapazität der DNA-Polymerase zu bewerten, indem wir 2′-Desoxycytidin-5′-triphosphat (dCTP) als zweites einzubauendes Nukleotid hinzufügten. Die Effizienz der DNA-Polymerasen bei der Verarbeitung korrekt eingebauter Nukleotide wurde anhand der Primerverlängerung nach dGMP-Einbau gemessen und zeigte, dass die KTq-Varianten 5 mC durch eine reduzierte Verlängerung nach dGMP-Einbau gegenüber der methylierten Templatbase diskriminierten (Abbildung S5). Die Verarbeitung und Verlängerung eines falsch eingebauten Nukleotids stellt jedoch eine Herausforderung für DNA-Polymerasen dar, was zu einer Verzögerung der DNA-Synthese führt und somit zur allgemeinen Replikationsgenauigkeit beiträgt.25g, 28 Daher konzentrierten wir uns auf die Auswahl von DNA-Polymerase-Mutanten, die Fehlbasenpaarungen effizient verlängern können. Die Analyse der Primerverlängerung nach dAMP-Fehleinbau ergab, dass lediglich die KTq-Varianten RII G7, RIII H20, RIV A8 und RIV D15 in der Lage waren, eine Fehlbasenpaarung effizient zu verlängern (Abbildung S6). Die Überprüfung der PCR-Effizienz und der Robustheit der DNA-Polymerasen bei reduziertem dGTP-Gehalt (komplementäres Nukleotid) bestätigte, dass alle KTq-Varianten PCR-aktiv waren und das korrekte PCR-Produkt amplifizierten (Abbildung S7). Unter den untersuchten Varianten wiesen RIII H20, RIV A8 und RIV D15 die höchste PCR-Effizienz auf (Abbildung S7c). Somit vereinten nur die KTq-Varianten RIII H20, RIV A8 und RIV D15 die gewünschten Kriterien, nämlich die Diskriminierung von 5 mC, eine erhöhte Fehleinbaurate, eine ausreichende Elongationsfähigkeit der nicht-komplementären Basenpaarung sowie Aktivität bei der DNA-Synthese.
Anschließend wurden die KTq-Varianten RIII H20, RIV A8 und RIV D15 auf die Erzeugung von 5 mC-abhängigen Mutationssignaturen während der DNA-Synthese untersucht (Abbildung 2a). Zur Verstärkung dieser Signaturen wurde zunächst eine lineare Amplifikation eines unmodifizierten C- und eines CpG-modifizierten 5 mC-Templates in Gegenwart einer reduzierten Konzentration des komplementären Nukleotids durchgeführt (Abbildung S8). Eine verringerte dGTP-Konzentration erhöht die Bildung von Fehlbasenpaarungen gegenüber Cs, da weniger komplementäres Nukleotid für den Einbau zur Verfügung steht.29 Wir vermuteten, dass die verminderte Effizienz der KTq-Varianten beim Einbau von dGMP gegenüber 5 mC die Bildung von Fehlbasenpaarungen an methylierten Stellen begünstigen könnte. Ein unausgeglichener dNTP-Pool fördert somit sowohl den Fehleinbau von dAMP als auch die Diskriminierung von 5 mC, was die spezifische Detektion von 5 mC erleichtern würde. Die erhaltenen PCR-Produkte dienten anschließend als Template für die Erstellung von NGS-Bibliotheken und wurden sequenziert. Für die Datenanalyse und die eindeutige molekulare Identifikatoren (Unique Molecular Identifiers, UMI)-basierte Fehlerberechnung wurde ein selbstgeschriebener KNIME30 Workflow verwendet (Abbildung 2b).
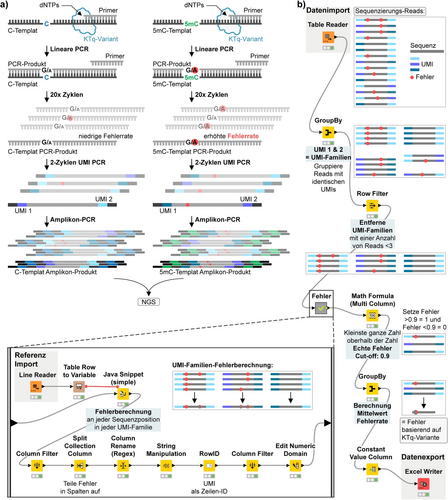
Strategie zum Nachweis von 5 mC durch Auslesen einer erhöhten Fehlerrate. a) KTq-Varianten wurden zur Primerverlängerung gegenüber C und 5 mC in einer linearen PCR verwendet. Die erhöhte Fehlerrate gegenüber 5 mC (durch Punkte gekennzeichnet) ist auf selektive Fehlbasenpaarungen während der DNA-Synthese durch dAMP-Fehleinbau zurückzuführen. Die Methylierungsinformation liegt in der Produkt-DNA als Mutation, d. h. als fehlgepaarte A-Base, vor. Das PCR-Produkt dient als Vorlage für die Markierung mit eindeutigen molekularen Identifikatoren (UMI) (Sequenzierprimer-Bindungsstelle in dunkelgrau und eine Farbe pro UMI). Fehler werden während der Amplifikation in der Amplikon-PCR konserviert (Sequenzieradapter P5 und P7 in schwarz, Indizes in blau oder grün) und die Bibliotheken werden mittels NGS analysiert. b) KNIME-Datenanalyse-Workflow zur Fehlerberechnung. Fehler, die auf fehlerhaften Einbau durch die KTq-Variante zurückzuführen sind, können durch die dargestellte UMI-Strategie von Sequenzierfehlern unterschieden werden. Die Reads werden nach identischen UMI-Sequenzen in UMI-Familiengruppen sortiert (eine UMI-Familie entsteht aus einem linearen PCR-Produkt), und UMI-Familien mit mindestens drei Reads werden weiterverarbeitet. Die Reads werden mit der Referenzsequenz verglichen, und die Fehlerrate an jeder Position innerhalb jeder Familie wird berechnet. Anschließend werden nur Fehler, die in 90 % aller Reads innerhalb einer Familie auftreten (Cut-off für echte Fehler: 0,9), für die Berechnung der mittleren Fehlerrate über alle UMI-Familien berücksichtigt. Dazu werden die echten Fehler der UMI-Familien auf 1 gesetzt, und die berechnete mittlere Fehlerrate stellt den von der KTq-Variante erhaltenden echten Fehler dar. Die Detektion von 5 mC wird dadurch ermöglicht, dass eine erhöhte Fehlerrate gegenüber 5 mC-Positionen abgelesen wird.
Tatsächlich zeigte die KTq-Variante RIV A8 eine bis zu doppelt so hohe Fehlerrate an mehreren methylierten CpG-Stellen, C24, C32 und C72, im 5 mC-Templat im Vergleich zum C-Templat (Abbildung 3a, schwarze Pfeile). Eine detaillierte Analyse der Fehlerraten nach der Amplifikation beider Templaten zeigte, dass die DNA-Polymerase bevorzugt nicht-komplementäre Basenpaare gegenüber C-Basen einbaute, mit einem durchschnittlichen Fehler von 3,2 % gegenüber C im Vergleich zu 0,27 % gegenüber allen Nicht-C-Basen. Darüber hinaus variierten die Fehlerraten an verschiedenen Sequenzpositionen, einschließlich Nicht-C-Basen. Die Genauigkeit der DNA-Polymerase hängt stark vom Kontext der DNA-Sequenz und den Sekundärstrukturen ab,31 was auch entsprechend die Eigenschaften der gewünschten Fehlbasenpaarungen beeinflussen würde. In diesem Zusammenhang ist es besonders bemerkenswert, dass RIV A8 beide Template an identischen Positionen mit vergleichbarer Genauigkeit prozessierte. Umso beachtlicher ist es daher, dass ein signifikanter Fehlerunterschied ausschließlich beim Vergleich von methylierten und unmethylierten CpG-Stellen festgestellt werden kann (Abbildung 3b links, schwarze Pfeile). An den methylierten CpG-Stellen C24, C32 und C72 zeigte die RIV A8-Variante eine durchschnittlich 16,5-fach höhere Fehlerratendifferenz (ΔFehlerrate) im Vergleich zum KTq-Wildtyp-Enzym, das nur an den Positionen C24 und C32 eine geringfügig höhere ΔFehlerrate gegenüber 5 mC aufwies (mit einer durchschnittlichen ΔFehlerrate von 5,9 % an methylierten CpG-Stellen für RIV A8 und einer durchschnittlichen ΔFehlerrate von 0,36 % für den KTq-Wildtyp an methylierten CpG-Stellen) (Abbildung S9). Bemerkenswerterweise zeigte die anfängliche Charakterisierung durch Primerverlängerungsreaktionen keine signifikanten Unterschiede in der Diskriminierung von 5 mC zwischen dem KTq-Wildtyp und RIV A8 (Abbildung S10 und Tabelle S2). Die Mutationssignaturanalyse zeigt, dass RIV A8 generell fehleranfälliger ist, bestätigt aber, dass ein erhöhter dAMP-Fehleinbau (nachgewiesen als T-Basen-Calls) zu einer erhöhten Bildung von Fehlbasenpaaren gegenüber 5 mC führt (Abbildungen 3b rechts und S11). Darüber hinaus war RIV A8 in der Lage, die spezifische 5 mC-abhängige Fehlersignatur in einem Wiederholungsexperiment mit vergleichbarem Ergebnis zu reproduzieren, was es zu einem geeigneten Kandidaten für die Detektion von 5 mC durch erhöhten Fehleinbau macht (Abbildung S12). Die Mutanten RIII H20 und RIV D15 zeigten ebenfalls einen erhöhten dAMP-Fehleinbau gegenüber 5 mC, allerdings mit geringerer Effizienz im Vergleich zu RIV A8 (Abbildungen S13 und S14).
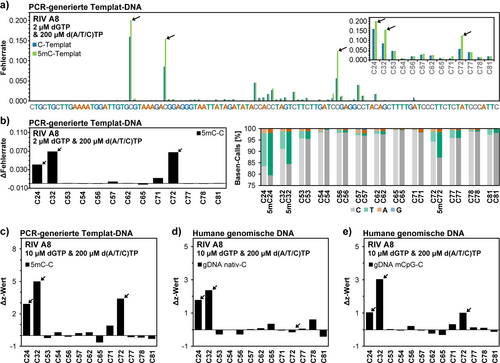
Die KTq-Variante RIV A8 erkennt 5 mC durch einen erhöhten Fehleinbau. a) Fehlerraten linearer PCR-Produkte, die von der RIV A8-Variante in Gegenwart eines unausgeglichenen dNTP-Pools mit 2 μM dGTP (komplementär) und 200 μM d(A/T/C)TP (jeweils) durch Amplifikation entweder des unmodifizierten C- oder des modifizierten 5 mC-Templats (50 pM) erzeugt wurden. b) Fehlerratendifferenz (ΔFehlerrate) (links) und Mutationssignatur (rechts) an C- und 5 mC-Positionen von RIV A8. Die Fehlerratendifferenzen wurden berechnet, indem die Fehlerraten, die durch die Amplifikation des C-Templates, von den Fehlerraten die durch die Amplifikation des 5 mC-Templats erhalten wurden, subtrahiert wurden. c) Differenz der z-Werte (Δz-Wert) an den C- und CpG-Positionen, berechnet durch Subtraktion der z-Werte der standardisierten Fehlerraten des C-Templates von den z-Werten der standardisierten Fehlerraten des 5 mC-Templates (3 fM). Die z-Werte wurden berechnet, indem die Differenz zwischen der Fehlerrate und der mittleren Fehlerrate von ausschließlich C-Basen-Positionen durch die Standardabweichung der letzteren dividiert wurde. Die Fehlerraten stammen von linearen PCR-Produkten, die von der RIV A8-Variante in Gegenwart eines unausgeglichenen dNTP-Pools mit 10 μM dGTP (komplementär) und 200 μM d(A/T/C)TP (jeweils) erhalten wurden. d) Differenz der z-Werte an den C- und CpG-Positionen, berechnet durch Subtraktion der z-Werte der standardisierten Fehlerraten des C-Templates von den z-Werten der standardisierten Fehlerraten der nativ methylierten humanen genomischen DNA (gDNA nativ) (62,5 ng gDNA). RIV A8 detektiert CpG-Methylierung an C24 und C32 durch erhöhten Fehleinbau gegenüber 5 mC in nativer gDNA. An der CpG-Stelle C72 (nur geringe Methylierung) wird kein erhöhter Fehleinbau detektiert. e) Differenz der z-Werte an den C- und CpG-Positionen, berechnet durch Subtraktion der z-Werte der standardisierten Fehlerraten des C-Templats von den z-Werten der standardisierten Fehlerraten der CpG-methylierten gDNA (gDNA mCpG). RIV A8 erkennt Methylierung an den CpG-Stellen C24, C32 und C72 durch erhöhten Fehleinbau gegenüber 5 mC in gDNA mCpG. Alle CpG-Stellen sind mit schwarzen Pfeilen markiert. Die Fehlerraten stammen von NGS-Bibliotheken, die einmal erstellt und mit dem Illumina NextSeq 2000 System sequenziert wurden.
Erfreulicherweise erkannte die RIV A8-Variante auch methylierte CpG-Stellen in humaner genomischer DNA (gDNA), indem sie die ortsspezifischen 5 mC-abhängigen Signaturen erzeugte (Abbildung 3d und 3e, schwarze Pfeile). Aufgrund der inhärent niedrigen Kopienzahl der gDNA wurden nach der Reaktion mit der RIV A8-Variante nur geringe Mengen an linearen PCR-Produkten erhalten. Daher wurde eine exponentielle Amplifikation mit einer High-Fidelity-DNA-Polymerase durchgeführt, um die für die Erstellung der NGS-Bibliothek erforderliche Konzentration zu erreichen. Dies führte zu einem Gemisch aus Ausgangsmaterial und RIV A8-Produkt (Abbildung S15), das eine weitere Datenanalyse erforderte. Basierend auf der früheren Beobachtung, dass RIV A8 identische Positionen in jedem Templat mit der gleichen Genauigkeit verarbeitete und ein signifikanter Fehlerunterschied nur durch die Methylierung verursacht wurde (Abbildung S14c), standardisierten wir die absoluten Fehlerraten zu angepassten z-Werten (Abbildung S16). Unter Verwendung der z-Werte bestätigte die Berechnung der Differenz zwischen dem modifizierten 5 mC- und dem unmodifizierten C-Templat (Δz-Wert) die Fähigkeit von RIV A8, 5 mC im PCR-generierten Templat zu erkennen. Dies wurde durch einen durchschnittlichen Anstieg des Δz-Wertes um das 11,4-fache an den methylierten CpG-Stellen C24, C32 und C72 im Vergleich zu den unmethylierten C-Positionen im 5 mC-Templat deutlich (Abbildungen 3c und S17a). Trotz der anspruchsvollen und komplexen Natur der Amplifikation von gDNA konnte RIV A8 Methylierungsgrade von mehr als 50 % an den CpG-Stellen C24 und C32 in nativ methylierter gDNA (gDNA nativ) nachweisen (Abbildung S8). Dies zeigte sich durch eine 9,5-fache Erhöhung des mittleren Δz-Wertes gegenüber diesen CpG-Stellen im Vergleich zu C-Basen (Abbildungen 3d und S17b). Darüber hinaus bestätigte RIV A8 die Detektion von DNA-Methylierung durch einen erhöhten Fehleinbau an den CpG-Stellen C24, C32 und C72 in der enzymatisch CpG-methylierten gDNA (gDNA mCpG), was durch eine 10,7-fache Erhöhung des mittleren Δz-Wertes im Vergleich zu C-Basen sichtbar wurde (Abbildungen 3e, S8 und S17c).
In den letzten Jahren wurde gezeigt, dass das 5 mC-Oxidationsprodukt 5-Hydroxymethylcytosin (5hmC) nicht nur als Zwischenprodukt im 5 mC-Demethylierungsweg fungiert, sondern auch als eigenständiger stabiler epigenetischer Marker wirkt.32 Die Evaluierung von RIV A8 zur Erkennung von DNA-Hydroxymethylierung ergab, dass die KTq-Variante 5hmC von unmodifizierten C-Basen durch ähnlich veränderte Einbaueigenschaften wie bei 5 mC unterscheidet (Abbildungen S18–S20).
Der Mechanismus, durch den die multimutante Variante RIV A8 5 mC diskriminiert und einen erhöhten dAMP-Fehleinbau aufweist, ist derzeit unklar. Da jedoch keine einzelne oder rational entworfene Variante die erforderlichen Eigenschaften aufweist, spekulieren wir, dass die individuellen Effekte der distalen Mutationen synergistisch zu einer Veränderung der Genauigkeit der DNA-Polymerase beitragen (Abbildung S21). Um eine DNA-Polymerase zu entwickeln, die 5 mC effizient erkennt und gleichzeitig katalytisch aktiv bleibt, mussten kritische DNA-Polymerase-Eigenschaften überwunden werden die zur Replikationsgenauigkeit beitragen, wie z. B. zuverlässige 5 mC-Verarbeitung, Substratselektivität und Diskriminierung bei der Verlängerung nicht-komplementärer Nukleotide. Die Mutationen könnten daher auf verschiedenen mechanistischen Ebenen wirken. Substitutionen in der Nähe des aktiven Zentrums, wie E507A, A570K und I614M, könnten den korrekten Einbau gegenüber methylierten Basen der Templat-DNA verhindern,25c, 33 und ebenfalls den Fehleinbau fördern, indem sie die korrekte Ausrichtung der Reste bei Fehlbasenpaarungen aufrechterhalten und so den Einbau und die Verlängerung falscher Nukleotide erleichtern.25d, 34 Vereint könnte dies zu einer verringerten katalytischen Effizienz für den dGMP-Einbau gegenüber 5 mC führen und somit die Toleranz für die Bildung von Fehlbasenpaarungen an dieser Stelle erhöhen. Darüber hinaus könnten die mutierten Reste N483K, S515K, K540N und V586G, die in Kontakt mit dem Primer und/oder Templat-Strang stehen, die DNA-Bindung verstärken und Übergangszustände stabilisieren, wodurch die Verlängerung der Fehlbasenpaarung unterstützt und die DNA-Synthese verbessert wird.25d, 35
Zusammenfassend stellen wir hier einen neuen Ansatz zur Detektion von 5 mC in hoch methylierter DNA vor, der die Eigenschaften der entdeckten KTq-Variante RIV A8 nutzt.
Unterstützende Informationen
Die Autoren haben zusätzliche Referenzen in den Hintergrundinformationen angegeben.36, 37
Danksagung
MH und AF bedanken sich für die Unterstützung durch die Konstanzer Research School Chemical Biology. Wir danken Martina Adam-Wels für die technische Unterstützung beim Klonen und Screening der Mutantenbibliotheken. Diese Arbeit wurde teilweise von der Deutschen Forschungsgemeinschaft MA 2288/22-1 finanziert. Open Access Veröffentlichung ermöglicht und organisiert durch Projekt DEAL.
Interessenkonflikt
MH and AM are inventors of a PTC application made by the University of Konstanz based on the most relevant discoveries of this research work.
Open Research
Data Availability Statement
High-throughput sequencing data are available in the NCBI GEO database, record GSE233599 and GSE270850. The KNIME workflow group created for processing of the sequencing data is available at https://hub.knime.com/-/spaces/-/~qkjAV-djKlP3LTTv/. All data needed to evaluate the conclusions in the paper are present in the paper and/or in the Hintergrundinformationen. Crystal structure information is available at the RCSB Protein Data Bank under the accession number 1QSS. Additional data may be requested from the authors.