Hochauflösende NMR-spektroskopische Bestimmung der dynamischen Struktur von Membranproteinen
Abstract
15N-Spinrelaxationsraten können wichtige Informationen über Fernstruktur und interne Bewegungen von Membranproteinen bereitstellen. Kombiniert mit einem verbesserten Berechnungsprotokoll offenbart die mittels Relaxationsraten abgeleitete Struktur des 283 Reste umfassenden humanen spannungsabhängigen Anionenkanals ein anisotrop geformtes Fass mit einer fest verankerten N-terminalen Helix. Diese Arbeit eröffnet somit einen NMR-spektroskopischen Ansatz, um die Struktur und Dynamik von Membranproteinen der Säugetiere mit hoher Exaktheit und Auflösung zu bestimmen.
Menschliche Membranproteine sind für eine Vielzahl essenzieller Funktionen verantwortlich und Hauptwirkziel von Medikamenten. Derzeit werden bei der Strukturbestimmung von Membranproteinen durch Röntgenkristallstrukturanalyse und Kryo-Elektronenmikroskopie große Fortschritte gemacht. Dennoch zeigt sich verstärkt, dass Informationen über die Struktur und Dynamik von Säugetier-Membranproteinen benötigt werden, um deren Funktion zu verstehen.1 Da die NMR-Spektroskopie Zugang zu beiden dieser entscheidenden Proteineigenschaften bei physiologisch relevanten Temperaturen2 bietet, kann sie eine Schlüsselrolle in diesem Zusammenhang spielen.
Die Herausforderung für die NMR-Spektroskopie bei der Untersuchung der dynamischen Struktur von Membranproteinen besteht in der Spärlichkeit von Fernstrukturinformationen, weil in Systemen mit hohem Molekulargewicht Deuterierung zwingend ist. Um die reduzierte Anzahl von Kern-Overhauser-Effekt(NOE)-basierten Abstandsbedingungen zwischen Protonen zu kompensieren, ist die Verwendung dipolarer Restkopplungen (residual dipolar couplings, RDCs) sehr hilfreich.3 Gerade für große integrale Membranproteine kann es sich jedoch als schwierig erweisen, partiell geordnete NMR-Proben mit ausreichender Qualität und Stabilität herzustellen. Alternativ kann durch kovalent gebundene paramagnetische Spin-Marker induzierte Relaxationsverstärkung wertvolle Informationen liefern,4 wobei jedoch die von paramagnetischen Markern abgeleiteten Entfernungen nur grobe Schätzungen über die Faltung des Proteinrückgrats zulassen.5
Wir begegnen diesen Beschränkungen hier, indem wir die Fernordnung nutzen, die in der Kernspinrelaxation anisotrop taumelnder Makromoleküle kodiert ist6 – ein Ansatz, der bislang noch nicht auf Membranproteine angewendet wurde. Mithilfe einer Kombination von heteronuklearen Relaxationsraten6 mit wissensbasierten Potentialen7 bestimmen wir die Dynamik und Struktur des 283 Reste umfassenden humanen spannungsabhängigen Anionenkanals (hVDAC1)8 in Lösung in einer bislang unerreichten Auflösung.
Das zweidimensionale 1H-15N-TROSY-Spektrum9 ist das zentrale Spektrum der NMR-basierten Analyse von Membranproteinen, die durch Detergenzien in Lösung gebracht wurden. Die hohe Qualität dieser Korrelationskarte bildet die Grundlage der Zuordnung der Resonanzen des Proteinrückgrats und damit der Bestimmung der Struktur und Dynamik von Membranproteinen. Abbildung 1 a zeigt das 1H-15N-TROSY-Spektrum der E73V-Mutante von hVDAC1, die bei 37 °C einen im Vergleich zum Wildtyp-Protein geringeren Konformationsaustausch10 und gute Signaldispersion in LDAO-Mizellen aufweist (Abbildung 1 a). Mithilfe von TROSY-basierten Tripel-Resonanz-Experimenten waren wir in der Lage, 92 % der HN-,N-,Cα-,C′-Resonanzen des Proteinrückgrates sequenzspezifisch zuzuordnen.10b Auf Basis der experimentellen chemischen Verschiebungen wurden die Torsionswinkel ψ, ϕ des Rückgrates und der Seitenketten-Winkel χ1 abgeschätzt (Abbildung S1 der Hintergrundinformationen). Zusätzlich lieferten HMQC-basierte 15N-editierte NOESY-Experimente 656 1HN-1HN-NOE-basierte Informationen über Proton-Proton-Abstände mittlerer und langer Entfernung (Abbildung S2). In Kombination ermöglichten diese Daten die Bestimmung der dreidimensionalen Struktur von E73V-hVDAC1. Dazu wurde die Struktur ausgehend von der gestreckten Polypeptidkette mittels in Xplor-NIH11 implementierter simulierter Abkühlung gefaltet (im weiteren Protokoll 1 genannt; Tabelle S1). Das so erhaltene Ensemble von 20 Strukturen niedrigster Energie wies eine mittlere quadratische Abweichung von 2.5 Å für das Rückgrat auf (Abbildung 1 b und Tabelle S2). Ähnlich wie im Fall anderer NOE-basierter Strukturen von hVDAC110a, 12 waren sowohl die Form des Fasses als auch die Position und Konformation der N-terminalen Helix nur mit mittlerer Genauigkeit bestimmt (Abbildung 1 b, Tabelle S3 und Abbildung S3).
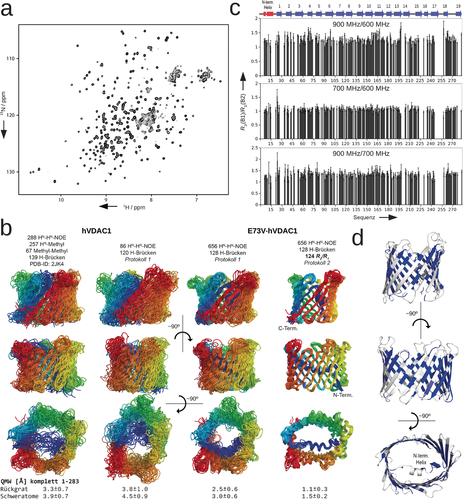
In 15N-Spinrelaxationsraten kodierte dynamische und strukturelle Informationen. a) Bei 900 MHz aufgenommenes 2D-1H-15N-TROSY-Spektrum der 283 Reste umfassenden E73V-Mutante von hVDAC1 in LDAO-Mizellen. b) Galerie von NMR-basierten Ensembles des β-Fasses des humanen Membranproteins VDAC1 (vom N- zum C-Terminus in blauer bis roter Färbung). Von links nach rechts: in12 beschriebene hVDAC1-Struktur; mit dem Protokoll 1 (simulierte Abkühlung) auf der Basis der in Lit. 10a veröffentlichten NMR-Daten berechnete hVDAC1-Struktur; aus NMR-Daten mit Protokoll 1 erhaltene E73V-hVDAC1-Struktur; das Ensemble von E73V-hVDAC1, das mit Protokoll 2 gegen die experimentellen 15N-R2/R1-Raten verfeinert wurde, zeigt einen drastischen Abfall in der Konformationsbreite. c) Reste-spezifische Verhältnisse der 15N-R2-Relaxationsraten, R2i(B1)/R2i(B2), bei drei Magnetfeldern. Die Sekundärstrukturelemente sind darüber mit blauen Pfeilen (β-Stränge) bzw. roten Rechtecken (Helix) gekennzeichnet. Die Fehlerbalken beziehen sich auf einfache Standardabweichung. d) Konformation von E73V-hVDAC1 niedrigster Energie, die gegen 124 15N-R2/R1-Raten verfeinert wurde. Reste, deren R2/R1-Raten während der Strukturverfeinerung genutzt wurden, sind blau gefärbt.
Um die Genauigkeit und Präzision der E73V-hVDAC1-Koordinaten zu verbessern und das dynamische Verhalten des Proteinrückgrates zu untersuchen, wurden 15N-Spinrelaxationsmessungen bei magnetischen Feldern zwischen 60 und 90 MHz Stickstoff-Frequenz durchgeführt. Da die chemischen Verschiebungen in diesem Experiment TROSY-basiert kodiert wurden (Abbildung S4),13 konnten 537 Paare von R1- und R2-Relaxationsraten des 32 kDa großen E73V-hVDAC1 mit mittleren Fehlern von 2.7 % bei 60 MHz, 3.1 % bei 70 MHz und 4.4 % bei 90 MHz erhalten werden (Abbildung 1 c,d und Abbildung S5 sowie Tabellen S4 und S5). Die Messungen bei drei unterschiedlichen Magnetfeldern wurden zur Bewertung der Gesamtqualität der erhaltenen Daten sowie zur Ermittlung möglicher Beiträge von Bewegungen auf der μs–ms-Zeitskala genutzt. Zudem ermöglichen Relaxationsraten bei unterschiedlichen Feldern eine genauere Bestimmung von Bewegungen auf der ps–ns-Zeitskala.2a Die Analyse der Relaxationsraten im Rahmen des modellfreien Ansatzes (model free approach, MFA)14 zeigte, dass die meisten Reste in den 19 β-Strängen von VDAC auf der ps–ns-Zeitskala unbeweglich sind (Ordnungsparameter nach Lipari und Szabo S2>0.85 und Korrelationszeit interner Bewegungen τi<500 ps; Abbildung S6).
Nur wenige Reste der N-terminalen β-Stränge zeigten erhöhte Werte für schnelle interne Bewegung. Zudem wurden keine Anzeichen für Beiträge durch chemischen Austausch zu den 15N-R2-Raten gefunden (Abbildung 1 c sowie die Analyse der Relaxationszeiten mithilfe erweiterter Modelle für lokale Bewegung; siehe Hintergrundinformationen). Die höchste Flexibilität wurde in den Schleifen beobachtet, besonders in der langen Schleife zwischen den β-Strängen 18 und 19 (Abbildung S6), die an der Bindung von β-Nicotinamid-Adenin-Dinukleotid beteiligt ist.12 Insgesamt zeigt diese Analyse, dass Proteinbewegungen in großen Membranproteinen mit einer Rotationskorrelationszeit von bis zu 40 ns mit hoher Genauigkeit bestimmt werden können.
Die Abhängigkeit der transversalen und longitudinalen Relaxationsraten von der Anisotropie der Rotationsdiffusion liefert nicht nur Informationen über interne Bewegungen, sondern auch über die Fernstruktur.6b Da durch den modellfreien Ansatz gezeigt werden konnte, dass die E73V-Mutante von hVDAC1 im Gegensatz zum Wildtyp-Protein sehr starr ist (Abbildung S6), wurden deren 15N-R2/R1-Raten zur Strukturverfeinerung genutzt. Die Selektion starrer Reste mit Ordnungsparametern von S2>0.85, wie sie hauptsächlich in sekundären Strukturelementen gefunden werden, ergab 124 R2/R1-Verhältnisse bei jedem der drei verwendeten Magnetfelder (124×3=372 R2- und 372 R1-Raten, insgesamt damit 744 Relaxationsraten). Um die in den Relaxationsraten enthaltenen Strukturinformationen zu extrahieren, müssen die Rotationskorrelationszeit (τc), die Anisotropie (A) und die Rhombizität (η) des Diffusionstensors a priori bekannt sein. Diese Parameter wurden durch modellfreie Analyse14 der NOE-basierten Struktur niedrigster Energie von E73V-hVDAC1 abgeschätzt (Tabelle S6). Um den Einfluss der gewählten Ausgangskoordinaten auf die Bestimmung der Parameter des Diffusionstensors zu testen, wurde die Analyse unter Verwendung der Röntgenstruktur von Maus-VDAC1 (mVDAC1; PDB-ID: 3EMN)15 wiederholt (Tabelle S6). Anschließend wurde die Struktur von E73V-hVDAC1 gegen die experimentellen 15N-R2/R1-Raten verfeinert. Dafür wurde Protokoll 2 (Tabelle S7) entwickelt, das die wissensbasierten Potentiale torsionDB und HBDB7 verwendet, die die Geometrie von Torsionswinkeln und Wasserstoffbrücken optimieren. Diese wissensbasierten Potentiale beschränken den dem Protein zur Verfügung stehenden Konformationsraum auf die physikalisch plausibelsten Konformationen, die in den bestaufgelösten Röntgenstrukturen zu finden sind.16 Zudem verhindern sie eine Überanpassung von Orientierungsbeschränkungen, besonders beim Auftreten experimenteller Ausreißer.6b Als weiterer Test der Auswirkung der Parameter des Diffusionstensors auf die erhaltenen Koordinaten wurden die Tensorparameter A und η, die aus der R2/R1-verfeinerten E73V-hVDAC1-Struktur abgeleitet wurden, um ±0.2 Einheiten bzw. −50 % und +100 % variiert. Alle Rechnungen konvergierten zu sehr ähnlichen dreidimensionalen Strukturen (Abbildung S7), in Übereinstimmung mit Ergebnissen globulärer Proteine und deren Komplexe.6, 17
Die unter Verwendung von Protokoll 2, das die wissensbasierten Potentiale anwendet, aus der Verfeinerung gegen die experimentellen longitudinalen und transversalen NMR-Relaxationsraten gewonnene dreidimensionale (3D-)Struktur von E73V-hVDAC1 ist in Abbildung 1, Spalte 4 gezeigt. Die 20 Strukturen niedrigster Energie erfüllen die Beschränkungen für Entfernungen und Torsionswinkel und weisen zudem verbesserte Z-Werte gegenüber der Ausgangsstruktur auf (Abbildung S8 und Tabelle S8). Das gesamte Ensemble gruppiert sich eng um eine einzige Konformation mit einer mittleren quadratischen Abweichung von 1.1 Å für das Rückgrat für alle 283 Reste (Abbildung 1 b). Jeder der 19 β-Stränge ist dabei wohldefiniert, und die N-terminale α-Helix, die vermutlich eine Rolle beim Schaltmechanismus von VDAC spielt,8 ist starr an der Wand des Fasses verankert (Abbildung 1 b,d). Der beweglichste Teil der Struktur ist das Verbindungsstück, das die N-terminale Helix mit β-Strang 1 verknüpft. Gegenüber der Kristallstruktur von mVDAC1 (Abbildung 2 a, blaue Struktur) ist die Form des Fasses in E73V-hVDAC1 (Abbildung 2 a, pinke Struktur) deutlich anisotroper. Diese Beobachtung folgt direkt aus den experimentellen R2/R1-Raten, da diese besser zur Struktur von E73V-hVDAC1 (R2=0.94) in Lösung als zur Kristallstruktur von mVDAC1 (R2=0.83) passen (Abbildungen 2 b und S9). Die größten Abweichungen zwischen den experimentellen Raten und den aus der Kristallstruktur von mVDAC1 berechneten Werten wurden in den β-Strängen 9 und 10 gefunden (Abbildung S9). Hervorzuheben ist, dass die durch R2/R1-Verfeinerung erhaltene, elliptische Form von E73V-hVDAC1 (QMW Cα, Reste 26–283=2.1 Å) einer anisotropen Fassform, die in einer 100-ns-Moleküldynamik-Trajektorie von in eine Membran eingebettetem mVDAC1 beobachtet wurde (Abbildung 2 c, schwarze Struktur),10b stärker ähnelt als der Ausgangs-Kristallstruktur von mVDAC1 (QMW Cα, Reste 26–283=3.3 Å;15 Abbildung 2 a, blaue Struktur). Dies lässt darauf schließen, dass die E73V-Mutation und/oder die Mizellenumgebung eine Konformation stabilisieren, die die VDAC1-Pore laut Moleküldynamik-Simulationen in einer Phospholipid-Doppelschicht annehmen kann. Studien haben bereits gezeigt, dass Mutationen von E73 das durch Hexokinase-I vermittelte Schalten von VDAC und die Inhibierung von Apoptose beeinflussen.18
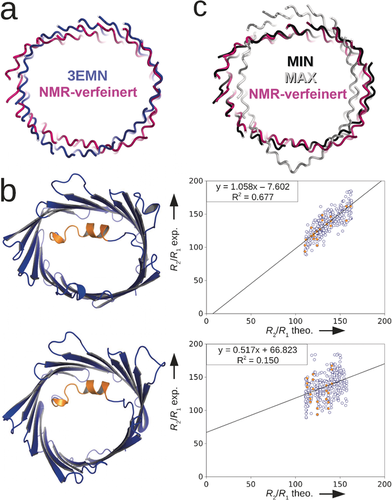
Überprüfung der hochaufgelösten Struktur von E73V-hVDAC1 in Lösung. a) Überlagerung der gegen die R2/R1-Raten verfeinerten 3D-Struktur von E73V-hVDAC1 (rosa) mit der Kristallstruktur von mVDAC1 (PDB-ID: 3EMN) (blau).15 Das Fass von E73V-hVDAC1 ist, bei Ansicht von oberhalb der Membran, stärker elliptisch geformt. b) Korrelation der experimentellen R2/R1-Raten dreier magnetischer Felder (skaliert entsprechend der theoretischen Feldabhängigkeit) mit den aus der Kristallstruktur von mVDAC1 (PDB-ID: 3EMN; untere Teilabbildung) bzw. mit der Struktur von E73V-hVDAC1 in Lösung (obere Teilabbildung) zurückgerechneten Werten. Die Reste der N-terminalen Helix sind ebenso wie ihre berechneten R2/R1-Raten in Orange dargestellt. Die experimentellen R2/R1-Raten stimmen besser mit der Struktur von E73V-hVDAC1 überein (vgl. auch Abbildung S9). c) Die Form des Fasses von E73V-hVDAC1 (rosa) ist identisch zu einer der beiden maximalen Auslenkungen (schwarz und grau), die in einer 100-ns-Moleküldynamik-Trajektorie von in einer Phospholipid-Doppelschicht eingebettetem mVDAC1 beobachtet wurden.10b
Proteinbewegungen verkomplizieren grundsätzlich die Interpretation von NMR-Daten auf Basis einer einzigen Konformation. Dies gilt sowohl für Relaxationsraten als auch für andere NMR-Parameter wie NOEs und RDCs. Die Verwendung von RDCs zur Strukturbestimmung ist jedoch sogar noch problematischer als die von 15N-Spinrelaxationsraten, weil RDCs auf einer viel größeren Zeitskala für dynamische Vorgänge empfindlich sind. Ausgehend davon hat der hier vorgeschlagene Ansatz, d. h. die Strukturverfeinerung auf der Basis von Relaxationsraten, den einzigartigen Vorteil, dass sowohl dynamische als auch strukturelle Informationen erhalten werden. Es ist somit möglich, flexible Reste zu identifizieren und von der Strukturverfeinerung auszuschließen (wie es in der hier beschriebenen Anwendung auf VDAC geschehen ist). Im Falle eines dynamischeren Proteins wäre es nötig, noch mehr Reste auszuschließen, einzelne Regionen könnten aber dennoch von der Verfeinerung gegen die experimentellen 15N-R2/R1-Raten profitieren. Zusätzlich sind Ensemblerechnungen, die Gebrauch von 15N-R2/R1-Raten und anderen NMR-Parametern machen, vorstellbar.19 Wir haben damit einen NMR-spektroskopischen Ansatz entwickelt, um die Struktur von Membranproteinen in hoher Exaktheit und Auflösung zu ermitteln.
Acknowledgements
M. und Ł. Jaremko möchten sich bei Prof. Andrzej Ejchart für das Teilen seiner Zeit und seiner Kenntnisse über Kernspinrelaxation bedanken. Wir danken R. Briones und B. L. de Groot für die MD-Koordinaten. Diese Arbeit wurde durch ein Stipendium des FCI (für S.V.), durch die DFG (SFB 803, für C.G. und M.Z.), durch die ERC-Finanzhilfevereinbarung 282008 (für M.Z.) und ein Erwin-Neher-Nobel-Stipendium (für M.J.) unterstützt. Das gegen die R2/R1-Raten verfeinerte NMR-Ensemble von E73V-hVDAC1 wurde zusammen mit experimentellen Daten in der Protein Data Bank (PDB-ID: 5JDP und 30065) hinterlegt.