DNA-Mimikry-Foldamer-Erkennung eines chromosomalen Proteins
Abstract
Es wurden helikale aromatische Oligoamid-Foldamere mit anionischen Seitenketten synthetisiert, die die Gesamtform und Ladungsoberflächenverteilung der DNA nachahmen. Ihre Wechselwirkungen mit dem chromosomalen Protein Sac7d, einem nicht-sequenzselektiven DNA-Binder, der die DNA knickt, wurden mittels Oberflächenplasmonenresonanzspektroskopie (SPR), isotherme Titrationskalorimetrie (ITC), Zirkulardichroismus (CD), Schmelzkurvenanalyse, Rasterkraftmikroskopie (AFM) und Kernspinresonanz (NMR), sowie durch Röntgenkristallographie untersucht. Es zeigte sich, dass die Foldamere besser an Sac7d binden als ein DNA-Duplex vergleichbarer Länge. Die Wechselwirkung ist diastereoselektiv und findet an der DNA-Bindungsstelle statt. Die Kristallographie zeigte, dass die DNA-Mimikry-Foldamere einen eigenen Bindungsmodus haben und dass sie an Sac7d binden können, ohne geknickt zu werden.
Das Konzept der biomolekularen Mimikry ist zwar bekannt, wird aber nicht immer vollständig verstanden. Ein Nachahmer reproduziert einige Merkmale des Biomoleküls, von dem er ursprünglich inspiriert wurde. Eine Mimikry soll jedoch in der Regel auch das Original durch seine Unterschiede übertrumpfen. Eine perfekte „Kopie“, die mit dem Original übereinstimmen würde, wäre nicht besser. Die Entwicklung von Mimikry-Produkten, die sich strukturell vom Original unterscheiden, ist daher eine Herausforderung, kann aber auch lohnend sein, da die Unterschiede eine Chance darstellen, das Original zu übertreffen. α-Helix-Mimetika1 die auf bestimmte Proteinoberflächen abzielen, und DNA-Analoga,2 die besser mit der DNA hybridisieren als die DNA selbst, sind prominente Beispiele für erfolgreiche Biomimikry. Im Gegensatz dazu sind Moleküle, die die Oberfläche einer DNA-Doppelhelix nachahmen und DNA-Protein-Wechselwirkungen kompetitiv hemmen, trotz ihres Potenzials für pharmakologische Anwendungen wenig entwickelt worden.
DNA-Mimikry kommt in der Natur in Form von DNA-nachahmenden Proteinen vor, die in der Regel reich an Asparaginsäure- und Glutaminsäureresten sind.3 Inspiriert durch die Natur wurde die Verwendung künstlicher Proteine als DNA-Mimikry in Angriff genommen, aber noch nicht umfassend betrieben.4 Die sogenannten Decoy-Oligonukleotide (ODNs) können auch als Zielproteine für DNA-bindende Proteine wie Transkriptionsfaktoren verwendet werden. Diese ODNs werden modifiziert, um ihre Bioverfügbarkeit zu erhöhen, aber sie binden möglicherweise nicht besser an ihr Ziel als ihre natürlichen Gegenstücke.5 In diesem Zusammenhang haben wir abiotische aromatische Oligoamide mit anionischen Phosphonat-Seitenketten vorgestellt, die sich in Wasser zu individuellen Helices falten, deren Form und Ladungsverteilung die Form und Ladungsverteilung der doppelsträngigen B-DNA nachahmen.6 Die Sequenzen 1 und 2 (Schema 1, Abbildung S1) entsprechen beispielsweise B-DNA-Duplexen mit acht bzw. sechzehn Basenpaaren (bp). Da sie jedoch bereits monomer sind, können sie nicht zu zwei Einzelsträngen schmelzen, wie dies bei DNA der Fall ist. Solche Foldamere weisen keine anderen Sequenzmerkmale auf als das Alternieren von M- und Q-Monomeren (Schema 1). Es hat sich gezeigt, dass sie die Zusammensetzung des Chromatins beeinflussen und die Progression des Zellzyklus stören6c und sich mit hoher Affinität an einige nicht-sequenzselektive DNA-bindende Proteine binden, was zu einer kompetitiven Hemmung therapeutisch relevanter Enzyme wie Topoisomerase 1 (Top1) und HIV-Integrase 1 (HIV-IN) selbst in Gegenwart eines großen Überschusses an DNA-Substrat führt.6b Diese Effekte zeigen eine gewisse Selektivität: die Bindung ist nicht bei allen nicht-sequenzselektiven DNA-bindenden Proteinen effektiv.6b Darüber hinaus hat sich gezeigt, dass strukturelle Veränderungen der Foldamere die Selektivität für Top1 oder HIV-IN verstärkt.6a Der Mangel an detaillierten strukturellen Informationen darüber, wie diese Moleküle Proteine erkennen, behindert jedoch die Entwicklung besserer und selektiverer Binder, auch für Proteine, die nicht selektiv DNA-Sequenzen binden. Hier präsentieren wir die erste Strukturuntersuchung der Wechselwirkungen zwischen einem DNA-Mimikry-Foldamer und – als Modellsystem – Sac7d, einem bakteriellen chromosomalen Protein, von dem bekannt ist, dass es DNA nicht-sequenzenselektiv im hyperthermophilen Archaeon Sulfolobus acidocaldarius bindet und knickt.7 In dieser Studie zeigen wir, dass Foldamere die DNA bei der Anvisierung der DNA-Bindungsstelle von Sac7d übertreffen, indem sie eine andere Bindungsorientierung annehmen und nicht geknickt werden.
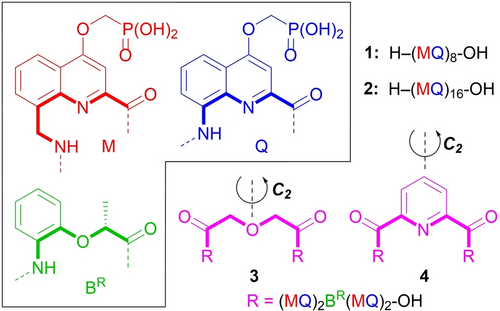
In dieser Studie verwendete Aminosäure-Chinolinmonomere M, Q sowie BR und Oligoamidsequenzen 1–4. Die fettgedruckten Bindungen zeigen den inneren Rand der helikal gefalteten Konformationen an. Die Sequenzen 1 und 2 haben sowohl einen N- als auch einen C-Terminus. Die Sequenzen 3 und 4 sind C2-symmetrisch und haben zwei C-Termini.
Sac7d wurde als Modellprotein für Strukturuntersuchungen mit DNA-Mimikry-Foldameren ausgewählt, da es eine hohe strukturelle Stabilität und Löslichkeit aufweist und umfangreiche Literatur über seine Wechselwirkungen mit der DNA vorliegt.7b, 7e, 7g, 7j In einem ersten Test untersuchten wir die Bindung von 1 und 2 an Sac7d mittels SPR mit dem auf dem Chip immobilisierten Protein und den Foldameren in der mobilen Phase (Abbildung 1a, Abbildungen S2–S3). Die steady-state-Daten zeigten einen Kd-Wert im einstelligen mikromolaren Bereich. Dies ist eine schwächere Bindung als die ähnlicher Foldamer an z. B. HIV-IN,6b aber sie ist mit der zweistelligen mikromolaren Bindung von Sac7d an Kalbsthymus-DNA zu vergleichen.7a Allerdings passten die Sensorgramme nicht gut zur Kinetik eines einzelnen Bindungsereignisses. Ein möglicher Grund dafür ist, dass 1 und 2 kein stereogenes Zentrum enthalten und daher als racemische Mischung aus linkshändigen (M) und rechtshändigen (P) Helices existieren, was mindestens zwei Arten von Wechselwirkungen mit dem Protein ermöglicht. ITC bestätigte die Bindung von Sac7d an 1 im niedrigen bis submikromolaren Bereich (Abbildung S4) und zeigte, dass die Wechselwirkung enthalpiegetrieben ist (ΔH=−6.9 kcal.mol−1, −TΔS=1.57 kcal.mol−1), im Gegensatz zur Sac7d-DNA-Wechselwirkung, die entropiegetrieben und endotherm ist.7k, 8
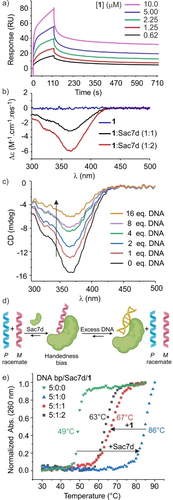
Beleg der Bindung von 1 an Sac7d. a) SPR-Sensorgramme der Wechselwirkung zwischen einem mit His6-Tag versehenem Sac7d und 1 (pH 7.4, 25 °C). Die Sensorgramme wurden nach Abzug des Signals der Referenz-Durchflusszelle aufgezeichnet. b) CD-Spektren von 1 (40 μM in 50 mM NH4HCO3, pH 8.5, 20 °C) in Gegenwart von 0, 1 oder 2 Äquiv. von Sac7d nach 24-stündiger Äquilibrierung. c) CD-Spektren einer 1 : 1-Mischung von 1 und Sac7d in Gegenwart zunehmender Mengen einer 10-bp-DNA. d) Cartoon-artige Darstellung der Gewichtung der Händigkeit der Helix in 1 bei der Bindung an Sac7d und der kompetitiven Assoziation der DNA. e) DNA-Schmelzprofile von poly[dAdT]*poly[dAdT] (500 μM bp-Konzentration) in Gegenwart verschiedener molarer Verhältnisse von Sac7d und 1, gemessen bei 260 nm.
Da 1 und 2 achiral sind, zeigen ihre CD-Spektren kein Signal. Bei der Inkubation von 1 und 2 mit Sac7d trat jedoch eine CD-Bande bei 365 nm als Funktion der Zeit (Abbildung 1b, Abbildungen S5–S6) auf, was auf eine Gewichtung der Händigkeit der aromatischen Helix durch Umwandlung eines enantiomeren Konformers in das andere hinweist. Das negative Vorzeichen der Bande deutet auf eine Präferenz für die M-Helix hin,9 d. h. für das Enantiomer, welches die rechtshändige B-DNA-Doppelhelix nachahmt (Abbildung S1). Diverse Schlussfolgerungen lassen sich aus dieser Beobachtung ziehen: (i) Die P- und M-Helices des DNA-Mimikry-Foldamers wandeln sich ineinander um, wenn auch langsam. Dies war unerwartet, wenn man die Längen von 1 und 2 und die kinetische Trägheit verwandter Foldamere bedenkt.10 Die schnellere Dynamik könnte auf die Abstoßung negativer Ladungen zurückzuführen sein; (ii) CD bestätigt, dass die Foldamere an Sac7d binden; (iii) die Wechselwirkung mit Sac7d ist diastereoselektiv und begünstigt das Enantiomer, das die B-DNA nachahmt – nicht sein Spiegelbild; und (iv) die Intensität der Bande (der Wert von Δϵ/Rest in Abbildung 1b) deutet darauf hin, dass die Beeinflussung groß ist (der geschätzte diastereomere Überschuss liegt bei 75 %).11 Die Ausgleichung der CD-Daten an eine 1 : 1-Bindungsisotherme (Abbildung S6) ergab einen Kd-Wert von 34 μM, der scheinbar höher ist als der durch SPR geschätzte Wert.12 Die CD-Spektroskopie bestätigte jedoch auch, dass die DNA weniger effektiv bindet als die Foldamere. Daher wurde ein Kompetitionsexperiment durchgeführt, bei dem ein DNA-Duplex von 10 bp zu dem bereits im Gleichgewicht befindlichen M-1-Sac7d-Komplex hinzugefügt wurde. Die Intensität der negativen CD-Bande bei 365 nm nahm allmählich ab, als sich der DNA-Sac7d-Komplex bildete und die freigesetzte Foldamer-Helix racemisierte (Abbildung 1c,d). Anhand der DNA-Menge, die erforderlich ist, um die CD-Intensität um die Hälfte zu verringern (~4 Äquivalente in Bezug auf das Foldamer), kann man abschätzen, dass 1 mindestens zehnmal besser bindet als die 10-bp-DNA (siehe unterstützende Informationen). Daraufhin haben wir getestet, ob ein DNA-Mimikry-Foldamer die Funktionen von Sac7d hemmen kann. Eine Wirkung der Bindung von Sac7d an B-DNA besteht darin, die thermische Stabilität der DNA erheblich zu erhöhen und die Schmelztemperatur von poly[dAdT]*poly[dAdT] um mehr als 30 °C zu steigern.7f Bei Zugabe von Foldamer 1 wurde dieser Effekt weitgehend abgeschwächt (Abbildung 1e). Diese Experimente zeigten auch, dass das Foldamer kein offensichtliches Schmelzen erfährt (Abbildung S7). Eine weitere Wirkung der Sac7d-Bindung ist die Knickung der DNA, die vermutlich zur Verpackung der DNA in Sulfolobus acidocaldarius beiträgt.7 Inspiriert von Arbeiten zu Abf2p, einem anderen DNA-bindenden und verdichtenden Protein,13 haben wir hochauflösendes AFM verwendet, um zu zeigen, dass Sac7d tatsächlich die DNA-Verdichtung fördert und dass dies in Gegenwart von Foldamer 1 rückgängig gemacht wird (Abbildungen 2, S8). Als Nächstes untersuchten wir die Erkennung von Sac7d durch DNA-Mimikry-Foldamere mit Hilfe der NMR-Spektroskopie. Zu diesem Zweck wurden die Foldamere 3 und 4 entwickelt und synthetisiert (Schema 1).14 Diese Sequenzen basieren auf einem zentralen Disäurelinker, der sie C2-symmetrisch, d. h. palindromartig macht, so dass sie unabhängig von ihrer Ausrichtung den gleichen Typ von Komplex mit Sac7d bilden würden. Außerdem besitzen sie zwei chirale BR-Reste (Schema 1), die ihre Händigkeit quantitativ in Richtung der M-Helix gewichten.11
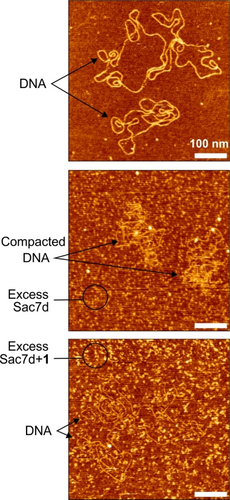
Repräsentative hochauflösende AFM-Bilder zeigen DNA allein (oben), verdichtete DNA in Gegenwart von Sac7d (Mitte) und unverdichtete DNA in Gegenwart von Sac7d und 1 (unten). Punkte, die überschüssigem Sac7d und Sac7d+1 entsprechen, lassen die mittleren und unteren Bilder etwas verschwimmen. Siehe Abbildung S8für Details.
Sequenz 3 besitzt einen aliphatischen, flexibleren zentralen Linker als der aromatische Linker von 4. Dieser Unterschied wurde im Zusammenhang mit der Fähigkeit von Sac7d eingeführt, DNA zu knicken, wobei ein flexibler Linker möglicherweise die Rolle eines Scharniers spielen könnte. Mehrere 2D- und 3D NMR-Experimente an 15N-markiertem Sac7d mit 1H-15N-HSQC (Hetronuclear Single Quantum Coherence), 15N-HSQC-NOESY (Nuclear Overhauser Effect Spectroscopy) und 15N-HSQC-TOCSY (Total Correlation Spectroscopy) ermöglichte die eindeutige Zuordnung des Protein-Backbones (Abbildung S9). Die Titrationen von Sac7d mit 3 und 4 wurden dann mittels 1H-15N-HSQC (Abbildungen 3a–c, Abbildungen S10–S13) verfolgt. Zwischen den beiden Foldameren 3 und 4 wurden keine signifikanten Unterschiede festgestellt. Beide verursachten ähnliche Veränderung der chemischen Verschiebung, was auf molekulare Assoziationen beim schnellen Austausch auf der NMR-Zeitskala hindeutet. Einige Änderungen der chemischen Verschiebung folgten einem monotonen Trend (gerade Pfeile in Abbildung 3a), der im Prinzip an eine 1 : 1-Bindungsisotherme angepasst werden konnte. Andere waren jedoch nicht monoton (geknickte Pfeile in Abbildung 3b), was darauf hinweist, dass sich mindestens zwei Komplexe mit unterschiedlicher Molekularität bilden, z. B. 1 : 1 und 1 : 2. Die Kartierung der chemical shift perturbations (CSPs) auf der Oberfläche der Sac7d-Struktur ergab, dass die von den Foldameren verursachten Perturbationen an und um die Beta-Faltblatt-DNA-Bindungsstelle auftreten und einige Schlüsselreste für die DNA-Erkennung (W24, V26, M29 und R42) betreffen. Im Gegensatz dazu blieb die gegenüberliegende Seite des Proteins im Wesentlichen unverändert. Dies spricht dafür, dass die DNA-Mimikry-Foldamere auch mit der DNA-bindenden Region von Sac7d interagieren.
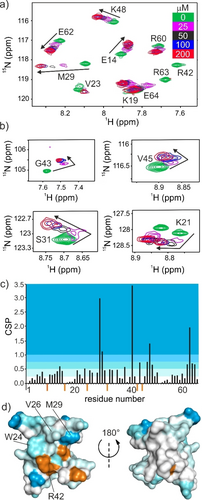
NMR-spektroskopischer Nachweis der Bindung von 3 an Sac7d in einer Konzentration von 100 μM in 50 mM Tris-d11-Puffer pH 7.5, 50 mM KCl, 10 % D2O. a, b) Teil der 1H-15N-HSQC-Titration von [15N]-Sac7d mit 3. Die farbige Skala zeigt eine zunehmende Konzentration von 3 an. c) CSPs des [15N]-Sac7d Backbone-Amid-1H-15N-HSQCs in Gegenwart von 3 (200 μM). Die CSPs wurden als Quadratwurzel des mittleren quadratischen Fehlers ((ΔδH)/0.14)2+(ΔδN)2)0.5 berechnet. d) Proteinoberfläche der Sac7d-Kristallstruktur7g eingefärbt nach den CSP-Werten wie im Schaubild c. Die Signale der orangefarbenen Reste verbreitern sich so stark, dass sie während der Titration verschwinden, was als starke chemical shift perturbation interpretiert wird.
Schließlich haben wir uns bemüht, Sac7d-Foldamer-Komplexe zu kristallisieren. Wie bei Komplexen zwischen DNA und nicht-sequenzselektiven DNA-bindenden Proteinen wurde dies durch die mögliche Entartung der Bindungsmodi über Rahmenverschiebungen von einem bp (hier ein MQ-Dimer) erschwert, die das Kristallwachstum nicht begünstigen. In 3 und 4 wurde diese Entartung durch die C2-Symmetrie und das Vorhandensein von anderen Einheiten als M und Q abgeschwächt. Es wurden Kristalle von Sac7d im Komplex mit 3 erhalten (Abbildungen 4a, S14), die bei 2.6 Å beugten. Die Struktur wurde durch molecular replacement unter Verwendung der Sac7d-Struktur7g und einem Molekülmodell des Foldamerfragments als Suchmodell verwendet wurden, gelöst (siehe unterstützende Informationen). Die verfeinerte Struktur zeigt die Bildung eines C2-symmetrischen 2 : 1-Komplexes mit zwei Proteinen, die an ein Molekül von 3 binden (Abbildung 4b). Diese Stöchiometrie könnte den Trend der CSPs bei den NMR-Titrationen erklären (Abbildung 4b). Eine kristallographische C2-Achse kreuzt also die Mitte des Linkers von 3. Die asymmetrische Einheit enthält daher die Hälfte dieses 2 : 1-Komplexes sowie die Hälfte eines zweiten Moleküls von 3, das nicht mit Sac7d interagiert. Bei der Kristallpackung handelt es sich um säulenförmige, pseudokontinuierliche Stapel von Foldamer-Helices, die den Stapeln von DNA-Duplexen ähneln, die häufig in Kristallen von Protein-DNA-Komplexen beobachtet werden (Abbildung S15). Die Struktur bestätigte, dass das Foldamer umfassend mit der DNA-Bindungsregion von Sac7d interagiert, d. h. über eine Kontaktfläche von 1500 Å. Die Ausrichtung der Helix von 3 ist jedoch fast senkrecht zu der der DNA (Abbildung 4c, d). Die Protein-Foldamer-Grenzfläche weist mehrere Wasserstoffbrückenbindungen auf, darunter ladungsverstärkte Wasserstoffbrückenbindungen, an denen Phosphonate und C-terminale Carboxylate von 3 beteiligt sind, sowie einige hydrophobe Kontakte (Abbildung 4e–h). Viele der beteiligten Reste sind auch Schlüsselreste für Sac7d-DNA-Interaktionen (z. B. Y8, W24 und R42, siehe Abbildung S16). Dennoch weist der Sac7d-3-Komplex eine einzigartige Geometrie auf, die die strukturellen Merkmale des Foldamers widerspiegelt. Zum Beispiel bildet BR einen hydrophoben Kontakt mit der Proteinoberfläche, der durch das Fehlen einer Seitenkette dieses Restes ermöglicht wird. Wenn BR durch Q oder M ersetzt würde, käme es zu einer sterischen Hinderung.
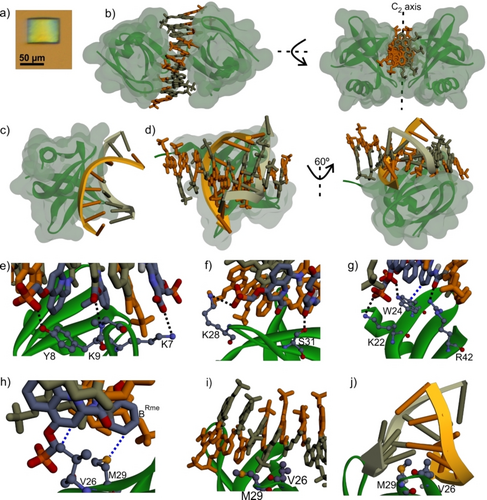
Kristallstruktur des Sac7d-3-Komplexes (PDB# 8CMN) und Vergleich mit einem Sac7d-DNA-Komplex (PDB# 1AZQ).7g a) Sac7d-3-Kristall in Röntgenbeugungsqualität. b) 2 : 1-Sac7d-3-Komplex. Sac7d-DNA-Komplex. c) Sac7d-DNA-Komplex. d) Überlagerung des Sac7d-3-Komplexes (Überlagerung von α-Kohlenstoffen) mit dem Sac7d-DNA-Komplex. e-i) Wichtige Wechselwirkungen im Sac7d-3-Komplex. Blaue und schwarze gestrichelte Linien markieren hydrophobe Kontakte bzw. H-Bindungen. j) DNA-Interkalation von V26 und M29 im Sac7d-DNA-Komplex. Sac7d ist in grüner Banddarstellung gezeigt. In b–d) wurde eine transparente grüne Iso-Oberfläche hinzugefügt. Die relevanten Reste sind in e–j) als Kugel und Stab dargestellt. In c), d) und j) ist die DNA in Form eines Bandes dargestellt. Foldamer 3 ist in Röhrenform mit abwechselnd beigen und orangenen Resten dargestellt. In e–j) haben die relevanten Reste klassische Atomfarben (C: dunkelgrau, O: rot, N: blau, P: orange).
Das scharfe 61°-Knicken der DNA durch Sac7d wird durch V26 und M29 vermittelt, die aus dem Protein herausragen und zwischen bps interkalieren (Abbildung 4j).7b Im Komplex mit 3 ragt die Seitenkette von M29 aus der intermolekularen Schnittstelle heraus, während die Seitenkette von V26 einen Hohlraum in der Foldamerstruktur ausfüllt, der durch die geringe Größe des zentralen Linkers entsteht (Abbildung 4i). Aufgrund der C2-Symmetrieachse, die den Linker kreuzt, sind es tatsächlich zwei V26-Seitenketten von zwei Proteinen, die diesen Hohlraum ausfüllen. Infolgedessen bindet die Foldamerhelix an Sac7d, ohne geknickt zu werden, eine Situation, von der man annimmt, dass sie bei DNA vorübergehend ist.7b, 7j
Alle Versuche, 4 im Komplex mit Sac7d zu kristallisieren, schlugen fehl: Ausfällungen wurden unter Bedingungen erhalten, unter denen Kristalle von Sac7d-3 wuchsen. Der größere zentrale Linker von 4 würde eindeutig den Raum ausfüllen, der von den beiden V26-Seitenketten der Struktur von 3 eingenommen wird, was die bei 3 beobachtete Bindungsgeometrie beeinträchtigen würde. Die NMR-Untersuchungen zeigten jedoch, dass 4 auch an Sac7d bindet. Die Doppelmutante Sac7d (V26A/M29A), der die für das DNA-Knicken verantwortlichen Seitenketten fehlen, wurde im Komplex mit 3 in einer Struktur kristallisiert, die ansonsten mit dem Sac7d-3-Komplex identisch ist (Abbildung S17). Darüber hinaus kristallisierte Sac7d (V26A/M29A) auch im Komplex mit 4. Die Struktur ähnelt wiederum der des Sac7d-3-Komplexes, wobei der zentrale Pyridinring von 4 den durch die V26A-Mutation freigewordenen Raum ausfüllt (Abbildung S18).
Zusammenfassend lässt sich sagen, dass DNA-nachahmende Foldamere eine neuartige Plattform darstellen, um das große Ensemble von Proteinen, die mit Nukleinsäuren interagieren, gezielt anzusprechen. Sie ergänzen andere aromatische Foldamer,15 Makrocyclen,16 and molekulare Pinzetten,17 die ebenfalls an Proteinoberflächen binden können. Die Struktur des Sac7d-3-Komplexes bietet eine solide Grundlage für das strukturbasierte Design des Foldamers, um die Bindungsaffinität und -spezifität zu verbessern und die inhärente Modularität von Foldamersequenzen zu nutzen. So betrachten wir beispielsweise die klare Protein-Foldamer-Ausrichtung als eine Möglichkeit, das Foldamer so zu gestalten, dass es nicht mit anderen DNA-bindenden Proteinen interagiert. Die spezifischen Besonderheiten des Foldamer-Sac7d-Komplexes wären schwer vorherzusagen gewesen, so dass weitere Designversuche ohne strukturelle Informationen ein eher schwieriges Unterfangen gewesen wären. Wir haben nun gezeigt, dass Protein-DNA-Mimikry-Foldamer-Komplexe kristallisiert werden können und auch ohne sequenzspezifische Erkennungsmerkmale detaillierte Strukturinformationen liefern. Derzeit weiten wir diese Arbeit auf andere Proteine aus, mit dem langfristigen Ziel, DNA-Mimikry-Foldamere zu schaffen, die hochspezifisch für ihr Protein-Targets sind.
Unterstützende Informationen
Die Autoren haben zusätzliche Referenzen in den unterstützenden Informationen angegeben.18-31
Autorenbeiträge
DD und JW führten die Proteinexpression und -aufreinigung durch. DD führte das Kristallwachstum und die kristallografische Analyse durch. VC und JW synthetisierten die Foldamere. VC führte CD-Experimente durch. JW führte ITC- und Schmelzstudien durch. LA und VC leiteten NMR-Experimente und Analysen. JW war an der TOCSY- und NOESY-Zuordnung beteiligt. MK und PT führten AFM-Beobachtungen durch und analysierten sie. IH überwachte das Projekt. Alle Autoren kommentierten und genehmigten das Manuskript.
Danksagung
Wir bedanken uns für die finanzielle Unterstützung durch die Deutsche Forschungsgemeinschaft (DFG) über die Projekte HU1766/2-1 und 325871075 (CRC1309-C7), durch den Europäischen Forschungsrat (ERC) im Rahmenprogramm Horizon Europe der Europäischen Union (Grant Agreement No. ERC-2021-ADG-320892), vom Freistaat Bayern im Rahmen der Exzellenzstrategie des Bundes und der Länder durch das ONE MUNICH-Projekt Munich Multiscale Biofabrication, vom Deutschen Akademischen Austauschdienst (DAAD, Promotionsstipendium für D. D.) und vom China Scholarship Council (CSC, Promotionsstipendium für J. W.). Diese Arbeit wurde durch die Einrichtungen und das Fachwissen der Plattform für biophysikalische und strukturelle Chemie (BPCS) der IECB, CNRS UMS3033, Inserm US001 und der Universität Bordeaux unterstützt. Wir danken L. Minder, T. Chakrabortty und V. Morozov für ihre Unterstützung bei SPR, Protein-expression und -reinigung bzw. ITC sowie P. K. Mandal für Voruntersuchungen. Wir danken L. McGregor (ID23-1, ESRF), M.S. Lopez (ID30B, ESRF) und I. Ibento (EMBL P13, Petra III, DESY) für die Unterstützung bei der Datenerfassung an Synchrotronstrahlrohren. Wir danken Prof. T. Carell für den Zugang zu seinem UV/Vis-Spektrophotometer für Tm-Messungen. Wir danken auch Prof. C. Y. Chen (Hubei University, Taiwan) für die freundliche Bereitstellung des Expressionsvektors für Sac7d und Sac7d V26A/M29A-Proteine. Open Access Veröffentlichung ermöglicht und organisiert durch Projekt DEAL.
Interessenkonflikt
Die Autoren erklären, dass keine Interessenkonflikte vorliegen.
Open Research
Data Availability Statement
Die Daten, die die Ergebnisse dieser Studie unterstützen, sind auf begründete Anfrage beim Autor erhältlich.