Gerichtete Evolution von Piperazinsäureeinbau durch eine Nichtribosomale Peptidsynthetase**
Eine frühere Version dieses Manuskripts ist auf einem Preprint-Server hinterlegt worden (https://doi.org/10.1101/2023.04.03.535426).
Abstract
Das Engineering biosynthetischer Enzyme wird zunehmend eingesetzt, um strukturelle Analoga von Antibiotika zu synthetisieren. Von besonderem Interesse sind nichtribosomale Peptidsynthetasen (NRPS), die für die Produktion wichtiger antimikrobieller Peptide verantwortlich sind. Hier beschreiben wir, wie durch gerichtete Evolution einer Adenylierungsdomäne eines Pro-spezifischen NRPS-Moduls dessen Substratspezifität vollständig auf die Nicht-Standard-Aminosäure Piperazinsäure (Piz) geändert wurde, welche eine labile N−N-Bindung enthält. Dieser Erfolg wurde durch UPLC-MS/MS-basiertes Screening kleiner, rational konzipierter Mutantenbibliotheken erzielt und kann vermutlich mit einer größeren Anzahl von Substraten und NRPS-Modulen repliziert werden. Die evolvierte NRPS ist in der Lage, ein Piz-beinhaltendes Gramicidin S-Analogon zu produzieren. Damit geben wir der zu früh verworfenen Idee neuen Auftrieb, dass weithin zugängliche Methoden mit niedrigem Durchsatz die Spezifität von NRPS in einer biosynthetisch nützlichen Weise verändern können.
Nichtribosomale Peptide (NRPs) und Derivate sind eine wichtige Klasse antimikrobieller Moleküle.1-3 Die zunehmende Gefahr durch antimikrobielle Resistenzen macht die Entwicklung und das Screening neuer bioaktiver Verbindungen erforderlich. NRPs werden von Enzymkomplexen, so genannten nichtribosomalen Peptidsynthetasen (NRPS), hergestellt, die wie ein Fließband arbeiten.1, 2 Auf diesem Fließband führen spezialisierte Domänen, die in Modulen organisiert sind – eins pro Aminosäure – unterschiedliche biochemische Aufgaben aus.1 Adenylierungsdomänen (A-Domänen) erkennen eine bestimmte Aminosäure, aktivieren sie unter ATP-Verbrauch und laden sie auf den flexiblen Phosphopantethein-Arm von Thiolierungsdomänen (T-Domänen). Kondensationsdomänen (C-Domänen) katalysieren die Bildung einer Peptidbindung zwischen zwei benachbarten, an T-Domänen gebundenen Substraten. Thioesterase-Domänen (TE-Domänen) lösen das Peptid durch Hydrolyse oder Zyklisierung von dem Enzymkomplex ab. GrsA und GrsB sind zwei NRPS-Proteine, die für die Synthese von Gramicidin S (GS, Abbildung 1a) verantwortlich sind.4 Zusammen synthetisieren das einzelne Modul von GrsA (d-Phe) und die vier Module von GrsB (Pro-Val-Orn-Leu) das Peptid Cyclo-(d-Phe-Pro-Val-Orn-Leu)2 in zwei biosynthetischen Iterationen (Abbildung 1a).
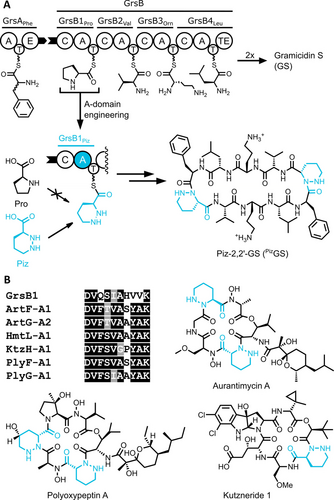
A. Biosynthese von Gramicidin S (GS) durch die beiden NRPS GrsA und GrsB.4 Durch Engineering der A-Domäne des Moduls GrsB1 werden die l-Pro-Reste in GS durch l-Piz ersetzt, was zu Piz-2,2′-GS (PizGS) führt. B. Vergleich der spezifitätsbestimmenden Reste von GrsB1 mit denen von Piz-A-Domänen. Die entsprechenden NRPs Aurantimycin A (Art),40 Polyoxypeptin A (Ply),41 und Kutznerid 1 (Ktz)42 enthalten Piz.
Der modulare Aufbau von NRPS hat verschiedene Engineering-Ansätze zur Herstellung neuer oder veränderter Peptide inspiriert.5-8 Versuche, ganze Module, Domänen oder kleinere Subdomänen zu ersetzen, führen häufig zu chimären Enzymen mit verringerter Aktivität oder vorzeitigem Abbruch der Peptidsynthese,9-11 was darauf hindeutet, dass auch Domäneninteraktionen berücksichtigt werden müssen.12, 13 Neuere Erkenntnisse zeigen, dass die langjährige Annahme, dass C-Domänen als sekundäre Spezifizitätsfilter fungieren, nicht generell zutrifft, was A-Domänen als ideale Ziele für NRPS-Engineering erscheinen lässt.14-16 Die Entdeckung eines “Spezifitätscodes” für A-Domänen17-19 war die Grundlage für ortsgerichtete Mutagenese der entsprechenden Reste, um die Substratspezifität von A-Domänen zu verändern. Erfolgreiche Anwendungen dieser Engineering-Strategie ermöglichten hauptsächlich konservative Substratänderungen oder führten zu verringerten Enzymaktivitäten.20-22 Nur wenige Beispiele für A-Domänen-Engineering, das die Akzeptanz von nicht natürlichen Aminosäuresubstraten ermöglicht, existieren.23-25 In jüngster Zeit werden in diesem Zusammenhang zunehmend gerichtete Evolution und Hochdurchsatz-Screening eingesetzt.26-31 Auch Hefe Display wurde verwendet, um große Mutantenbibliotheken zu analysieren, was zu einem effizienten Einbau von β-Aminosäuren und α-Hydroxysäuren führte.30, 31 Diese Technik erfordert jedoch für Click-Chemie geeignete Reste im Substrat und Proteinexpression auf der Hefeoberfläche, was die breitere Anwendbarkeit einschränkt.
Piz ist ein ungewöhnlicher NRPS-Baustein, für den nur etwa 150 Piz-haltige NRPs beschrieben wurden.32-34 Bei vielen von ihnen wird angenommen, dass Piz für die Bioaktivität verantwortlich ist. Piz ist ein Pro-Analogon mit einem sechsgliedrigen Ring, der ein Hydrazin enthält, was zu einer noch höheren strukturellen Steifheit als bei Pro führt (Abbildung 1a).35 Es wird aus Ornithin biosynthetisiert und dann in NRPs eingebaut.36, 37 Die Hydrazide von Piz sind ein seltenes Beispiel für N−N-Bindungen, die an der Bildung von Peptidbindungen beteiligt sind.38 Hier haben wir die Substratspezifität der A-Domäne des ersten Moduls der GS-produzierenden NRPS GrsB (GrsB1) von Pro auf Piz geändert. Die labile N−N-Bindung in Piz könnte eine Möglichkeit für eine spätere Modifikation und die Einführung kationischer Ladungen bieten, um die Membranselektivität von NRPs zu beeinflussen.39 Nach vier Evolutionsrunden und UPLC-MS/MS-basierten Screenings von nur 1.200 Mutanten hinsichtlich der Peptidproduktion haben wir eine GrsB1-Mutante erzeugt, die eine 140.000-fache Verbesserung der Piz-Präferenz aufweist.
Um den Ausgangspunkt unseres Engineering-Prozesses, das Modul GrsB1, zu charakterisieren, haben wir zunächst seine Substratselektivität bestimmt. Um den Einbau von Piz und Pro in Peptide durch GrsB1 zu analysieren, haben wir Diketopiperazine (DKPs) gemessen, die GrsB1 zusammen mit GrsAPhe in vitro bildet (Abbildung 2a). Die Messung der DKP-Bildung ist eine einfache Möglichkeit, alle relevanten, von NRPS-Modulen katalysierten Reaktionen (Adenylierung, Thiolierung, Kondensation) zu berücksichtigen. Die A-Domäne des nativen Moduls GrsB1Pro zeigte eine geringe DKP-Bildungsrate mit Piz als Substrat zusätzlich zu seiner Hauptaktivität für Pro (Abbildung 2b). Um unsere Designbemühungen zu lenken, lösten wir die Kristallstruktur der apo-Acore-Domäne von GrsB1 mit einer Auflösung von 2.6 Å (Abbildung 2c, Abbildung S1). Versuche, GrsB1 entweder mit gebundenem Piz oder Pro zu kristallisieren, waren erfolglos. Wir identifizierten die für die Substratspezifität der Piz-A-Domänen40-43 verantwortlichen Reste mithilfe von NRPSpredictor244 und verglichen diese Reste mit der Pro-A-Domäne von GrsB1 (Abbildung 1b). Auf der Grundlage der Unterschiede im aktiven Zentrum haben wir sechs Mutanten von GrsB1 erzeugt, um Piz zum bevorzugten Substrat zu machen. Diese Proteine wurden aus 400 mL Kulturen durch Ni-Affinitätschromatographie aufgereinigt und in vitro in Gegenwart von äquimolaren Konzentrationen von Pro und Piz (10 mM) auf DKP-Produktion getestet. Das Produktverhältnis wurde mittels UPLC-MS/MS bestimmt, und Mutanten, die eine erhöhte Spezifität für Piz aufwiesen, wurden ebenfalls in Kombination getestet (Abbildung S2). Aus diesen von natürlichen Piz-A-Domänen inspirierten Experimenten erhielten wir die Kombinationsmutante GrsB1-AYA mit drei Mutationen (H755A, V763Y, V764A). Während GrsB1-AYA im Vergleich zu GrsB1 eine 80-fach höhere Piz-Spezifität aufwies, blieb Pro das bevorzugte Substrat (Abbildung 2b und Abbildung S2), was einmal mehr bestätigte, dass allein das Kopieren von Spezifitätscodes nicht für das Engineering von A-Domänen ausreicht.
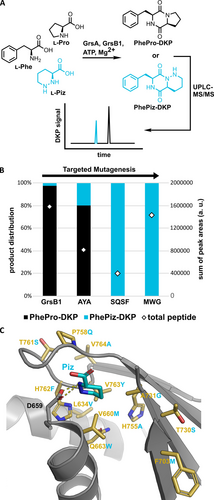
A. Bildung von DKP mit den NRPS-Modulen GrsA und GrsB1. Wenn Pro und Piz in äquimolarer Konzentration als Substrate für GrsB1 zugeführt werden, werden PhePro-DKP oder PhePiz-DKP gebildet. Das durch UPLC-MS/MS gemessene Verhältnis der jeweiligen Signale wird zum Vergleich der Piz-Spezifitäten von GrsB1-Mutanten verwendet. B. Gerichtete Evolution der Piz-Spezifität. Die in vitro-Bildung von PhePro- und PhePiz-DKP wird für GrsB1-Mutanten verglichen. Die mittels UPLC-MS/MS gemessenen Peakflächen der einzelnen Verbindungen sind als Anteil des Gesamtpeptids dargestellt (linke Achse). Die gemessenen Peakflächen der Produkte werden für jedes Protein kombiniert, um die Gesamtmenge des gebildeten Peptids zu bestimmen (rechte Achse). Die Werte stellen den Mittelwert biologischer Duplikate dar. C. Struktur der GrsB1-Substratbindungstasche von GrsB1, gelöst mit einer Auflösung von 2.6 Å. Um das Substrat im aktiven Zentrum zu platzieren, wurde die Struktur von GrsB1 zunächst mit der Struktur von PheA47 (PDB 1AMU) überlagert und in Pymol visualisiert. Anschließend wurde der Phe-Ligand in 1AMU durch Piz ersetzt, indem die α-Amino- und Carboxylgruppen überlagert wurden. Reste, die in GrsB1-MWG mutiert wurden (gelb, neue Identität in türkis) und das konservierte Asp659, das mit der α-Aminogruppe von Piz interagiert (grau), sind als Stäbchen dargestellt.
Um die Piz-Spezifität weiter zu verbessern, haben wir Positionen innerhalb eines Radius von 5 Å um die Substratbindungsstelle betrachtet, welche mit den in GrsB1-AYA mutierten Aminosäuren interagieren (als Aminosäurereste der “zweiten Schale” bezeichnet). Diese Positionen zeigten mehrere weniger konservierte Unterschiede zwischen den A-Domänen. Um dieser Ambiguität Rechnung zu tragen, entwarfen wir Mutantenbibliotheken mit limitierten Codonsets, die alle in der zweiten Schale der Piz-A-Domänen beobachteten Identitäten der Seitenketten abdeckten (Abbildung S3). Um diese Bibliotheken zu screenen, führten wir den DKP-Assay in Zelllysat von E. coli-Kulturen durch, die in 96-Well-Platten kultiviert wurden. Dem Lysat, welches die GrsB1-Mutanten enthielt, wurden Aminosäuren, ATP und Ni-affinitätsgereinigtes GrsA zugesetzt. Mittels UPLC-MS/MS konnten die Peptide von Hunderten von Varianten parallel quantifiziert werden. Durch Screening von 264 Mutanten identifizierten wir GrsB1-SQSF, das gegenüber GrsB1-AYA vier zusätzliche Mutationen aufweist (T730S, P758Q, T761S, H762F) und eine perfekte Spezifität für Piz zeigt. Die Gesamtproduktion von DKP war jedoch reduziert (Abbildung 2b, Abbildung S4).
Um die Ausbeute des von Piz abgeleiteten Peptids zu verbessern, führten wir eine Sättigungsmutagenese mit NNK-Codons45 an 13 Positionen durch, die an zuvor mutierte Aminosäuren angrenzten, und kombinierten die Treffer in einer weiteren Screening-Runde. Beim Screening von Lysat auf DKP-Bildung wurde die Variante GrsB1-MWG mit fünf zusätzlichen Mutationen identifiziert (L634V, V660M, Q663W, F703M, A731G; Abbildung S5, Abbildung S6). GrsB1-MWG hatte nicht nur eine perfekte Piz-Spezifität wie GrsB1-SQSF, sondern auch eine DKP-Bildungsaktivität, die mit der von GrsB1 mit Pro vergleichbar war (Abbildung 2b). Während GrsB1 0.5 % Piz bei äquimolarem Angebot von Piz und Pro als Substrat einbaute, hatte GrsB1-MWG keine messbare Pro-Aktivität, was eine ausgezeichnete molekulare Erkennung durch das veränderte Enzym zeigte. Um die Bedeutung der Mutationen besser zu verstehen, haben wir versucht, sowohl apo- als auch substratgebundene Formen der GrsB1-MWG-Mutante mit Aminosäuren und AMS-Inhibitoren48 zu kristallisieren, was jedoch nicht gelang. Die Übertragung der Mutationen auf die Struktur von GrsB1 ergab, dass die Reste, die die Piz-Substratspezifität kontrollieren, über die gesamte Bindungstasche verteilt sind, was auf eine umfassende Umgestaltung des aktiven Zentrums hindeutet (Abbildung 2c). Homologiemodellierung von GrsB1-MWG auf Grundlage der GrsB1-Kristallstruktur und Docking von Piz in die Bindetasche zeigte die potenzielle Rolle der Mutation V763Y, eine Wasserstoffbrückenbindung mit dem distalen Stickstoffatom von Piz einzugehen und so die Substratorientierung zu unterstützen (Abbildung S7b). Darüber hinaus könnte die Mutation P758Q die Flexibilität des benachbarten β13β14-Loops erhöhen, dessen Konformation für die Erkennung von β-Aminosäuren wichtig ist.30, 46
Wir haben die thermische Stabilität repräsentativer Varianten durch Thermal Shift Assays bestimmt (Abbildung S8). GrsB1-SQSF war das thermisch am wenigsten stabile Enzym mit einem Schmelzpunkt (Tm) von 40,7 °C, der abfiel gegenüber 46,4 °C bei GrsB1, was möglicherweise seine geringere Aktivität erklärt. Durch die zusätzlichen Mutationen in GrsB1-MWG wurde die Proteinstabilität weitgehend wiederhergestellt (Tm=43,8 °C). Diese Ergebnisse korrelieren mit der beobachteten Proteinausbeute nach der Ni-Affinitätsreinigung. Die Ausbeuten für GrsB1 und GrsB1-MWG waren vergleichbar (volumetrische Ausbeute: 51–62 mg/L), während GrsB1-SQSF eine geringere Produktion von löslichem Enzym zeigte (38 mg/L, Abbildung S9).
Um die Affinität von GrsB1 und GrsB1-MWG für ihre jeweiligen Liganden zu vergleichen, titrierten wir die Proteine mit stabilen Adenosylmonosulfamat (AMS)-Analoga48 der aktivierten Adenylate Pro-AMP und Piz-AMP und maßen die Schmelzpunkte. Die Veränderung der Schmelzpunkte wurde gegen die Ligandenkonzentration aufgetragen, um die Dissoziationskonstanten (KD’s, Abbildungen S10–12) zu bestimmen. Da sich die Synthese von Piz-AMS als schwierig erwies, wurde stattdessen Nipecotinsäure-AMS (Nip-AMS, Abbildung S12) verwendet. Piz und die β-Aminosäure Nip haben beide einen sechsgliedrigen Ring als Seitenkette, aber in Nip ist das Stickstoffatom in der α-Position von Piz durch ein Kohlenstoffatom ersetzt. Der KD für Nip-AMS stieg leicht von GrsB1 (14 μM) auf GrsB1-MWG (52 μM) an, während der KD für Pro-AMS dramatisch um das 2000-fache anstieg (0,7 μM bzw. 1,5 mM). Somit wurde ein Wechsel von der 20-fachen Pro-AMS- zur 29-fachen Nip-AMS-Präferenz durch Evolution für Piz-Präferenz erreicht. Die Verbindung zwischen Piz- und Nip-Spezifität deutet wahrscheinlich darauf hin, dass die β-Aminogruppe von Nip und das entsprechende Stickstoffatom in Piz eine analoge und wichtige Rolle bei der molekularen Erkennung spielen.
Darüber hinaus haben wir die Adenylierungskinetik für Pro und Piz mit den GrsB1-Varianten gemessen, welche während der gerichteten Evolution erzeugt wurden. Die Adenylierungskinetik wurde mittels MesG-Assay gemessen, welcher die Geschwindigkeit der Freisetzung von Pyrophosphat (PPi) während der Adenylierung enzymatisch an die Produktionsrate eines photometrisch nachweisbaren Nukleosidanalogons koppelt (Tabelle 1 und Abbildung S13).49 Die Zugabe von Hydroxylamin verhindert die Akkumulation von hemmendem Aminoacyladenylat.49, 50 Wie erwartet, hatten GrsB1 und GrsB1-MWG die höchste katalytische Effizienz (kcat/KM) für das native Substrat Pro (17,4 min−1 mM−1) bzw. das Zielsubstrat Piz (1,2 min−1 mM−1). Interessanterweise zeigten alle Mutanten ähnliche oder sogar höhere Umsatzraten (kcat) für Piz zwischen 2,8 und 5,0 min−1, welche alle im Bereich typischer Peptidbildungsraten von NRPS liegen.51, 52 Allerdings stieg die Michaelis–Menten-Konstante (KM) für Pro nach der ersten Runde der gerichteten Evolution dramatisch von 0,14 mM (GrsB1) auf 10 mM (GrsB1-AYA) an und konnte dann in den nachfolgenden Runden nicht mehr bestimmt werden. Im Gegensatz dazu fiel der KM für Piz von 9,2 mM in GrsB1 auf 2,4 mM in GrsB1-MWG. Insgesamt hat sich die gerichtete Evolution drastisch auf die molekulare Erkennung der strukturell ähnlichen Substrate Pro und Piz ausgewirkt, wobei biosynthetisch nützliche katalytische Raten beibehalten wurden.
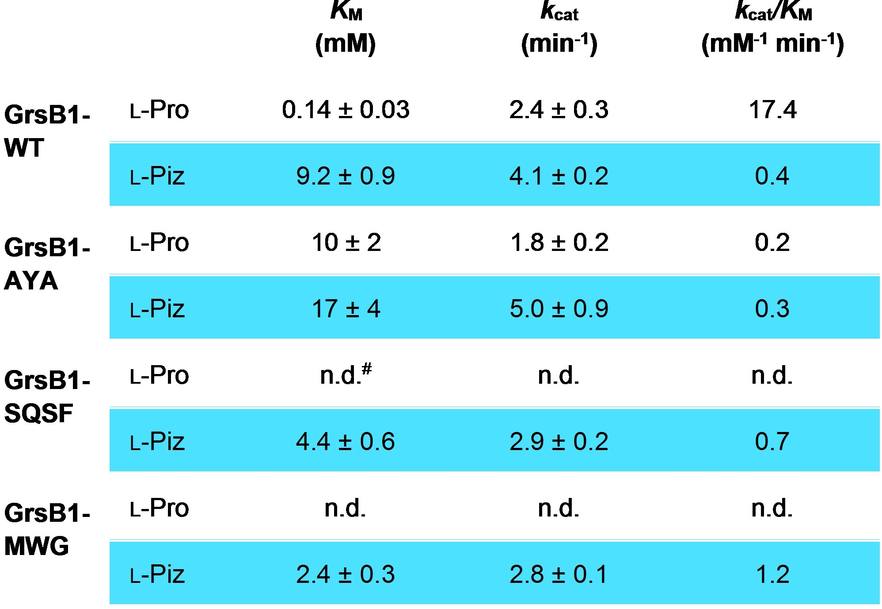
- [a] Die Adenylierungskinetik wurde mit dem MesG/Hydroxylamin-Assay bestimmt. Anfangsgeschwindigkeiten wurden gegen Substratkonzentrationen gemäß dem Michaelis–Menten-Modell aufgetragen, um Werte für KM und kcat zu erhalten (Abbildung S13). Jedes Enzym wurde als biologisches Duplikat gemessen. Die Fehler geben den Fehler des Fits an. #n.d.: nicht detektierbar.
Nach erfolgreichem Umstellen der Spezifität von GrsB1 haben wir das modifizierte NRPS-Modul verwendet, um PizGS in E. coli herzustellen. Zu diesem Zweck wurde das grsB1-Gen in grsB durch das modifizierte grsB1-MWG-Gen im Plasmid pSU18-grsTAB ersetzt, welches das gesamte GS-Biosynthesegencluster trägt.53 Das Gencluster wurde anschließend heterolog in E. coli unter Verwendung eines zuvor etablierten Protokolls exprimiert,53 während Piz dem Medium zugesetzt wurde. Überraschenderweise zeigte der evolutionäre Zwischenschritt GrsB1-SQSF-VM unter in vivo-Bedingungen die höchste Ausbeute an PizGS anstelle von GrsB1-MWG. In einer kurzen Optimierung der Produktionsbedingungen (Abbildung S14) stellten wir fest, dass die Zusammensetzung des Mediums, die Piz-Konzentration, die Schüttelgeschwindigkeit und das Verhältnis von Kultur zu Flaschenvolumen die Produktionsrate stark beeinflussen. Unter Verwendung von 50 mL LB-Medium in 2-L-Kulturgefäßen, die mit 400 U/min geschüttelt wurden, erzielten wir eine Ausbeute von 15 mg/L PizGS in Extrakten des Zellpellets, während keine Produktion von nativem GS nachweisbar war. Die Konzentrationen wurden durch Quantifizierung der UPLC-MS/MS-Signale unter Verwendung eines GS-Standards abgeschätzt, wobei angenommen wurde, dass GS und PizGS eine ähnliche Ionisierungseffizienz aufweisen. PizGS wurde identifiziert, indem die spezifische Massentransition 586>85 im MRM-Modus ([M+2H]2+ für das Vorläuferion von PizGS zum Iminiumion der Piz-Seitenkette nach Decarboxylierung als Tochterion, Abbildung 3a) nachgewiesen wurde sowie durch HRMS-Messung (berechnet: m/z=586,3712 [M+2H]2+ für C60H96N14O102+, gemessen: m/z=586.3713 [M+2H]2+, Abbildung 3c). Im Vergleich zu nativem GS erreichte die Produktion von PizGS ein Niveau von 20 % (Abbildung 3b). Wenn dem Produktionsmedium kein exogenes Piz zugesetzt wurde, wurden weder PizGS noch natives GS produziert, was erneut auf die hohe Selektivität für Piz gegenüber Pro im evolvierten GrsB1 hinweist. Zellextrakte, die PizGS enthalten, waren in der Lage, Liposomen, die einen selbstquenchenden Fluoreszenzfarbstoff enthalten, zu lysieren, wenn ein zuvor etabliertes Protokoll zur Messung der Membranaktivität von GS befolgt wurde. Extrakte, die PizGS enthielten, lysierten Liposomen schneller als Extrakte mit der gleichen Konzentration an nativem GS (Abbildung S15), was darauf hindeutet, dass die Piz-Substitution die Membranaktivität verbessert.
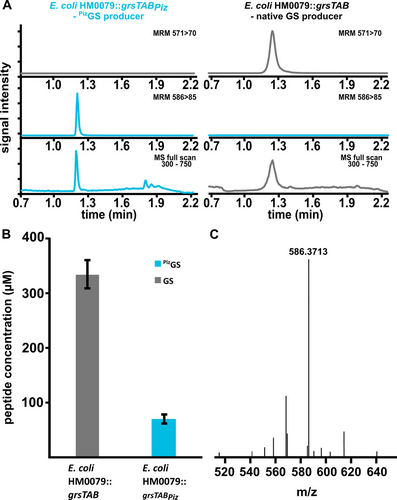
A. Totalionenchromatogramm für komplette MS-Scans sowie spezifische MRM-Scans für PizGS (586>85) und natives GS (571>70) für Rohextrakte von Zellpellets aus E. coli-Kulturen, die natives GS bzw. PizGS produzieren. Die Messungen sind auf den höchsten Peak jeder Messung (MS-Scan, 586>85 und 571>70) normiert. B. Vergleich der Produktausbeute in Zellextrakten für Kulturen, die natives GS (E. coli HM0079::grsTAB) bzw. PizGS (E. coli HM0079::grsTABPiz) unter optimierten Bedingungen (TB-Medium, 6 mM Ornithin, 30 °C, 5 Tage bzw. LB-Medium, 6 mM Ornithin, 15 mM Piz, 30 °C, 5 Tage) produzieren. Zur Berechnung der Konzentrationen wurde ein GS-Standard bekannter Konzentration verwendet. Die PizGS-Konzentration wurde unter der Annahme abgeschätzt, dass GS und PizGS vergleichbar gut ionisieren. Die Fehlerbalken geben die Standardabweichung aus drei biologischen Replikaten an. C. HRMS-Spektren für aufgereinigtes PizGS (berechnet: m/z=586,3712 [M+2H]2+, gemessen: m/z=586,3713 [M+2H]2+, Δ=0,2 ppm).
Die gerichtete Evolution ist ein leistungsstarkes Werkzeug zur Entwicklung maßgeschneiderter Enzymkatalysatoren.54-56 In dieser Arbeit haben wir eine gerichtete Evolutions-Strategie auf eine A-Domäne aus dem GS-Biosynthesecluster angewendet, um die nicht-proteinogene Aminosäure Piz zu integrieren. Inspiriert von natürlichen Spezifitätscodes haben wir eine rationale Mutagenese durchgeführt und die resultierenden Varianten durch zufällige Mutagenese weiter verfeinert. Dadurch haben wir eine vollständige Umkehr der Substratspezifität erreicht, nachdem wir insgesamt nur 1200 Mutanten untersucht haben. Interessanterweise trägt GrsB1-SQSF, die erste Mutante, die eine vollständige Veränderung der Substratspezifität zeigt (Abbildung 2b), Mutationen an Positionen, für die zuvor gezeigt wurden, dass sie die Selektivität für β-Aminosäuren oder α-Hydroxysäuren beeinflussen (GrsB1-SQSF: P758Q, T761S, H762F; TycAβPhe: T761C, I762L; TycAPLA: P758L; Nummerierung von TycAβPhe und TycAPLA entsprechend GrsB1).30, 31 Angesichts des Hauptunterschieds zwischen Pro und Piz, nämlich dem zusätzlichen Stickstoff in einer “β-ähnlichen” Position von Piz, überrascht es nicht, dass Positionen, die die β/α-Spezifität kontrollieren, auch die Piz/Pro-Spezifität beeinflussen können. Darüber hinaus wäre es zu erwarten, dass der Austausch der Aminogruppe von Pro durch ein Hydrazin in Piz sowohl die Kondensations- als auch die Adenylierungsreaktion stark beeinflusst. Da der “α-ähnliche” Stickstoff von Piz, der für die Peptidbindungsbildung verwendet wird, nur schwach nucleophil ist,57, 58 wurde spekuliert, dass C-Domänen möglicherweise zusammen mit A-Domänen ko-evolvieren müssen, um die Integration von Piz in Peptide zu erreichen.34 Die effiziente Integration von Piz in GS legt jedoch nahe, dass die unveränderte C-Domäne nicht gegen Piz diskriminiert. Daher könnte der Ersatz von Pro durch Piz auch bei anderen nichtribosomalen Peptiden möglich sein. Die Integration von Piz könnte weitergehende Modifikationen von Naturstoffen ermöglichen, wenn die N−N-Bindung in Piz chemisch gespalten werden kann, wodurch Piz in Orn umgewandelt würde.
Frühere, allein auf A-Domänenspezifitätscodes basierende Versuche der NRPS-Modifikation waren selten vollständig erfolgreich.20-22 Während das Übertragen des Spezifitätscodes hier auch kein zufriedenstellendes Ergebnis lieferte, konnten wir durch Kombination mit zufälliger Mutagenese die Substratspezifität selektiv verändern. Der resultierende Spezifitätscode (DMWSIGAYAK) ist ähnlich, aber nicht identisch mit dem Konsens der natürlichen Codes, der von Wei et al.34 für Piz-A-Domänen identifiziert wurde (DVFSVAxYAK). Die zweite und dritte Position sind in natürlichen Spezifitätscodes hoch konserviert (Val und Phe), jedoch unterschiedlich in unserem Fall (Met und Trp). Diese Unterschiede sind wahrscheinlich auf die Kontextabhängigkeit des Spezifitätscodes zurückzuführen und unterstreichen die Notwendigkeit eines unvoreingenommenen Mutanten-Screenings für die erfolgreiche gerichtete Evolution von A-Domänen.
Zur Lösung einer ähnlich schwierigen A-Domänen-Engineering-Aufgabe, nämlich der Umwandlung einer Phe-A-Domäne in eine βPhe-A-Domäne, wurde zuvor Hefedisplay verwendet.30 Diese Bemühungen erfordern jedoch einen spezialisierten Hochdurchsatz-Assay, der bezüglich der breiten Anwendbarkeit begrenzt und zeitaufwendig in der Umsetzung ist. In dieser Studie haben wir direkt die Peptidbildung gemessen, um eine Optimierung von Teilreaktionen auf Kosten der Gesamtfunktionalität zu verhindern.29 Screening-Assays, die in der Lage sind, Mutanten im 96-Well-Plattenformat unter nativen Bedingungen zu analysieren, wie der hier verwendete UPLC-MS/MS-Assay, könnten für viele andere NRPS implementiert werden.
Der Anstieg antibiotikaresistenter Bakterien macht die Entdeckung neuer oder modifizierter Antibiotika zu einer vorrangigen Aufgabe. Unser hoch effizienter und breit anwendbarer Screening-Assay kann in diesem Zusammenhang helfen, indem er schnellere und einfachere Engineering-Strategien für NRPS ermöglicht, die eine wichtige Quelle für bioaktive Peptide sind.
Danksagung
Wir möchten uns bei Farzaneh Pourmasoumi, Oliver Waldmann, Katharina Heise und Benno Nuissl für hilfreiche Diskussionen und technische Unterstützung bedanken, bei Heike Heinecke für die NMR-Messungen und bei Felix Trottmann für die HRMS-Messungen. Wir danken der Daimler und Benz Stiftung, dem Fonds der Chemischen Industrie und der International Leibniz Research School (ILRS) für finanzielle Unterstützung. Open Access Veröffentlichung ermöglicht und organisiert durch Projekt DEAL.
Interessenkonflikt
Die Autoren erklären, dass keine Interessenkonflikte vorliegen.
Open Research
Data Availability Statement
Die Koordinaten und Reflektionsdaten der in diesem Manuskript beschriebenen Kristallstruktur sind in der Protein Data Bank (PDB-ID: 8P5O) verfügbar. Weitere primäre Daten sind in den Hintergrundinformationen dargestellt und können auf Anfrage von den Autoren erhalten werden.