Eine Strategie zur Ligandenselektion identifiziert chemische Sonden für die Markierung von SARS-CoV-2-Proteasen
Abstract
Aktivitätsbasierte Sonden sind wertvolle Werkzeuge in der chemischen Biologie. Nach wie vor ist es jedoch eine Herausforderung, molekulare Sonden zu entwickeln, die spezifisch an das aktive Zentrum eines bestimmten Enzyms binden. Wir stellen hier eine Strategie zur Ligandenselektion vor, die es ermöglicht, rasch elektrophile Sonden auf ausgewählte Enzyme zuzuschneiden, und zeigen in einer Machbarkeitsstudie ihre Anwendung für die beiden Cysteinproteasen von SARS-CoV-2. Die resultierenden Sonden markieren spezifisch die aktiven Zentren von 3CLpro und PLpro mit hinreichender Selektivität sowohl in einem lebenden Zellmodell als auch vor dem Hintergrund eines nativen menschlichen Proteoms. Durch die Nutzung der Sonden als Werkzeuge für das kompetitive Screening einer Bibliothek von Naturstoffen wurden Salvianolsäurederivate als vielversprechende 3CLpro-Inhibitoren identifiziert. Unsere Strategie zur Ligandenselektion wird für die schnelle Entwicklung von maßgeschneiderten Sonden von großem Nutzen sein und die Entdeckung von Inhibitoren für eine Vielzahl von Zielproteinen ermöglichen, die auch über Coronavirus-Proteasen hinausgehen.
Einleitung
Das Coronavirus SARS-CoV-2 ist der verursachende Erreger der sich derzeit entwickelnden COVID-19-Pandemie. Innerhalb von weniger als einem Jahr wurden über 60 Millionen Menschen infiziert und die Krankheit hat bereits mehr als 1,4 Millionen Todesfälle verursacht. In Ermangelung eines global abdeckenden Impfprogramms sowie angemessener therapeutischer Möglichkeiten stellt das Virus weiterhin eine große Bedrohung für die öffentliche Gesundheit dar.1 Weltweit wird daher daran gearbeitet, Proteine ins Visier zu nehmen, die für die Invasion und Replikation des Virus im eukaryotischen Wirt essenziell sind. In Homologie zu anderen Coronaviren codiert der erste offene Leserahmen (ORF1ab) des viralen RNA-Genoms viele Proteine, die für die Replikation in der Wirtszelle erforderlich sind, und wird durch einen ribosomalen Frameshifting-Mechanismus abgelesen. Das daraus resultierende Polyprotein wird nach der Expression von der viralen 3C-ähnlichen Protease (3CLpro) und der Papain-ähnlichen Protease (PLpro) prozessiert (Abbildung 1 a).2 Folglich sind die beiden Proteasen für die Replikation von SARS-CoV-2 essenziell, was sie zu einem der Hauptziele für die Entwicklung eines Wirkstoffs gegen das Virus macht.3 Unser Bestreben war es daher, diese Proteasen mit chemischen Sonden gezielt zu untersuchen, um deren Enzymaktivität zu verfolgen und sie zur Entdeckung von Proteaseinhibitoren einsetzen zu können. Aktivitätsbasierte Sonden sind wichtige Werkzeuge sowohl für die Identifizierung von Zielproteinen als auch von Inhibitoren. Darüber hinaus können sie zur Charakterisierung der enzymatischen Reaktivität des aktiven Zentrums in komplexen Proteomen in vitro sowie in lebenden Zellen eingesetzt werden.4 Aufgrund ihrer inhärenten Reaktivität gegenüber Elektrophilen haben Proteasen großes Interesse an der Entwicklung und Anwendung chemischer Sonden geweckt.5 Wir haben kürzlich über Strategien zum kompetitiven Profiling berichtet, welche die Entwicklung maßgeschneiderter Enzyminhibitoren und das Screening synthetischer Bibliotheken ermöglichen sowie für die Aufreinigung von Naturstoffen eingesetzt werden können.6
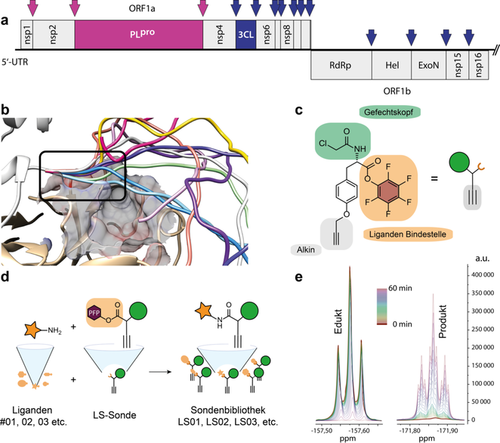
SARS-CoV-2-Proteasen und LS-Sondenstrategie. a) Aufbau des SARS-CoV-2-Polyproteins, translatiert aus ORF1ab mit Spaltstellen der Proteasen 3CLpro und PLpro, welche die nichtstrukturellen Proteine (nsp) aus dem Polyprotein herausschneiden. b) Modellierung des N-Terminus eines 3CLpro-Dimers. c) Molekülstruktur und schematische Darstellung der LS-Sonde mit kovalent bindendem Gefechtskopf, Alkin-Tag und PFP-Ester zur orthogonalen Ligandenmodifikation. d) Schematische Darstellung der parallel durchgeführten Diversifikationsreaktionen der LS-Sonde mit verschiedenen Amin-Liganden. e) Kinetik der Diversifikationsreaktion mit Ligand 09 zur Sonde LS09, Verfolgung durch 19F-NMR in 2-min-Schritten über 60 min.
Aktivitätsbasierte Sonden beinhalten typischerweise einen elektrophilen Gefechtskopf, um kovalent an einen nukleophilen Rest im aktiven Zentrum eines Enzyms zu binden. Die Spezifität einer Sonde, die einzig auf das aktive Zentrum abzielt, wird durch eine gut abgestimmte Kombination aus der Elektrophilie ihres Gefechtskopfes und einer maßgeschneiderten Molekülstruktur erreicht, die den sterischen und elektronischen Anforderungen der Substratbindungstaschen entspricht. Da die meisten Elektrophile auch mit Aminosäureresten in der Peripherie eines Enzyms leicht reagieren, besteht die größte Herausforderung bei der Entwicklung chemischer Sonden darin, diese so anzupassen, dass sie ausschließlich an das aktive Zentrum binden. Wir berichten hier über die Entwicklung einer Ligandenselektionsstrategie für aktivitätsbasiertes Protein-Profiling (LS-ABPP), die eine schnelle Entwicklung von spezifisch bindenden chemischen Sonden ermöglicht. Außerdem präsentieren wir in einer Machbarkeitsstudie deren Anwendbarkeit für die Entwicklung von Sonden und die Identifizierung von Inhibitoren für die beiden Proteasen von SARS-CoV-2.
Ergebnisse und Diskussion
Proteaseaktivität. Sowohl 3CLpro als auch PLpro werden aus dem Polyprotein freigesetzt, indem sie ihre jeweiligen N- und C-terminalen Sequenzen selbst spalten, was bereits für die homologen Sequenzen von SARS-CoV-1 beschrieben wurde.3a, 7
Hierbei ist die Dimerisierung und Reifung von 3CLpro durch N-terminale Selbstspaltung für die vollständige Aktivierung der Protease und die Fähigkeit zur enzymatischen Umsetzung anderer nichtstruktureller Proteine (nsp) erforderlich.8 Aus der Kristallstruktur von reifer, als Dimer vorliegender 3CLpro geht hervor, dass der N-Terminus des einen 3CLpro-Monomers in unmittelbarer Nähe zur Bindetasche des aktiven Zentrums des anderen Monomers liegt und der N-Terminus bereits während der Expression zwischen der kurzen Konsensussequenz Leu-Gln und Ser gespalten wird.9 Molekulare Modellierung der flexiblen Peptidsequenz vor der Spaltung hat gezeigt, dass die ersten Aminosäuren vor der Spaltstelle einen Teil der Substratbindungstasche einnehmen (Abbildung 1 b), was mit mechanistischen Modellen der Protease-Reifung übereinstimmt.8 Die Bedeutung eines gespaltenen N-Terminus für die vollständige enzymatische Aktivität wurde bereits für die 3CLpro von SARS-CoV-1 beschrieben.10 Wir schlussfolgerten daraus, dass Inhibitoren, die auf Proteasen vor ihrer vollständigen Aktivierung abzielen, für die Wirkstoffentwicklung gegen SARS-CoV-2 von großem Wert sein könnten. Daher konstruierten wir eine 3CLpro-Version, die mit einem nicht spaltbaren N-terminalen Strep-Tag II (t3CLpro) fusioniert war. Die Modellierung prognostizierte ein identisches Verhalten des Fusionspeptids am aktiven Zentrum im Vergleich zur nativen Sequenz des Wildtyps vor der Spaltung (Hintergrundinformationen Abbildung S1). Codon-optimierte Sequenzen der 3CLpro- und PLpro-Domänen von nsp3 und nsp5 von SARS-CoV-2 wurden schließlich in einen IPTG-induzierbaren Vektor kloniert und heterolog in Escherichia coli exprimiert.
Die Proteasen wurden entweder durch Affinitätschromatographie aufgereinigt oder in situ in der nativen Zelle des Expressionssystems verwendet. Die aufgereinigte t3CLpro-Version bestätigte unsere Vorhersagen bezüglich der N-terminalen Modifikation und war in einem Protease-Assay unter Verwendung von Oligopeptidsubstraten, die bei enzymatischer Spaltung eine fluorogene 7-Amino-4-methylcumarin-Gruppe freisetzen, tatsächlich inaktiv (Hintergrundinformationen Abbildung S2). Dies bot die einmalige Gelegenheit, die Nützlichkeit der LS-ABPP-Strategie gegen PLpro und den Präaktivierungszustand 3CLpro von SARS-CoV-2 zu validieren und ihre Vielseitigkeit für die Anpassung von chemischen Sonden an verschiedene Zielproteine zu demonstrieren.
LS-ABPP ermöglicht rasches Screening von aktiven Zentren. Wir haben die LS-ABPP-Strategie entwickelt, um die Molekülstruktur einer Sonde in kurzer Zeit an die Bindetasche des aktiven Zentrums eines betreffenden Enzyms anpassen zu können, und demonstrieren das daraus resultierende Potenzial anhand der Entwicklung spezifischer Sonden gegen t3CLpro und PLpro des pandemischen Coronavirus SARS-CoV-2. Eine chemische LS-Sonde wurde basierend auf der Struktur von Tyrosin synthetisiert. Sie beinhaltet einen Chloracetamid-Rest, der mit dem aktiven Zentrum des Proteins reagiert, sowie ein terminales Alkin, über welches das an die Sonde gebundene Enzym mittels bioorthogonaler Klickreaktion über eine 1,3-dipolare Huisgen-Alkin-Azid-Cycloaddition fluoreszent markiert werden kann (Abbildung 1 c). Als zentrales Element des LS-Sondenkonzepts wurde zusätzlich ein Pentafluorphenyl (PFP)-Ester eingeführt, um die schnelle orthogonale Diversifizierung des Molekülgerüsts durch die Reaktion mit einer Bibliothek von primären und sekundären Aminen zu ermöglichen (Abbildung 1 d). Die LS-Sonde wurde mit jeweils einem von 27 Amin-Liganden im Mikroliter-Maßstab umgesetzt (Hintergrundinformationen Tabelle S1). Zeitaufgelöste 19F-NMR-Spektroskopie zeigte bereits nach 60 min eine vollständige Umsetzung, selbst für die am wenigsten reaktiven aromatischen Amine (Abbildung 1 e). Primäre aliphatische Amine reagierten innerhalb von Minuten, was die Nachverfolgung mittels NMR-Spektroskopie unmöglich machte (Hintergrundinformationen Abbildung S3). Obwohl von einer quantitativ ablaufenden Reaktion ausgegangen werden kann, wurden potenzielle Rückstände der nicht umgesetzten Sonde auf Amin-funktionalisiertem Polystyrol-Granulat immobilisiert, um eine unspezifische Markierung der Proteine zu verhindern. Anschließend wurde das Reaktionsgemisch lyophilisiert, der Rückstand in DMSO gelöst und direkt für Proteinmarkierungsexperimente verwendet.
Ligandenselektion verleiht Spezifität für die Markierung des aktiven Zentrums. Um Sonden mit hoher Spezifität für das aktive Zentrum der Proteasen zu identifizieren, konstruierten wir zusätzlich die entsprechenden inaktiven Mutanten Cys145Ala (t3CLpro) und Cys114Ala (PLpro). Anschließend erfolgte ein In-situ-Screening der Sonden in lebenden E.-coli-Zellen gegen t3CLpro und PLpro sowie gegen die entsprechenden inaktiven Mutanten (Abbildung 2 a). Dazu wurden die Zellen eine Stunde lang mit 20 μm der Liganden-modifizierten Sonden (LS01–LS29; Abbildung 2 b) inkubiert; nach darauffolgender Lyse der Zellen wurde Tetramethylrhodamin (TAMRA)-Azid als fluoreszenter Reporter-Tag mittels Klickreaktion an die Sonde gebunden. Die Sondenmarkierung der Enzyme konnte nach anschließender SDS-PAGE durch Fluoreszenzaufnahmen verifiziert werden (Abbildung 2 a). Einige der Sonden lieferten stark markierte Banden von t3CLpro und PLpro, während andere in Bezug auf eine oder beide Proteasen inaktiv waren. Sonden, die eine deutliche Markierungsintensität zeigten, wurden weiter auf ihre Spezifität untersucht, indem die Markierung des Wildtyps (wt) und der jeweiligen Mutante des aktiven Zentrums (m) verglichen wurde (Abbildung 2 c,d; Hintergrundinformationen Abbildung S4). Die Fluoreszenzintensitäten wurden relativ zur DMSO-Kontrolle quantifiziert, und Sonden mit einem Verhältnis von wt/m>2 wurden als spezifisch betrachtet. Die meisten Sonden zeigten keine selektive Markierung des aktiven Zentrums oder waren inaktiv, was die Schwierigkeit unterstreicht, eine aktive, spezifische Bindung von Sonden zu erreichen. Jedoch wurden mehrere Sonden identifiziert, die eine hohe Spezifität ausschließlich für die Markierung des Wildtyps, nicht jedoch der Mutanten t3CLpro und PLpro zeigten (Abbildung 2 d). Verschiedene Liganden-modifizierte LS-Sonden wiesen außerdem unterschiedliche Bindungsaffinität zu den jeweiligen Enzymen auf. Zum Beispiel zeigten die Sonden LS18 und LS20 spezifische Markierung von t3CLpro, jedoch nicht von PLpro, während LS12, LS14, LS17 und LS24 spezifisch an PLpro, jedoch nicht an t3CLpro banden. Somit konnten komplementäre Sonden für die beiden Proteasen identifiziert werden. Um ihre Empfindlichkeit abzuschätzen, wurden Markierungsexperimente mit den spezifischsten Sonden in Abhängigkeit von der Konzentration durchgeführt. Bemerkenswerterweise wurden t3CLpro von LS18 und PLpro von LS17 und LS24 als empfindlichste Sonden bei Konzentrationen von nur 1 μm markiert (Abbildung 2 e). Außerdem waren die überexprimierten t3CLpro und PLpro die einzigen Proteine, die von den Sonden LS18 und LS24 in lebenden E.-coli-Zellen markiert wurden, was deren Selektivität vor dem Hintergrund eines nativen Proteoms unterstreicht (Abbildung 2 f).
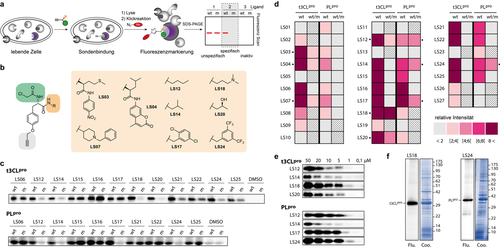
Spezifische und selektive Markierung von SARS-CoV-2-Proteasen. a) Markierung der Zielproteine in lebenden E.-coli-Zellen. t3CLpro und PLpro exprimierende Zellen wurden mit den Sonden behandelt, gefolgt von Zell-Lyse, Klickreaktion mit einem fluoreszenten Reporter-Tag und SDS-PAGE. b) Molekülstrukturen der mit Liganden umgesetzten LS-Sonden mit der höchsten Spezifität. c) Ausschnitt aus dem Screening der Liganden mittels In-situ-Markierung in E. coli des Wildtyps (wt) und der Mutante des aktiven Zentrums (m) bei einer Sondenkonzentration von 20 μm (repräsentative Abbildung aus unabhängigen Triplikaten). d) Quantifizierung der Markierungsintensitäten für die Wildtyp-Proteine (wt), normiert auf DMSO-Kontrollen und Spezifität (wt/m) von LS-Sonden bei 20 μm. Spezifische Sonden zeigen ein hohes Intensitätsverhältnis von Wildtyp zu Mutante (n=3). Kreuzmuster: nicht ermittelt. e) Verdünnungsreihe der spezifischsten Sonden zur Markierung von t3CLpro und PLpro. f) Selektivität der Sonden LS18 und LS24 bei 20 μm zur Markierung der jeweiligen überexprimierten Zielproteasen in situ vor dem Hintergrund eines nativen E.-coli-Proteoms. Flu: Fluoreszenzaufnahme; Coo: Coomassie-gefärbte Gele. Repräsentative Abbildungen aus unabhängigen Triplikaten.
LS18 bindet kovalent an das Cystein im aktiven Zentrum von t3CLpro. Zur beispielhaften Validierung der Identität und Aktivität der Liganden-modifizierten LS-Sonden wurde ein präparativer Syntheseweg zur Sonde LS18 entwickelt (Abbildung 3 a). Ausgehend von Boc-geschütztem l-Tyrosin wurde das terminale Alkin durch Veretherung mit Propargylbromid eingeführt und anschließend über Amidkupplung mittels N,N-Dimethylpropan-1,3-diamin mit dem Amin-Ligand verknüpft. Zuletzt wurde der Chloracetamid-Gefechtskopf eingebracht, wobei LS18 als Chloracetat- beziehungsweise Formiat-Salz erhalten wurde.
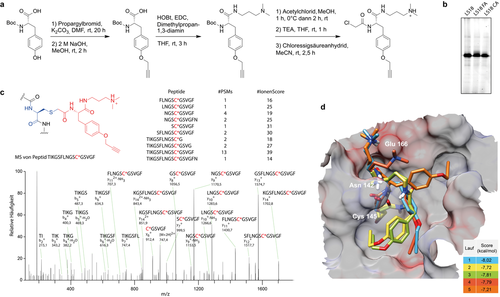
Auf das aktive Zentrum ausgerichteter Mechanismus der Sonde LS18. a) Präparative Direktsynthese von LS18 als Chloracetat- (LS18 CA) und Formiat-Salze (LS18 FA). b) Markierung von t3CLpro in lebenden Zellen von E. coli unter Verwendung von in situ generierter LS18 im Vergleich zu aufgereinigtem synthetischem LS18 CA und LS18 FA bei jeweils 20 μm. c) Proteinsequenzierung von LS18-markiertem t3CLpro durch Massenspektrometrie ergab eine kovalente Modifikation des Cysteins im aktiven Zentrum. Repräsentative Ergebnisse von zwei unabhängigen Replikaten. d) Molekulares Docking von LS18 an das aktive Zentrum der Kristallstruktur von 3CLpro (PDB-ID: 6YB7). Ein Video mit den am besten bewerteten Docking-Posen von LS18 finden Sie in den Hintergrundinformationen.
Im Vergleich zu in situ durch LS-Sonden-Diversifikation generierter LS18 zeigten beide synthetischen Versionen der Sonde identische Aktivität und Selektivität im nativen E.-coli-Proteom (Abbildung 3 b). Um die Wirkungsweise im aktiven Zentrum zu bestätigen und die Stelle der kovalenten Modifikation zu identifizieren, wurde t3CLpro in lebenden E.-coli-Zellen mit Sonde LS18 markiert, gefolgt von Zelllyse und SDS-PAGE ohne vorherige Klickreaktion. Die Coomassie-gefärbte Bande von t3CLpro wurde aus dem Gel ausgeschnitten, mit Pepsin verdaut und mittels Massenspektrometrie sequenziert. Die Überprüfung auf Modifikationen der einzelnen Aminosäuren durch LS18 bestätigte die kovalente Bindung der Sonde an das aktive Zentrum (Abbildung 3 c). Aufbauend auf diesen Ergebnissen wurde ein kovalentes molekulares Docking von LS18 an das aktive Zentrum der Kristallstruktur von t3CLpro von SARS-CoV-2 (PDB-ID: 6YB7) durchgeführt.9a Obwohl die fünf am besten bewerteten Docking-Ergebnisse eine gewisse Streuung aufweisen, fiel die allgemeine Orientierung der Sonde in allen Fällen ähnlich aus (Abbildung 3 d). Das molekulare Docken von LS18 ergab vergleichbare Ergebnisse, sowohl für die native Form von 3CLpro mit verkürztem N-Terminus als auch mit unseren Strukturmodellen des verlängerten nativen N-Terminus vor der Selbstspaltung von 3CLpro und dem nicht spaltbaren Strep-Tag-II-modifizierten N-Terminus (Hintergrundinformationen Abbildung S5). Um einen Einfluss des N-terminalen Strep-Tags II auf die Bindung der Sonde auszuschließen, wurde der Tag über eine Enterokinase-Schnittstelle entfernt, woraus eine 3CLpro-Version resultierte, die im Vergleich zur nativen Form einen lediglich um zwei Aminosäuren verlängerten N-Terminus aufwies. Die erfolgreiche Markierung dieser verkürzten Variante von t3CLpro mit der Sonde LS18 bestätigte, dass dank deren geringer Molekülgröße eine Markierung des aktiven Zentrums der Protease unabhängig von ihrer Aktivierungsstufe möglich ist (Hintergrundinformationen Abbildung S6).
Vergleich von 3CLpro-Homologen. Um zu untersuchen, ob unsere LS-Sondenstrategie auch die Markierung von 3CLpro des eng verwandten SARS-CoV-1, das ebenfalls der Untergattung Sarbecovirus angehört, ermöglicht, wurden auch dessen Wildtyp- und Mutantensequenzen exprimiert und LS-Sondenmarkierungsexperimente in situ durchgeführt. Für die meisten Liganden-modifizierten LS-Sonden waren die Markierungsintensität und -spezifität für das aktive Zentrum beider t3CLpro-Homologe praktisch identisch (Hintergrundinformationen Abbildung S7a), was eine Robustheit der Methode auch über verschiedene Virusstämme hinweg zeigt.
Die Sonden LS06 und LS17 zeigten jedoch eine konzentrationsabhängige Spezifität für t3CLpro von SARS-CoV-1, während sie für SARS-CoV-2 unspezifisch waren (Hintergrundinformationen Abbildung S7b–d). Diese geringfügigen, aber wichtigen Unterschiede verdeutlichen die Anwendbarkeit der LS-Sondenstrategie zur Feinabstimmung chemischer Sonden auf die jeweiligen Eigenschaften des aktiven Zentrums des entsprechenden Enzyms.
Kompetitives Screening identifiziert Enzyminhibitoren. Da unsere Sonden eine empfindliche und für das aktive Zentrum spezifische Markierung von PLpro sowie des Präaktivierungszustands von 3CLpro zeigten, wollten wir ihr Potenzial als chemische Hilfsmittel für das kompetitive Screening von Enzyminhibitoren nutzen. Die Molekülstrukturen von mehr als 1000 kommerziell erhältlichen Lebensmittelzusatzstoffen, Naturstoffen und Proteaseinhibitoren wurden durchsucht, von denen die meisten für eine menschliche Anwendung zugelassen sind. Daraus wurden manuell 44 Verbindungen mit elektrophilen Strukturmotiven wie Aldehyden, Michael-Akzeptoren, Epoxiden und Estern ausgewählt, die potenziell mit dem nukleophilen Cystein des aktiven Zentrums von 3CLpro und PLpro kovalent reagieren könnten, und in Form von DMSO-Stocks in einer Bibliothek zusammengestellt (Hintergrundinformationen Tabelle S2). In einem vorläufigen Screening wurden die aufgereinigten Proteasen mit den einzelnen elektrophilen Verbindungen vorinkubiert und anschließend mit den Sonden markiert (LS18 für t3CLpro und LS24 für PLpro). Wirksame Inhibitoren würden das aktive Zentrum blockieren und dadurch eine nachfolgende Sondenmarkierung verhindern, was als verringerte Fluoreszenzintensität in der SDS-PAGE ausgelesen werden könnte (Abbildung 4 a). Bei einer Anfangskonzentration von 200 μm wurde die Sondenmarkierung von einigen Verbindungen vollständig unterbunden (Abbildung 4 b, Hintergrundinformationen Abbildung S8). Durch anschließende Quantifizierung des Fluoreszenzsignals relativ zur Kontrolle wurden die Verbindungen ausgewählt, die zu einer mindestens 50-prozentigen Hemmung der kompetitiven Sondenmarkierung führten, um ihre Dosis-Wirkungs-Beziehung zu untersuchen (Abbildung 4 c). Die jeweilige mittlere inhibitorische Konzentration (IC50) wurde aus Kurvenanpassungen der Quantifizierung der konzentrationsabhängigen kompetitiven Markierung berechnet (Abbildung 4 d).
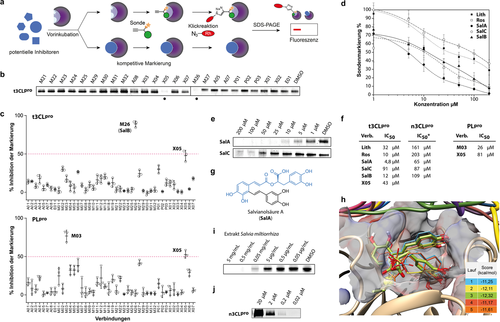
Anwendung chemischer Sonden zur Markierung der Proteasen und zum kompetitiven Profiling. a) Kompetitive Profiling-Strategie, bei der die Proteasen mit potenziellen Inhibitoren vorinkubiert werden, gefolgt von einer kompetitiven Markierung mit einer spezifischen Sonde. Die Fluoreszenz in der SDS-PAGE wird bei erfolgreicher Inhibition des aktiven Zentrums aufgehoben oder verringert. b) Beispiel einer kompetitiven Markierung von 3CLpro mit Verbindungen der zusammengestellten Bibliothek bei einer Konzentration von 200 und 5 μm der Sonde LS18. c) Ergebnisse des vorläufigen kompetitiven Screenings der elektrophilen Verbindungen der Bibliothek bei 200 μm gegen 3CLpro (LS18) und PLpro (LS24) (n=3). d) Inhibitionskurven von 3CLpro-Inhibitoren, bestimmt durch Quantifizierung der Fluoreszenzmarkierungsintensität durch LS18 (n=3). Lith: Lithospermsäure; Ros: Rosmarinsäure; SalA: Salvianolsäure A; SalC: Salvianolsäure C; SalB: Salvianolsäure B. e) Beispiele für konzentrationsabhängige Fluoreszenzintensität im SDS-PAGE mit kompetitiver Markierung von 3CLpro. f) IC50-Werte der aktivsten Inhibitoren, quantifiziert durch kompetitive Fluoreszenzmarkierung. g) Molekülstruktur von Salvianolsäure A (SalA) mit dem für Salvianolsäurederivate typischen Strukturmotiv in Blau. h) Kovalentes Docking von SalA an das aktive Zentrum von t3CLpro (PDB-ID: 6YB7). i) Kompetitive Sondenmarkierung mit Verdünnungsreihen von Wurzelextrakten aus Salvia miltiorrhiza. j) Markierung von reifer n3CLpro (0,2 mg mL−1) durch LS18 in Abhängigkeit von der Sondenkonzentration.
Phenethylisothiocyanat, das von Pflanzen aus der Brassicaceae-Familie aus seinem Vorläufer Gluconasturtiin synthetisiert wird,11 wurde als Inhibitor beider Proteasen identifiziert, was wahrscheinlich auf die unspezifische Thiol-Reaktivität von Isothiocyanaten zurückzuführen ist; jedoch konnte nur eine unvollständige Hemmung festgestellt werden (X05, Hintergrundinformationen Abbildung S9a,b). Hingegen inhibierte Curcumin (M03) die Markierung von PLpro mit einem IC50 von 26 μm fast vollständig und war gegen t3CLpro inaktiv (Hintergrundinformationen Abbildung S9b). Curcumin ist jedoch eine bekannte Pan-Assay-Interferenz-Verbindung (PAIN) und als solche von keinem besonderen Interesse.12 Sowohl X05 als auch M03 hemmten die enzymatische Spaltung eines fluorogenen Peptidsubstrats durch PLpro, was die Validität des kompetitiven Screening-Ansatzes bestätigte (Hintergrundinformationen Abbildung S10). Interessanter war die Aktivität von Salvianolsäure B (M26, SalB), welche die Markierung von t3CLpro mit einem IC50 von 12 μm inhibierte (Abbildung 4 d–f). Wir konzentrierten uns daher auf ähnliche Verbindungen mit entsprechendem Kaffeesäureester-Strukturmotiv. Alle Verbindungen zeigten eine klare Dosis-Wirkungs-Beziehung, wobei Rosmarinsäure (Ros) und Salvianolsäure A (SalA) mit IC50-Werten von 10 bzw. 4,8 μm die höchste inhibitorische Wirkung aufwiesen. Interessanterweise war Salvianolsäure C (SalC), die sich von SalA nur durch eine an einen Benzofuranring gebundene Hydroxygruppe unterscheidet, mit einem IC50 von 91 μm erheblich weniger aktiv (Abbildung 4 e–g). Auch die eng verwandte Lithospermsäure (Lith) ergab lediglich einen IC50 von 32 μm. Diese Ergebnisse zeigen eine fein abgestimmte Struktur-Aktivitäts-Beziehung von Salvianolsäurederivaten für die Inhibition von t3CLpro. Um ein besseres Verständnis der molekularen Abläufe der Proteasehemmung zu erhalten, wurde ein molekulares Docking von SalA an das aktive Zentrum von t3CLpro durchgeführt (Abbildung 4 h). Die besten Docking-Ergebnisse zeigten alle nahezu identische Orientierungen im aktiven Zentrum mit mehreren Wasserstoffbrückenbindungen über die Kresolreste und lieferten erheblich höhere Docking-Werte im Vergleich zu der LS18-Sonde. SalA füllte den größten Teil der Bindungstasche des aktiven Zentrums, und das Docking wurde durch die N-terminale Modifikation nicht behindert, was nahelegt, dass die kompetitive Markierung mit individuell angepassten LS-Sonden die Entdeckung von Inhibitoren ermöglicht, die 3CLpro bereits in der Präaktivierungsphase blockieren können. Da SalA sowie andere potente Salvianolsäurederivate von chinesischem Rotwurzelsalbei (Salvia miltiorrhiza) biosynthetisch hergestellt werden, wurden getrocknete Wurzeln von S. miltiorrhiza extrahiert und in einem kompetitiven Markierungsassay mit der Sonde LS18 gegen t3CLpro getestet, wobei eine starke Hemmung bis zu einer Konzentration von 1 mg mL−1 Extrakt beobachtet wurde (Abbildung 4 i). Um den Einfluss des N-Terminus der Präaktivierungsstufe von 3CLpro auf die Sonden- und Inhibitorenbindung zu untersuchen, führten wir zusätzliche Experimente mit nativer 3CLpro (n3CLpro) durch, die einen gespaltenen N- und C-Terminus aufweist. Die Sonde LS18 markierte n3CLpro mit ausgezeichneter Sensitivität bei Sondenkonzentrationen bis zu 0,2 μm (Abbildung 4 j). Im Gegensatz zu t3CLpro zeigte die vollständig gereifte n3CLpro Aktivität bei der enzymatischen Spaltung fluorogener Substrate (Hintergrundinformationen Abbildung S11). Daraufhin wurden mittels der Substratspaltungsassays die IC50-Werte für die Inhibitoren bestimmt, die für die Präaktivierungsstufe von 3CLpro identifiziert wurden. Alle Salvianolsäurederivate hemmten die enzymatische Aktivität von n3CLpro vollständig, wenn auch bei vergleichsweise hohen Konzentrationen (Abbildung 4 f, Hintergrundinformationen Abbildung S12). Zum Beispiel ergab SalA einen IC50 von 65 μm für die Hemmung von n3CLpro. Umgekehrt reduzierte GC376,13 ein bekannter nanomolarer Inhibitor von reifer n3CLpro, die kompetitive Markierung von t3CLpro vergleichsweise schwach (Hintergrundinformationen Abbildung S13).
Unsere Ergebnisse legen nahe, dass Inhibitoren der Präaktivierungsstufe von 3CLpro etwas andere strukturelle Anforderungen umfassen als Inhibitoren von reifer 3CLpro und in Standard-Screening-Assays möglicherweise nicht erfasst werden.
Protease-Markierung im menschlichen Proteom. Als nächstes wollten wir als mögliche Anwendung der Sonden die Markierung die Proteasen im Hintergrund eines nativen menschlichen Proteoms untersuchen. Wir verwendeten Lysate von menschlichen Hepatozellulären Karzinomzellen (HepG2) und versetzten diese mit verschiedenen Konzentrationen von aufgereinigter t3CLpro und PLpro. Mithilfe der Sonden LS18 für t3CLpro und LS24 für PLpro bei 20 μm und anschließender Klickreaktion konnten beide Proteasen als starke Banden bei Proteasekonzentrationen bis zu 77 μg mL−1 nachgewiesen werden (Hintergrundinformationen Abbildung S14). Schließlich verglichen wir die Markierung von t3CLpro und n3CLpro, wofür absteigende Konzentrationen der Protasen zum Proteom menschlicher Alveolarepithelzellen (A549), die bereits in Infektionsmodellen für SARS-CoV-2 Einsatz fanden, zugegeben wurden.14 Während t3CLpro mit der Sonde LS18 mit vergleichbarer Empfindlichkeit wie im HepG2-Proteom markiert wurde (Abbildung 5 a), konnte n3CLpro sogar bei Konzentrationen von 1 μg mL−1 nachgewiesen werden, wobei der Anteil von n3CLpro im nativen Proteom nur 0,09 % betrug (Abbildung 5 b).
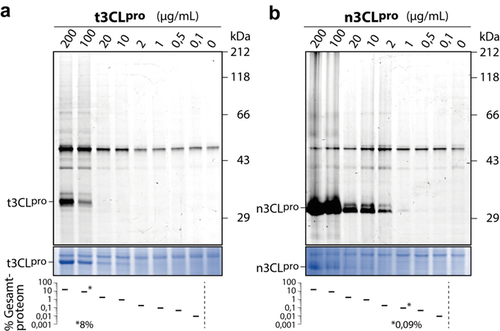
Nachweis von 3CLpro vor dem Hintergrund eines nativen Proteoms (1,14 mg mL−1) von menschlichen A549-Alveolarepithelzell-Lysaten. a) Markierung von t3CLpro und b) n3CLpro durch die Sonde LS18 bei 20 μm Sondenkonzentration. Die Prozentsätze an zugesetzter 3CLpro sind in Bezug auf die Gesamtproteinkonzentration angegeben. Der Absolutwert ist für die entsprechende Nachweisgrenze angegeben.
In eukaryotischen Zelllysaten wurden nur relativ wenige zusätzliche Off-Target-Banden markiert, was darauf hindeutet, dass die beschriebenen Sonden zusätzlich verwendet werden könnten, um SARS-CoV-2-Proteasen vor dem Hintergrund eines komplexen Proteoms zu markieren und ihre Aktivität nachzuweisen. Die Proteasen von SARS-CoV-2 stehen derzeit im Fokus der Forschung zur Wirkstoffentwicklung gegen COVID-19.9b, 15 Obwohl aktivitätsbasierte Sonden wichtige chemische Werkzeuge für die Grundlagenforschung und die Wirkstoffentdeckung darstellen, wurde diesbezüglich bisher überraschend wenig Arbeit an Corona-Viren geleistet. Lediglich eine Sonde auf Peptidsubstratbasis wurde kürzlich für 3CLpro beschrieben.16
Wir haben eine Ligandenselektionsstrategie entwickelt, mit der chemische Sonden in kurzer Zeit auf die spezifische Markierung des aktiven Zentrums eines zu untersuchenden Enzyms zugeschnitten werden können, und ihre Vielseitigkeit bei der Entwicklung aktivitätsbasierter Sonden gegen die Coronavirus-Proteasen 3CLpro und PLpro demonstriert. Während alle Sonden einen identischen elektrophilen Chloracetamid-Gefechtskopf aufwiesen, zeigten nur wenige von ihnen eine hohe Spezifität bei der Markierung des nukleophilen Cysteins im aktiven Zentrum der Enzyme. Diese Ergebnisse zeigen, dass die gewünschte Spezifität von der Auswahl des richtigen Liganden abhängt, und legen nahe, dass ein umfassendes Ligandenscreening der Schlüssel zur Entwicklung optimierter chemischer Sonden ist. Unser Ansatz zur Ligandenselektion ermöglicht ein schnelles Screening der strukturellen Diversität an der Ligandenstelle, ohne dass die zeitaufwändige Synthese größerer Sondenbibliotheken erforderlich ist. Aktivitätsbasierte Sonden sind außerdem nützlich, um Enzyme funktionell zu charakterisieren, und für das Profiling ihrer Aktivität in komplexen Proteomen.17 Wir gehen davon aus, dass unsere 3CLpro- und PLpro-spezifischen Sonden bedeutende Werkzeuge zur weiteren Untersuchung der Aktivitätsfunktion dieser wichtigen Coronavirus-Proteasen darstellen werden. Die Sonden könnten von besonderem Interesse sein, um die Aktivierung von 3CLpro durch Selbstspaltung oder die Deubiquitinierungsaktivität (DUB) von PLpro zu untersuchen, welche die Proteostase der Wirtszelle während des Infektionsprozesses moduliert.18 Darüber hinaus haben wir gezeigt, dass unsere chemischen Sonden nützliche Werkzeuge für die Entdeckung von Inhibitoren gegen 3CLpro und PLpro durch kompetitives Profiling sind. Eine große Menge Arbeit wurde bereits aufgewendet, um Inhibitoren des reifen Stadiums von 3CLpro mithilfe virtueller19 und substratbasierter Screening-Ansätze9b, 15b, 20 sowie rationaler design- und strukturgesteuerter Optimierungsansätze zu finden.15a, 21 Diese jüngsten Entwicklungen profitierten von einer Vielzahl vorangehender Arbeiten zu 3CLpro-Inhibitoren von SARS-CoV-1.3a, 22 Unsere 3CLpro-spezifischen Sonden bieten die einzigartige Gelegenheit, diese Protease vor der N-terminalen Selbstspaltung zu untersuchen und Inhibitoren einer Präaktivierungsstufe des Enzyms zu entdecken, die mit herkömmlichen Substratspaltungsassays nicht erfasst werden können. Wir haben Salvianolsäurederivate als wirksame Inhibitoren von 3CLpro identifiziert, darunter Salvianolsäure A, welche die Sondenmarkierung bei einstelligen mikromolaren Konzentrationen wirksam blockiert. Auch Wurzelextrakte von Salvia miltiorrhiza (chinesischer Rotwurzelsalbei), in dem Salvianolsäuren natürlicherweise vorkommen, zeigten eine starke inhibitorische Wirkung. Extrakte von S. miltiorrhiza und SalA haben bekanntermaßen antithrombotische Wirkungen,23 und Salvianolat-Injektionen (hauptsächlich SalB) sind in China klinisch zugelassen und werden häufig zur Behandlung von koronaren Herzerkrankungen eingesetzt.24 Da Herz-Kreislauf-Erkrankungen einer der größten Risikofaktoren für die von COVID-19 hervorgerufene Morbidität und Mortalität darstellen,25 wären Herz-Kreislauf-Medikamente mit zusätzlicher Aktivität als Corona-Virus-Protease-Inhibitoren von großem Interesse. Unsere Ergebnisse sind daher von unmittelbarer Relevanz für die Forschung an SARS-CoV-2 und die Entwicklung von Wirkstoffen, und unsere neuartige Strategie zur Ligandenselektion ist von allgemeiner Bedeutung für die Entwicklung von Werkzeugen für die Enzymologie und die chemische Proteomik.
Schlussfolgerung
Wir berichten die Entwicklung einer äußerst anpassungsfähigen Strategie zur Ligandenselektion, mit der sich chemische Sondengerüste schnell anpassen lassen, um die Spezifität für das aktive Zentrum einer zu untersuchenden Protease zu erreichen. Als Machbarkeitsstudie untersuchten wir die beiden Cysteinproteasen des Coronavirus SARS-CoV-2, um zu zeigen, dass Selektivität und Effektivität tatsächlich über die Ligandenstruktur gesteuert und feinabgestimmt werden können. Wir konnten Sonden mit einstelliger mikromolarer Aktivität für die Coronavirus-Proteasen 3CLpro und PLpro entwickeln und deren Wert als chemische Werkzeuge für die Markierung der aktiven Zentren dieser Enzyme in komplexen Proteomen sowie für das Screening von Inhibitoren zur Entwicklung eines Wirkstoffs gegen COVID-19 demonstrieren.
Acknowledgements
Diese Forschungsarbeit wurde vom Emmy Noether-Programm (DFG), dem SFB969 und dem EU-RP7 Marie Curie ZIF finanziert. Wir danken Prof. Dr. Andreas Marx und seiner Gruppe für die großzügige Unterstützung. Wir möchten uns auch bei der Proteomics Core Facility der Universität Konstanz für die Unterstützung bei der Proteomanalyse bedanken. Besonderer Dank gilt der Notfallkinderbetreuung an der Universität Konstanz. Open Access Veröffentlichung ermöglicht und organisiert durch Projekt DEAL.
Conflict of interest
Die Autoren erklären, dass keine Interessenkonflikte vorliegen.