Die Bindung von Cyanid an [FeFe]-Hydrogenasen stabilisiert die alternative Konfiguration des Protonentransferpfads
Abstract
Hydrogenasen sind H2-umsetzende Metalloenzyme und enthalten katalytische Kofaktoren, deren Eisenionen durch biologisch ungewöhnliche Kohlenmonoxid- (CO) und Cyanid- (CN−) Liganden koordiniert sind. Externes CO und CN−hemmt Hydrogenasen jedoch. Der molekulare Mechanismus der Bindung von CN− an [FeFe]-Hydrogenasen ist unbekannt. In dieser Studie präsentieren wir Kristallstrukturen der mit CN− behandelten [FeFe]-Hydrogenase CpI aus Clostridium pasteurianum. Auf Grund der hohen Auflösung von 1.39 Å können wir die intrinsischen CO- und CN−-Liganden voneinander unterscheiden. Wir zeigen, dass externes CN− die offene Bindestelle des Kofaktors besetzt, an die auch externes CO bindet. Im Gegensatz zu anderen Inhibitoren zeigen die CN−-behandelten Kristalle Konformationsänderungen konservierter Reste des Protonentransferpfads, die einen direkten Austausch von Protonen zwischen den Aminosäuren E279 und S319 ermöglichen. Diese Konformation wurde als notwendig für einen effizienten Protonentransfer vorgeschlagen, doch wurde sie bisher nicht strukturell nachgewiesen.
Hydrogenasen sind Metalloenzyme, die die reversible Umwandlung von Protonen (H+), Elektronen (e−) und molekularem Wasserstoff (H2) katalysieren. Je nach Metallgehalt und Konfiguration der katalytischen Kofaktoren unterscheidet man [Fe]-, [NiFe]- und [FeFe]-Hydrogenasen. Dabei zeigen [FeFe]-Hydrogenasen die höchste Aktivität für H2-Produktion und H2-Oxidation.1 Der katalytische Kofaktor der [FeFe]-Hydrogenasen, H-cluster genannt, besteht aus einem [4Fe4S]-Subcluster ([4Fe]H) und einer Einheit aus zwei Eisenionen ([2Fe]H). Diese Einheiten sind kovalent durch die Seitenkette eines Cysteins verbunden (Abbildung 1A).2, 3 Relativ zu der [4Fe]H-Einheit werden die Eisenionen der [2Fe]H-Einheit als “proximal” (Fep) und “distal” (Fed) bezeichnet. Fep und Fed werden durch einen Azadithiolat-Liganden verbrückt, dessen Amingruppe ein frustriertes Lewis-Paar mit Fed bildet und auf diese Weise die Aktivierung von H2 ermöglicht.4-6 Um einen effizienten Austausch von Protonen zu gewährleisten, ist das Amin über einen konservierten Protonentransferpfad (PTP) mit der Oberfläche des Proteins verbunden.7-10 Fep und Fed sind jeweils mit einem terminalen Kohlemonoxid- (tCO) und einem terminalen Cyanid-Liganden (tCN−) koordiniert, außerdem binden die Eisenionen gemeinsam einen verbrückenden CO-Liganden, μCO. In dieser Konfiguration zeigt Fed eine offene Bindestelle (Engl. open coordination site, OCS), an welche Substrate binden können. Die OCS ist in der Kristallstruktur des Enzyms im oxidierten Zustand Hox (der auch als “active ready” bezeichnet wird) klar sichtbar (Abbildung 1A).11
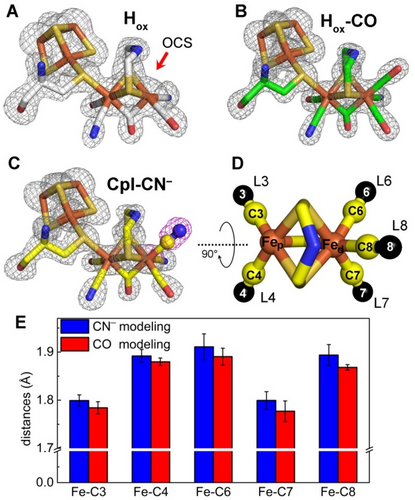
Strukturen des H-Clusters und strukturelle Zuordnung der terminalen CO- und CN−-Liganden. A bis C zeigen H-Cluster in Ketten B von CpI im Hox-Zustand (PDB: 4XDC)11 (A), in mit CO behandelten Hox-Kristallen (Hox-CO-1; PDB: 8ALN) (B) und in CN−-behandelten Hox-Kristallen (CpI-CN−-1; PDB: 8AP2) (C). Simulated Annealing Omit Maps wurden bei 3, 4 und 3.5 σ für A, B bzw. C konturiert. Die H-Cluster weiterer Ketten und Strukturen sind in Abbildung S2 dargestellt. D) zeigt die [2Fe]H-Einheit eines CN−-behandelten Kristalls (PDB: 8AP2), um die in (E) verwendete Nomenklatur zu veranschaulichen. Die terminalen Eisen-Liganden sind mit L3, L4, L6, L7 und L8 abgekürzt, und die Kohlenstoffatome sind entsprechend nummeriert (C3, C4, C6, C7 und C8). E) zeigt die verfeinerten Abstände zwischen jedem Eisenion und den Kohlenstoffatomen seiner terminalen Liganden, wenn sie als ausschließlich CN− (blaue Balken) oder CO (rote Balken) modelliert wurden.
Die Bindung von CO oder CN− an Metalloenzyme ist wohlbekannt. Gleichzeitig findet man diese Moleküle in Hydrogenasen als natürliche Liganden der Eisenionen, wo sie den Low-Spin-Zustand der Eisenzentren bedingen und damit die niedrigen Redoxpotentiale des H-clusters gewährleisten.12 Die Inhibition von [FeFe]-Hydrogenasen durch externes CO ist gut charakterisiert, etwa mittels Röntgenkristallographie,13, 14 die Interaktion zwischen CN− und [FeFe]-Hydrogenasen wurde jedoch bisher kaum untersucht.15
In dieser Arbeit präsentieren wir die röntgenkristallographische Charakterisierung der CN−-behandelten Form der [FeFe]-Hydrogenase CpI aus Clostridium pasteurianum. Zwei Kristalle mit Auflösungen von 1.39 Å und 1.5 Å, im Folgenden als “CpI-CN−-1” und “CpI-CN−-2” bezeichnet, wurden im Detail untersucht und mit zwei Strukturen der CO-behandelten Hydrogenase verglichen, für die höhere Auflösungen als bisher beschrieben erzielt werden konnten (1.34 Å und 1.52 Å; im Folgenden “Hox-CO-1” und “Hox-CO-2” genannt).13, 14 Dies ermöglichte uns einen direkteren Vergleich der vier Strukturen untereinander sowie mit der zuvor veröffentlichten Struktur des CpI-Enzyms im Hox-Zustand (PDB: 4XDC).11, 16 Alle Kristalle wurden entsprechend etablierter Protokolle gewonnen,8, 11 und die CO- und CN−-behandelten Zustände wurden durch Zugabe der entsprechenden Reaktanden in das Kristallisationsreservoir erzielt (Details sind dem methodischen Abschnitt in den Hintergrundinformationen zu entnehmen). Die Behandlung mit CO oder CN− hatte keinen nennenswerten Einfluss auf die Kristallparameter (Tabelle S1). Die Proteine lagen in derselben Raumgruppe P1 21 1 vor, wobei jede der asymmetrischen Einheiten zwei Ketten, A und B, enthielt. Die Strukturen der CO- und CN−-behandelten CpI-Enzyme weisen sowohl untereinander als auch im Vergleich mit CpI im Hox-Zustand eine große Ähnlichkeit auf (die Root Mean Square Deviation der cα-Atome betrug 0.327–0.736 Å, siehe Abbildung S1; Tabellen S1 und S2).
Die vier neuen Strukturen zeigen jeweils eine hohe Elektronendichte an der OCS, die in der Struktur von unbehandelter CpI nicht sichtbar ist (Abbildung 1; Abbildung S2). Die Verfeinerung der Besetzungswahrscheinlichkeiten zeigt, dass die OCS von CN−-behandelter CpI komplett durch einen extrinsischen Liganden besetzt ist, im Falle der CO-behandelten Kristalle aber nur zu 71–93 % einen externen Liganden aufweist (Tabelle S3). Vermutlich ist dies auf einen nicht ausreichend hohen CO-Partialdruck in der Kristallisationskammer zurückzuführen.
Um den Elektronendichten spezifische Moleküle zuordnen zu können, wurden verschiedene Modellierungsstrategien verfolgt. Die Modellierung mit H2O/OH−, welche in früheren CpI-Strukturen als Liganden identifiziert wurden,2, 17 resultierte in deutlichen, unerklärten Elektronendichteresten (Abbildung S3). Dies legt nahe, dass die Elektronendichten in CO- oder CN−-behandelten Kristallen keine H2O/OH−-Moleküle darstellen. Eine deutlich bessere Übereinstimmung wurde bei der Modellierung von CO- oder CN−-Liganden erreicht (Abbildung S3).
CO bindet an die OCS des H-clusters,13, 14 und da CO und CN− isoelektronisch sind, ist die Unterscheidung auf Basis der Elektronendichte herausfordernd. Hochaufgelöste Hydrogenase-Strukturen sowie die Struktur des biomimetischen [2Fe]H-Komplexes zeigen jedoch, dass sich die Abstände zwischen den Eisenionen des aktiven Zentrums und den Kohlenstoffatomen für CO und CN− mit 1.75 Å und 1.9 Å signifikant unterscheiden.17-19 Um CO- und CN−-Liganden in der Struktur von CN−-behandelter CpI zuzuweisen, wurden daher ungerichtete Strukturverfeinerungen vorgenommen und die fünf terminalen Liganden (L3, L4, L6, L7 und L8; Abbildung 1D) dabei entweder als ausschließlich CO oder ausschließlich CN− modelliert (Details sind dem methodischen Abschnitt in den Hintergrundinformationen zu entnehmen). Abbildung 1E zeigt die verfeinerten Abstände zwischen den Eisenionen der [2Fe]H-Einheit und den Kohlenstoffatomen der direkten Liganden (C3, C4, C6, C7 und C8) basierend auf der 1.39 Å Struktur der CN−-behandelten Kristalle CpI-CN−-1. Für jeden Liganden ergab die Modellierung als CN−- oder als CO-Molekül sehr ähnliche Bindungslängen, wobei die Annahme von CO zu etwas kürzeren Abständen führte. Beide Modellierungsansätze ergaben Fe−C3- und Fe−C7-Abstände von durchschnittlich 1.78 Å, während die Abstände zwischen den Eisenionen und C4, C6 und C8 im Durchschnitt 1.9 Å betrugen. Diese Werte entsprechen somit den Fe−CO- und Fe−CN−-Abständen, die in früheren hochaufgelösten Strukturen von Hydrogenasen sowie in der chemisch synthetisierten [2Fe]H-Einheit beobachtet wurden.17-19 Daher sind wir überzeugt davon, die intrinsischen Liganden als CO- (L3 und L7) oder CN−-Liganden (L4 und L6) zuordnen zu können und folgern, dass das an der OCS (L8) gebundene Molekül tatsächlich ein CN−-Ligand ist. Abgesehen von diesem zusätzlichen Liganden liegen die vier natürlichen Liganden in der gleichen Konfiguration vor wie in CpI im Hox-Zustand.17, 20 Die gleiche ungerichtete Verfeinerungsstrategie wurde zur Berechnung der Bindungslängen in der 1.34 Å Struktur der CO-behandelten Kristalle (Hox-CO-1) angewandt. Trotz der hohen Auflösung konnten wir hier keine eindeutigen Ergebnisse erzielen. Wir vermuten, dass dies auf die geringeren Besetzungen der [2Fe]H-Einheit sowie des extrinsischen Liganden zurückzuführen ist (Tabelle S3).
Die CO- und CN−-Liganden der Eisenionen der [2Fe]H-Einheit weisen eine charakteristische Infrarot- (IR) Absorption auf, und ihre Absorptionsfrequenzen sind abhängig von der elektronischen Konfiguration der Eisenionen sowie von der Masse der Liganden.1, 21 Wir führten daher eine IR-spektroskopische Untersuchung der CpI-Kristalle durch (Abbildung S4A). Die unbehandelten Kristalle zeigten das typische Spektrum des Hox-Zustands,16 während die mit CN−-behandelten Kristalle ein neues Spektrum mit einem dritten CN−-Signal bei 2112 cm−1 aufwiesen. Wenn 13CN− anstelle von 12CN− verwendet wurde, verschob sich dieses Signal selektiv um 49 cm−1 zu niedrigeren Energien. Diese Daten bestätigen, dass extrinsisches CN− an den H-cluster bindet. Bemerkenswerterweise verschob sich die Absorption aller Liganden im Vergleich zum Hox-Zustand zu höheren Energien, was auf eine Oxidation der [2Fe]H-Einheit hinweist. Sehr ähnliche Ergebnisse wurden für die [FeFe]-Hydrogenase HydA1 aus Chlamydomonas reinhardtii erzielt (Abbildung S4B). Dies impliziert, dass extrinsisches CN− an den H-cluster unterschiedlicher [FeFe]-Hydrogenasen binden kann. Wir testeten zudem, ob CN− die Aktivität von CpI hemmt. Tatsächlich ging die H2-Produktionsaktivität in Gegenwart von 20 mM KCN um über 64 % zurück (Abbildung S5). In Anbetracht der Strukturanalyse und der IR-Daten legt dies nahe, dass CN− die Katalyse durch Bindung an das OCS hemmt.
Im nächsten Schritt konzentrierten wir uns auf katalytisch wichtige Aminosäure-Seitenketten in den CN−-behandelten Strukturen. Seitenketten in direkter Nähe zum H-cluster zeigten die gleiche Konformationen wie in den Strukturen von Hox und Hox−CO. So sind beispielsweise die zahlreichen H-Brückenbindungen der natürlichen CO- und CN−-Liganden mit dem Proteinrückgrat20 in CpI-CN−-1 und Hox nahezu identisch (Abbildung S6). Demgegenüber beobachteten wir deutliche Abweichungen im PTP (Abbildung 2, Abbildungen S7 und S8). Dieser hochkonservierte Pfad für den katalytischen Protonentransfer wird von C299, E279, S319 und E282 gebildet. Der PTP reicht von der Amingruppe der [2Fe]H-Einheit bis zur Proteinoberfläche und enthält außerdem zwei Wassermoleküle (Wat1 und Wat2), die zwischen C299 und E279 liegen und mit diesen H-Brückenbindungen eingehen (Abbildung 2).7, 8 In jeweils beiden Ketten A und B beider Strukturen CpI-CN−-1 und CpI-CN−-2 sind die Abstände zwischen C299 und der Amingruppe der [2Fe]H-Einheit größer als in Hox (Abbildung 2B, Abbildungen S7 und S8). Die Seitenkette von E279 ist leicht verdreht und damit näher an S319, während der Abstand zwischen E279 und Wat1 in einigen Fällen zunimmt (Abbildung 2B, Abbildungen S7 und S8). Wat1 und Wat2 sind ebenfalls verschoben; in allen Fällen liegt Wat1 um 0.6–1.0 Å näher an der Oberfläche. Folglich ist der Abstand zwischen C299 und Wat1 größer als im Hox-Zustand (Abbildung 2B, Abbildungen S7 und S8). Die Elektronendichten von E282 zeigten unterschiedliche Konformationen. In Kette B von CpI-CN−-2 ist E282 zur Oberfläche hin verdreht und befindet sich außerhalb des H-Bindungsabstands zu S319. In Kette B von CpI-CN−-1 und Kette A von CpI-CN−-2 wurde dieser Rest in zwei Konformationen modelliert (Abbildungen S7 und S8).
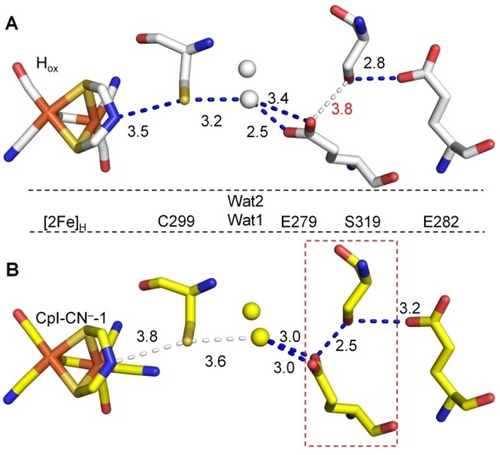
Vergleich der H-Bindungsmuster innerhalb des Protonentransferpfads in Strukturen von CpI im Hox- (A) und im CN−-gebundenen Zustand (B). Die Kohlenstoffatome für CpI-Hox (PDB: 4XDC, Kette A)11 (A) und CpI-CN−-1 (PDB: 8AP2, Kette A) (B) sind weiß bzw. gelb gefärbt. Die Wassermoleküle sind als Kugeln dargestellt und wie die Kohlenstoffatome gefärbt. Die Zahlen zwischen benachbarten Atomen stellen die Abstände in Å dar. Blau gestrichelte Linien repräsentieren Abstände, die für starke bis mittlere Wasserstoffbrücken geeignet sind (<3.5 Å), weiß gestrichelte Linien zeigen schwache H-Bindungen an (Abstände ≥3.6 Å). Der kurze Abstand zwischen E279 und S319 in den mit CN−-behandelten Kristallen wird durch den rot gestrichelten Kasten hervorgehoben.
Die strukturellen Veränderungen im PTP, die wir in den CN−-behandelten Kristallen beobachten konnten, wurden in der Vergangenheit als notwendig für einen effizienten katalytischen Protonentransfer vorgeschlagen, allerding nie strukturell in wildtypischen [FeFe]-Hydrogenasen beobachtet. Im Hox-Zustand wurde der PTP als diskontinuierlich beschrieben, und in der Hox-Kristallstruktur von CpI legt der Abstand zwischen E279 und S319 von 3.6–3.8 Å tatsächlich eine Barriere für effizienten Protonentransfer nahe (Abbildung 2B; Abbildung S8).9, 11, 22 In theoretischen Studien wurde eine Flexibilität der Seitenketten innerhalb des PTPs vorgeschlagen, durch die das Enzym den potenziellen Engpass zwischen E279 und S319 überwindet.23, 24 Wir haben in einer früheren Arbeit den Protonentransfer in der [FeFe]-Hydrogenase HydA1 durch IR-Differenzspektroskopie analysiert. Diese Daten deuteten darauf hin, dass bei der Reduktion des H-clusters vom Hox- in den Hred-Zustand der HydA1-Rest E141 (E279 in CpI) eine stärkere Wechselwirkung mit S189 (S319 in CpI) eingeht, auf Kosten der Wechselwirkung mit Wat1.9 Unerwarteterweise zeigen die CN−-behandelten Strukturen nun Konfigurationen, die diese vorhergesagten Konformationsänderungen unterstützen (Abbildung 2B, Abbildungen S7 und S8).
Soweit uns bekannt ist, wurden Konformationsänderungen von Aminosäure-Seitenketten im PTP noch nie in Strukturen unbehandelter CpI-Wildtypenzyme beobachtet. Sie sind auch nicht in drei unabhängig voneinander erzeugten Strukturen von CO-behandelter CpI13 (Abbildungen S9 und S10), oder der mit Sulfid behandelten [FeFe]-Hydrogenase aus Desulfovibrio desulfuricans vorhanden.25 Im Zusammenhang mit dem Protonentransfer ist es jedoch bemerkenswert, dass das neue H-Bindungsmuster in den CN−-behandelten Kristallen Ähnlichkeiten mit einer Konformation aufweist, die zuvor in unserer CpI-Variante E279D (PDB: 6YF4) (Abbildung S8) beobachtet wurde.8, 10 Diese Enzymvariante hat ein niedrigeres pH-Optimum für die H2-Produktion, behält aber im Vergleich zu wildtypischer CpI eine signifikante Aktivität der H2-Produktion und H2-Oxidation bei.8, 10 Die kürzere Seitenkette des eingeführten D279-Restes befindet sich in H-Bindungsabstand sowohl zu S319 als auch zu Wat1, jedoch deutlich näher an S319. Leichte Verschiebungen von C299 und Wat1 führen zu einer Lücke von 4.0 Å zwischen C299 und Wat1 (Abbildung S8). Vergleicht man die besprochenen Strukturdaten aus früheren Studien nun mit dieser Arbeit, so scheint es, dass sie Momentaufnahmen zweier diskontinuierlicher PTPs darstellen, die sich während der aktiven Katalyse wahrscheinlich abwechseln, um einen kontinuierlichen Weg für den schnellen Protonentransfer zu gewährleisten.
Bislang haben wir keine stichhaltige Erklärung dafür, warum die Bindung von CN− an CpI-Kristalle im Hox-Zustand zu den beobachteten Konformationsänderungen führt. Es scheint möglich, dass die Chemie von CN− (das sich unter den für die Kristallbehandlung angewandten Bedingungen hauptsächlich im protonierten HCN-Zustand befinden sollte) das Protonierungs-Gleichgewicht in einer Weise beeinflusst, die die neue Konformation stabilisiert. Alternativ könnte CN− direkt oder indirekt den Redoxzustand des H-clusters beeinflussen. Unsere IR-Daten deuten in der Tat auf eine stärkere Oxidation der [2Fe]H-Einheit hin. Im Falle des katalytischen Kofaktors von [NiFe]-Hydrogenasen fördert CN− (oder HCN) die Bildung des inaktiven Ni−B-Zustands auf Kosten des Ni-SIr-Zustandes. Dies beinhaltet eine Oxidation von NiII zu NiIII und die Bildung eines negativ geladenen, brückenbildenden Hydroxid-Liganden zwischen den Ni- und Fe-Ionen.26
Die Charakterisierung der Wechselwirkung zwischen [FeFe]-Hydrogenasen und Cyanid verspricht tiefere mechanistische Einblicke in den Katalysemechanismus des H2-Umsatzes und insbesondere des schnellen Protonentransfers, der es [FeFe]-Hydrogenasen ermöglicht, so hohe Effizienzen zu erreichen. Die hier vorgestellten hochauflösenden Strukturdaten bieten eine konkrete Grundlage für die Interpretation von Daten, die mit anderen Methoden gewonnen werden.
Danksagung
J.D. bedankt sich für die Förderung durch die Deutsche Forschungsgemeinschaft (DFG; Projektnummer 461338801). Dieses Projekt wurde von der DFG im Rahmen der deutschen Exzellenzstrategie – EXC 2033–390677874 – RESOLV gefördert. T.H. dankt der DFG (HA 2555/10-1) und der VolkswagenStiftung (Az 98621) für finanzielle Unterstützung. S.T.S. wird von der DFG im Rahmen des Schwerpunktprogramms SPP 1927 (STR1554/5-1) gefördert. Ein Teil dieser Forschung wurde durch das DFG-Graduiertenkolleg GRK 2341 gefördert (A.H., E.H., T.H.). D.J.B. bedankt sich für die Förderung durch das Deutsche Zentrum für Luft- und Raumfahrt (DLR; 50WB2023). Wir bedanken uns bei Shanika Yadav und Ulf-Peter Apfel für die Synthese und Bereitstellung der [2Fe]H-Einheit für die In vitro-Reifung. Die Autorinnen und Autoren danken den Mitarbeiterinnen und Mitarbeitern der Beamlines P13 und P14 (DESY, Hamburg, Deutschland), ID30B (ESRF, Grenoble, Frankreich) und X10SA (SLS, Villigen, Schweiz) für ihre technische Unterstützung bei den Röntgenbeugungsexperimenten. Open Access Veröffentlichung ermöglicht und organisiert durch Projekt DEAL.
Interessenkonflikt
Die Autoren erklären, dass keine Interessenkonflikte vorliegen.
Open Research
Data Availability Statement
Die Daten, die die Ergebnisse dieser Studie unterstützen, sind auf begründete Anfrage beim Autor erhältlich.