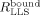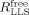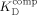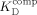Untersuchung schwacher Protein-Ligand-Wechselwirkungen mithilfe langlebiger NMR-Zustände: verbesserter Kontrast für das fragmentbasierte Wirkstoffscreening†
Diese Arbeit wurde von der Swiss National Science Foundation (SNSF), der Swiss Commission for Technology and Innovation (CTI), der EPFL, dem Europäischen Forschungsrat (ERC) und der französischen CNRS gefördert. Diese Arbeit wurde von Dr. Herbert Kogler übersetzt. Wir danken Dr. Nicola Salvi, Harvard Medical School, für hilfreiche Diskussionen.
Abstract
Die vorteilhaften Eigenschaften langlebige Zustände (LLS, “long-lived states”) in der NMR-Spektroskopie ermöglichen ein effizientes Affinitätsscreening von Substanzbibliotheken. Wir beschreiben hier die Anwendung der LLS in kompetitiven Bindungsstudien, um präzise auch schwache Bindungskonstanten von Fragmenten an die ATP-Bindungsstelle der N-terminalen ATPase-Domäne des Hitzeschockproteins 90 (Hsp90), einem therapeutischen Ansatzpunkt in der Krebstherapie, zu bestimmen. Der LLS-Ansatz ermöglicht es, Liganden über einen außergewöhnlich weiten Bereich von Affinitäten aufzuspüren, denn er kann auch bei Ligandenkonzentrationen [L], die einige Größenordnungen kleiner als die Dissoziationskonstanten KD sind, verwendet werden. Diese Eigenschaft macht das LLS-Experiment besonders für die erste Stufe des fragmentbasierten Screenings attraktiv, in dem kleine Moleküle (Fragmente) mit geringer Bindungsneigung identifiziert werden müssen, was mit allen anderen biophysikalischen Methoden nur schwer gelingt.
Im letzten Jahrzehnt hat sich der Ansatz eines fragmentbasierten Screenings als ein Erfolg versprechender Weg erwiesen, um neue Leitstrukturen zu finden.1 Die fragmentbasierte Wirkstoff-Findung (FBDD, “fragment-based drug discovery”) ist zu einer in der pharmazeutischen Industrie akzeptierten Methode gereift, wie die Zahl der Fragmente belegt, die inzwischen zu Leitstrukturen und klinischen Kandidaten entwickelt werden konnten.1a, 2 Diese Vorgehensweise verwendet Bibliotheken sorgfältig ausgewählter, niedermolekularer Verbindungen (MW = 150–250 Da) zur Identifikation von schwach bindenden Liganden an das Zielprotein. Aussichtsreiche Fragmente weisen üblicherweise Dissoziationskonstanten (KD) zwischen 0.1 bis 10 mM auf. Die anschließende Optimierung erfordert in der Regel strukturbiologische Methoden, um den Bindungsmodus zu bestimmen. Für das Fragmentscreening finden Röntgenstrukturanalyse, Oberflächenplasmonenresonanz(SPR)-Spektroskopie, isotherme Kalorimetrie (ITC) sowie biologische Assays bei hoher Konzentration Verwendung. Der Messwert dieser proteinbasierten Methoden ist abhängig vom Anteil des gebundenen Proteins im Vergleich zur Gesamtkonzentration an Target-Protein.3 Wenn die Bindungsaffinität schwach ist, lässt sich dieses Gleichgewicht nur durch hohe Konzentration der Liganden verschieben, eine Anforderung die sich nur schwierig mit Substanzbibliotheken realisieren lässt. Ligandendetektierte Methoden führen zu Signalen, die proportional zum Verhältnis aus gebundenem und freiem Liganden sind, und dieses Verhältnis wäre bei Ligandenkonzentrationen maximal, die der Proteinkonzentration entsprechen. Damit ist ein Screening bei Ligandenkonzentrationen [L] durchführbar, die Größenordnungen unter der Dissoziationskonstanten KD liegen.
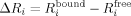 (1)
(1)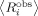 ist der mit den molaren Anteilen gewichtete Mittelwert von
ist der mit den molaren Anteilen gewichtete Mittelwert von 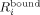 und
und 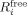 im Fall des schnellen Austauschs,7 wenn also die Austauschraten schneller als der Unterschied der chemischen Verschiebungen sind [Gleichung (2)].
im Fall des schnellen Austauschs,7 wenn also die Austauschraten schneller als der Unterschied der chemischen Verschiebungen sind [Gleichung (2)].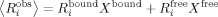 (2)
(2)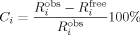 (3)
(3)Kürzlich wurden langlebige Zustände (LLS) für Protein-Ligand-Screening8 vorgeschlagen. Die Differenz ΔRLLS kann deutlich größer werden als die entsprechenden Unterschiede in den Relaxationsraten ΔR1, ΔR2, usw. Damit wird es möglich, guten Kontrast auch bei hohem Ligandenüberschuss zu erzielen, was ein LLS-Screening für den fragmentbasierten Ansatz besonders attraktiv macht, bei dem zunächst nur schwach affine Liganden zu erwarten sind.
LLS sind Zustände von Kernspins, die gegenüber den dipolaren Wechselwirkungen der beiden beteiligten Spins unempfindlich sind.9 Infolgedessen zerfällt ein LLS langsam mit einer Rate RLLS, die oft kleiner als die longitudinale Relaxationsrate R1 ist. Es wurde schon eine Verringerung der Rate um bis zu einem Faktor R1/RLLS=37 gemessen,10 was langlebige Zustände wertvoll für die Beobachtung langsamer Diffusion11 und langsamer Austauschphänomene12 macht und es ermöglicht, die durch DNP induzierte Hyperpolarisation zu erhalten.13 Da die LLS durch ein externes Radiofrequenzfeld aufrechterhalten werden müssen (siehe die Hintergrundinformationen), das exakt in der Mitte der beiden beteiligten Resonanzfrequenzen liegen muss, wird bereits eine kleine, durch die Bindung des Liganden an das Zielprotein verursachte, Änderung der chemischen Verschiebung diesen Effekt aufheben und damit die Relaxationsrate 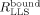 vergrößern und den Kontrast CLLS verstärken. Ein großer Kontrast CLLS in Verbindung mit einer verringerten Relaxationsrate
vergrößern und den Kontrast CLLS verstärken. Ein großer Kontrast CLLS in Verbindung mit einer verringerten Relaxationsrate 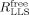
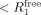 macht das Screening mit LLS besonders vorteilhaft, weil Dissoziationskonstanten KD über einen weiten Bereich beobachtet werden können. Hier konnten wir mittels LLS Dissoziationskonstanten bis 12 mM nachweisen, ein Bereich in dem alle anderen biophysikalischen Methoden, und selbst ligandenbasierte NMR-Methoden, versagen. Bisher machte der Nachweis solcher schwachen Affinitäten die aufwendige Analyse von 15N- und/oder 13C-markierten Proteinen erforderlich.
macht das Screening mit LLS besonders vorteilhaft, weil Dissoziationskonstanten KD über einen weiten Bereich beobachtet werden können. Hier konnten wir mittels LLS Dissoziationskonstanten bis 12 mM nachweisen, ein Bereich in dem alle anderen biophysikalischen Methoden, und selbst ligandenbasierte NMR-Methoden, versagen. Bisher machte der Nachweis solcher schwachen Affinitäten die aufwendige Analyse von 15N- und/oder 13C-markierten Proteinen erforderlich.
Um die Vorteile des LLS-Screenings zu verdeutlichen, wurde der Kontrast CLLS für die LLS-Signale der beiden aromatischen Protonen I und S von Vanillinsäurediethylamid (Ligand II, Abbildung 1; die beiden Kerne sind in Rot markiert) durch Titration gegen die N-terminale ATPase-Domäne des Hitzeschockproteins 90 (Hsp90) bestimmt. Die Anregung der langlebigen Zustände (Details siehe die Hintergrundinformationen) erfolgte mit der Methode von Sarkar et al.10 Drei nichtselektive rf-Impulse 90°−τ−180°−τ−45° wurden zur Anregung der Nullquanten-Kohärenz und des 2IzSz-Terms verwendet. Ein zusätzliches Radiofrequenzfeld (CW, “continuous wave”) mit einer exakt zwischen den beiden skalar gekoppelten Resonanzen8 liegenden Frequenz wird anschließend während einer Wartezeit Δ angelegt, um die beiden Kernspins zur magnetischen Äquivalenz zu zwingen. Solange das Radiofrequenzfeld aktiv ist, tragen die dipolaren Wechselwirkungen zwischen den Spins I und S nicht zur Relaxation des LLS bei, daher ist 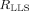
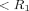 . Abschließend überführt die Abfolge von zwei Pulsen 45°−τ−180°−τ die LLS zurück in beobachtbare Iy- und Sy-Terme.
. Abschließend überführt die Abfolge von zwei Pulsen 45°−τ−180°−τ die LLS zurück in beobachtbare Iy- und Sy-Terme.
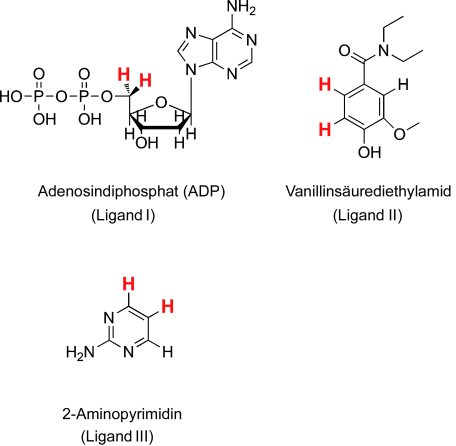
Schwach bindende Liganden an die ATP-Bindungstasche des Hitzeschockproteins 90 (Hsp90). Die für die Anregung von LLS geeigneten Paare von Protonen sind mit roten Buchstaben gekennzeichnet.
In Tabelle 1 sind die molaren Anteile Xbound an gebundenem Ligand für verschiedene Ligand/Protein-Verhältnisse und der zugehörige Kontrast CLLS aufgelistet. Selbst für einen großen Ligandüberschuss [L]tot/[P]tot=272 wird ein starker Kontrast von 45 % beobachtet.
|
[L]tot/[P]tot |
Xbound [Mol-%] |
Kontrast CLLS [%] |
|---|---|---|
|
56 |
0.74 |
72 |
|
125 |
0.49 |
63 |
|
202 |
0.36 |
54 |
|
272 |
0.28 |
45 |
|
366 |
0.22 |
41 |
|
548 |
0.16 |
29 |
|
707 |
0.13 |
23 |
Ein Kontrast CLLS=23 %, der einem Verhältnis von Robs/Rfree=1.3 entspricht, kann noch bei einem Ligandenüberschuss von [L]tot/[P]tot=707 beobachtet werden, also bei Bedingungen, unter denen weniger als 0.2 % des vorhandenen Liganden an das Protein gebunden ist. Im Vergleich mit anderen NMR-Methoden, die alle einen geringeren Kontrast aufweisen, ermöglicht dies den Nachweis von Ligandbindungen bei geringerer Proteinkonzentration und/oder geringeren Bindungsaffinitäten. Für die Entwicklung eines Screening-Assays lassen sich so die geeigneten Konzentrationen für Protein und Liganden über einen größeren Bereich anpassen. Um beispielsweise Liganden mit einem KD≤1 mM und [L]=500 μM noch nachweisen zu können, bedarf es einer Proteinkonzentration [P]=3 μM. Alternativ würden bei [P]=20 mM noch Bindungskonstanten KD>10 mM nachweisbar. Derart geringe Affinitäten sind ein Kennzeichen von Fragmenten, die an Protein-Protein-Wechselwirkungsflächen binden. Auch dieses bietet einen Vorteil gegenüber dem Fragmentscreening mit herkömmlichen ligandenbasierten Methoden.
Langlebige Zustände (LLS) können am besten in isolierten Zweispinsystemen angeregt werden, obwohl auch größere Spinsysteme LLS bilden können.14 Viele kleine Fragmente enthalten geeignete Paare von 1H- oder 19F-Kernen. LLS-Screening wird, wie von Dalvit et al.15 für die klassischen R1- und R2-Experimente vorgeschlagen, am besten in einem Konkurrenzexperiment durchgeführt: ein besser bindender Ligand verdrängt einen schwach bindenden Reporterliganden teilweise aus der Bindungsstelle, sodass eine Verringerung der Relaxationsrate RLLS des freigesetzten Reporterliganden beobachtet wird. So wird es möglich, die Affinität eines stärker bindenden Liganden aus der Änderung der RLLS des Reporterliganden zu berechnen. Da man die Konzentration des Reporterliganden gering halten kann, ist es möglich, auch von wenig löslichen konkurrierenden Liganden die Dissoziationskonstante KD zu bestimmen. Das ist ein entscheidender Vorteil beim Screening von Mischungen und auch um die Bindungskonstanten schwach bindender Fragmente zu bestimmen.
Vor dem Screening einer ganzen Bibliothek von Fragmenten wird üblicherweise eine kleine Auswahl von Verbindungen mit unterschiedlichen NMR-Methoden untersucht, um den späteren NMR-Assay auf das Zielprotein hin zu optimieren. Bereits zu diesem Zeitpunkt kann eine kleine Zahl von Hits identifiziert werden, aus denen dann ein geeigneter Reporterligand für die Konkurrenzexperimente ausgewählt wird. Zur Demonstration der Anwendbarkeit unserer Methode wurden die Relaxationsraten RLLS von drei bekannten Liganden an die ATP-Bindungsstelle der N-terminalen ATPase-Domäne von Hsp90 bestimmt.16 Adenosindiphosphat (ADP, Ligand I) ist das Produkt der ATPase-Reaktion. Vanillinsäurediethylamid (Ligand II) und 2-Aminopyrimidin (Ligand III) wurden als schwach bindende Liganden in einem Fragmentscreening identifiziert.16
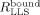 zu messen, wurden Titrationsreihen mit 0.3 mM<[L]tot<6 mM in Gegenwart von [P]tot=10 μM Hsp90 aufgenommen. Die Daten wurden anschließend an die folgende Funktion17 angepasst, um KD und
zu messen, wurden Titrationsreihen mit 0.3 mM<[L]tot<6 mM in Gegenwart von [P]tot=10 μM Hsp90 aufgenommen. Die Daten wurden anschließend an die folgende Funktion17 angepasst, um KD und 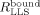 nach Gleichung (4) zu berechnen.
nach Gleichung (4) zu berechnen.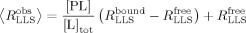 (4)
(4)Dabei stellt [PL]/[L]tot eine Funktion der Dissoziationskonstanten KD dar.17 In Tabelle 2 sind zum Vergleich auch die Dissoziationskonstanten KD aufgelistet, die mit isothermer Kalorimetrie (ITC)16 bestimmt wurden.
|
Ligand |
KD [μM] LLS |
KD [μM] ITC |
|
|
|---|---|---|---|---|
|
I |
15±10 |
10 |
77±6 |
731±7 |
|
II |
708±97 |
790 |
94±3 |
228±11 |
|
III[a] |
– |
>1000 |
– |
– |
- [a] Für Ligand III konnte kein verwertbares LLS-Signal beobachtet werden.
Die mit ITC und LLS bestimmten Gleichgewichtskonstanten sind in guter Übereinstimmung. Um auch Fragmente mit Affinitäten 100 μM<KD<10 mM in Konkurrenzexperimenten nachzuweisen, wurde der Ligand mit der geringsten in der ITC nachweisbaren Affinität (Ligand II) als Reportermolekül verwendet. In Abbildung 2 sind die LLS-Spektren von Ligand II in Abwesenheit (oben) und in Anwesenheit von Hsp90 (mittleres Spektrum) dargestellt. Wird der klinische Kandidat AT 13387 (ASTEX Pharmaceuticals) zugegeben (unterstes Spektrum), ist das LLS-Signal des Reportermoleküls wieder zu erkennen, was beweist, dass beide Verbindungen um dieselbe ATP-Bindungsstelle von Hsp90 konkurrieren.
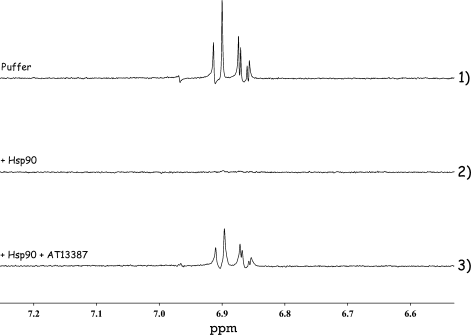
LLS-NMR-Spektren von Ligand II in drei unterschiedlichen Lösungen. Die langlebigen Zustände wurden während Δ=2.5 s aufrechterhalten: 1) 500 μM Ligand II in Abwesenheit von Hsp90; 2) 500 μM Ligand II in Anwesenheit von 10 μM Hsp90; 3) 500 μM Ligand II in Anwesenheit von 10 μM Hsp90 und 10 μM AT13387. In Spektrum (3) wird der Ligand II teilweise aus der ATP-Bindungstasche von Hsp90 freigesetzt und das LLS-Signal wiederhergestellt.
Der effizienteste Weg, um eine Bibliothek von zum Beispiel 1000 Fragmenten gegen ein Zielprotein zu testen, ist die Verwendung von Mischungen aus 3 bis 10 Verbindungen, um die notwendige Proteinmenge und die Dauer der Messungen zu reduzieren. Die Leistungsfähigkeit, LLS in Konkurrenzexperimenten für ein realitätsnahes Screening zu verwenden, wurde mit Mischungen aus bekannten bindenden und nicht bindenden Verbindungen untersucht. Die Verbindungen V, VI und VII (Abbildung 3) waren bereits zuvor als schwache mit ADP konkurrierende Liganden in einem Screening-Assay bei ASTEX gefunden worden.16
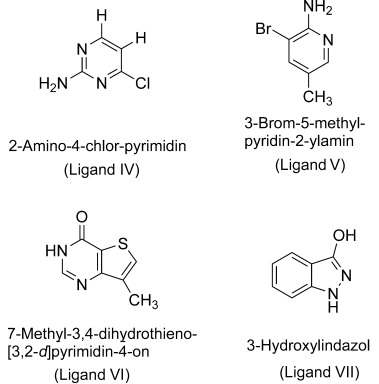
Fragmente, die als Liganden von Hsp90 identifiziert wurden. Die Dissoziationskonstanten KD (Tabelle 3) wurden erstmals mit LLS bestimmt (siehe Text).
In Abwesenheit eines konkurrierenden Liganden führt die Bindung des Reporterliganden an das Protein zu einer schnellen Relaxation der LLS und damit zu einer Abschwächung des Signals (Spektrum 1 in Abbildung 4). Dagegen führt der Zusatz eines konkurrierenden Liganden zu einer teilweisen Freisetzung des Reporterliganden und damit zu einer verlangsamten Relaxation des LLS, was zu einer Signalverstärkung führt (Spektrum 2 in Abbildung 4).
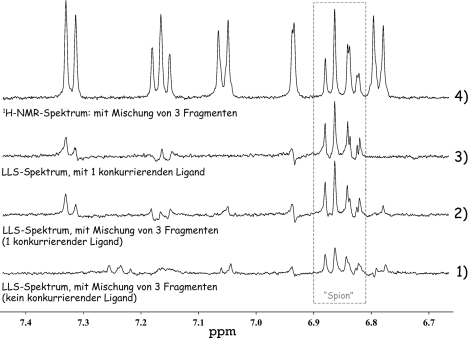
Identifizierung eines schwach binden Liganden in einer Mischung. 1) Schwaches LLS-Signal des Reporterliganden in Abwesenheit eines konkurrierenden Liganden, nachdem der langlebige Zustand während Δ=2.5 s aufrechterhalten wurde (siehe die Hintergrundinformationen). Die Mischung 1 enthält 500 μM des Reporterliganden [II] mit einem KD=790 μM, 2.5 μM Hsp90-Protein und drei nicht bindenden Verbindungen: 600 μM Tyrosin, 600 μM 3,4-Difluorbenzylamin und 600 μM 4-Trifluormethylbenzamidin. (2) Verstärktes LLS-Signal in Anwesenheit eines schwach bindenden Liganden. Die Mischung 2 enthält 600 μM des schwach bindenden Liganden [V] 3-Brom-5-methylpyridin-2-ylamin (KD=2.2 μM) anstelle des nicht bindenden Liganden 3,4-Difluorbenzylamin. 3) LLS-Signal in Anwesenheit eines bindenden Liganden. Die Mischung 3 enthält 500 μM Reporterligand II, 2.5 μM Hsp90-Protein und 600 μM des schwach bindenden Liganden [V] 3-Brom-5-methylpyridin-2-ylamin. 4) Normales 1H-NMR-Spektrum der Mischung 2.
Dieser Effekt beruht auf der relativen Änderung von 13 % der gebundenen Menge an Reporterligand, absolut werden nur 0.3 % des Reporterliganden an das Protein gebunden. Wird eine Mischung von Fragmenten verwendet, muss nach dem Nachweis eines Effekts der einzelne bindende Ligand identifiziert werden (Dekonvolution). In Abbildung 4 ist beispielhaft die Identifizierung von 3-Hydroxyimidazol als schwach bindender Ligand an Hsp90 gezeigt.
Es sei angemerkt, dass für die Verdrängung eines schwach bindenden Reporterliganden durch einen geringer affinen Liganden nur eine vergleichbare Konzentration von Letzterem erforderlich ist. Dies stellt einen erheblichen Vorteil gegenüber anderen NMR-basierten Konkurrenzexperimenten dar, die eine deutlich höhere Konzentrationen für eine nachweisbare Verdrängung erforderlich machen, weil schwach bindende Liganden nur bei einem starken Verhältnis von Ligand zu Protein nachweisbar sind. Im Gegensatz zu anderen NMR-Methoden, bei denen in einem Konkurrenzexperiment von komplexen Mischungen das Signal des Reportermoleküls durch überlagerte Resonanzen verdeckt sein kann,18 ist dieses Risiko in LLS-Experimenten aufgrund der hohen Selektivität bei der Entwicklung des langlebigen Zustands beinahe ausgeschlossen. Wie in Abbildung 4 (Spektrum 2) gezeigt, werden in einem LLS-Spektrum die Signale aller anderen Komponenten im Vergleich mit einem konventionellen 1H-NMR-Spektrum (Spektrum 4) deutlich reduziert. Die Selektivität des LLS-Experiments kann noch weiter verbessert werden, wenn man einen Reporterliganden verwendet, bei dem die beiden verwendeten Resonanzen eine möglichst große Differenz der chemischen Verschiebung aufweisen und damit die Wahrscheinlichkeit einer Überlagerungen mit anderen Paaren gekoppelter Signale verringert wird.
Nachdem ein schwach bindender Ligand identifiziert wurde, kann dessen Dissoziationskonstante KD im Verhältnis zu  des Reportermoleküls durch Titration bestimmt werden, indem man entweder den Reporterliganden in Anwesenheit einer konstanten Konzentration des schwach bindenden Liganden titriert oder umgekehrt.15 Die Titration mit dem Reporterliganden ermöglicht es aber, dieselben experimentellen Bedingungen für verschiedene Fragmente beizubehalten. Die maximale Konzentration des konkurrierenden Liganden wird nur durch dessen Löslichkeit begrenzt. Die Relaxationsrate des langlebigen Zustands RLLS kann bei jeder Konzentration aus den Signalintensitäten bei mindestens zwei unterschiedlichen Wartezeiten τa und τb, während derer der langlebige Zustand aufrecht erhalten wird, bestimmt werden. Um zu überprüfen, dass das hier geschilderte Vorgehen zu selbstkonsistenten Resultaten führt, wurde die Dissoziationskonstanten KD von ADP (Ligand I) bestimmt, indem die Rate RLLS von Vanillinsäurediethylamid (Ligand II) in einem Konzentrationsbereich 500 μM<[LII]<5 mM mit 10 μM Hsp90 und einer konstanten ADP-Konzentration [LI]=15 μM gemessen wurde. Der daraus abgeleitete KD(LI)=8±3 μM ist in Übereinstimmung mit dem KD(LI)=15±10 μM, der durch direkte Titration von ADP mit Hsp90 bestimmt wurde (Tabelle 2). Mit demselben Protokoll wurde die Affinität von 2-Aminopyrimidin (Ligand III) bestimmt (Abbildung 5) in Anwesenheit von 10 μM Hsp90, einer konstanten Konzentration von [LIII]=7 mM und einer Titration im Bereich 500 μM<[LII]<5 mM. Die gemessene Dissoziationskonstante KD (LIII)=11±2 mM legt eine sehr schwache Bindung dieses Liganden an das Protein nahe, die aber spezifisch erfolgen muss, um die Beobachtung zu erklären. Dieses Fragment wurde durch Röntgen-Kristallographie als Ligand an Hsp90 gefunden und in einer Veröffentlichung von Murray et al.16 beschrieben.
des Reportermoleküls durch Titration bestimmt werden, indem man entweder den Reporterliganden in Anwesenheit einer konstanten Konzentration des schwach bindenden Liganden titriert oder umgekehrt.15 Die Titration mit dem Reporterliganden ermöglicht es aber, dieselben experimentellen Bedingungen für verschiedene Fragmente beizubehalten. Die maximale Konzentration des konkurrierenden Liganden wird nur durch dessen Löslichkeit begrenzt. Die Relaxationsrate des langlebigen Zustands RLLS kann bei jeder Konzentration aus den Signalintensitäten bei mindestens zwei unterschiedlichen Wartezeiten τa und τb, während derer der langlebige Zustand aufrecht erhalten wird, bestimmt werden. Um zu überprüfen, dass das hier geschilderte Vorgehen zu selbstkonsistenten Resultaten führt, wurde die Dissoziationskonstanten KD von ADP (Ligand I) bestimmt, indem die Rate RLLS von Vanillinsäurediethylamid (Ligand II) in einem Konzentrationsbereich 500 μM<[LII]<5 mM mit 10 μM Hsp90 und einer konstanten ADP-Konzentration [LI]=15 μM gemessen wurde. Der daraus abgeleitete KD(LI)=8±3 μM ist in Übereinstimmung mit dem KD(LI)=15±10 μM, der durch direkte Titration von ADP mit Hsp90 bestimmt wurde (Tabelle 2). Mit demselben Protokoll wurde die Affinität von 2-Aminopyrimidin (Ligand III) bestimmt (Abbildung 5) in Anwesenheit von 10 μM Hsp90, einer konstanten Konzentration von [LIII]=7 mM und einer Titration im Bereich 500 μM<[LII]<5 mM. Die gemessene Dissoziationskonstante KD (LIII)=11±2 mM legt eine sehr schwache Bindung dieses Liganden an das Protein nahe, die aber spezifisch erfolgen muss, um die Beobachtung zu erklären. Dieses Fragment wurde durch Röntgen-Kristallographie als Ligand an Hsp90 gefunden und in einer Veröffentlichung von Murray et al.16 beschrieben.
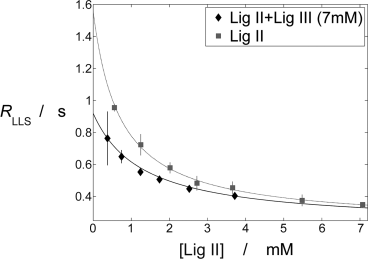
Graue Linie: direkte Titration von Vanillinsäurediethylamid (Ligand II) in Anwesenheit von 10 μM Hsp90. Schwarze Linie: Konkurrenzexperiment mit einer Titration von Vanillinsäurediethylamid (Ligand II) als Reporterligand in Anwesenheit von 10 μM Hsp90 und einer konstanten Konzentration von 7 mM 2-Aminopyrimidin (Ligand III).
Dasselbe Vorgehen wurde auch verwendet, um die Dissoziationskonstanten KD der vier in Abbildung 2 gezeigten Fragmente zu bestimmen. KD-Werte, die aus einer kompletten Titration erhalten wurden, sind in guter Übereinstimmung mit einer Einfachbestimmung bei nur einer einzigen Konzentration (Tabelle 3). Letzteres Vorgehen ist zu bevorzugen, wenn nach dem Screening einer Bibliothek die gefundenen Hits nach ihrer Affinität verglichen werden sollen.
|
Ligand |
|
|
Konzentration [L] der konkurrierenden Liganden [mM] |
|---|---|---|---|
|
I |
0.008±0.003 |
0.010 |
0.015 |
|
II |
(Reporter) |
– |
– |
|
III |
11±2 |
12 |
7 |
|
IV |
0.9±0.2 |
0.7 |
0.9 |
|
V |
1.4±0.9 |
2.2 |
1 |
|
VI |
1.7±0.9 |
2.2 |
1.6 |
|
VII |
7±1 |
5 |
2.9 |
Es sei noch darauf hingewiesen, dass die Wahl des Reporterliganden die experimentellen Bedingungen des LLS-Screenings bestimmt. Mit unserer Wahl eines Reportermoleküls (Ligand II, KD=790 μM) ergibt der konkurrierende Ligand III (KD=12 mM) bei einer Konzentration von 7 mM einen Kontrast von 19 % (erster Punkt der schwarzen Kurve in Abbildung 5). Die Konzentration könnte also auf 3.3 mM reduziert werden, um mit einem dann immer noch ausreichenden Kontrast von 10 % die Bindung eines Liganden in einem Screening-Experiment nachzuweisen, wie das in Abbildung 4 für den Liganden V gezeigt wird (Unterschiede zwischen Spektren 1 und 2). Wenn die erwarteten Affinitäten der Fragmente für ein Ziel in der Größenordnung von 5 mM oder mehr liegen, wäre es günstiger einen Reporterliganden mit einer geringen Affinität zu verwenden, damit man für 10 % Kontrast eine geringere Konzentration der Liganden verwenden kann [siehe Gl. (4)]. Als Konsequenz lässt sich so effizient nach schwach bindenden Liganden suchen, selbst wenn diese nur eine geringe Löslichkeit aufweisen.
Der Nachweis langlebiger Zustände in der NMR-Spektroskopie erfordert Liganden, die ein (bevorzugt isoliertes) System aus zwei gekoppelten Kernspins aufweisen, eine Voraussetzung, die sich nicht generell von allen Bestandteilen einer Mischung für das Fragmentscreening erfüllen lässt. Wir haben daher die LLS-Methode als Konkurrenzexperiment durchgeführt, bei dem sehr effektiv die Freisetzung eines geeigneten Reportermoleküls durch die Änderung seines LLS nachgewiesen werden kann. Wir konnten zeigen, dass in LLS-gefilterten Konkurrenzexperimenten sowohl ein Screening als auch die Bestimmung von Bindungskonstanten von sehr schwach bindenden Fragmenten mit einem KD von bis zu 12 mM möglich ist und dies bei einem bis zu 200-fachen Ligandenüberschuss. Diesen Vorteil kann man nutzen, um die Proteinkonzentration im Assay zu senken und die Empfindlichkeit des Nachweises im Vergleich mit etablierten Screeningmethoden wie STD, LOGSY, T1ρ usw. zu erweitern. Ein hinreichender Kontrast kann noch mit einem bis zu 700-fachen Überschuss des Liganden im Verhältnis zur Proteinkonzentration erreicht werden, was den Nachweis von Fragmenten mit einem KD=790 μM ermöglicht. Die Möglichkeit mit LLS auch sehr geringe Konzentrationen an Protein-Ligand-Komplexen nachzuweisen, lässt sich aber auch dazu nutzen schwache Bindungen (KD>10 mM) zu bestimmen, was eine Voraussetzung für das Fragmentscreening schwieriger Zielproteine ist. Wir konnten außerdem die Dissoziationskonstante des Reporterliganden und verschiedener konkurrierender Liganden quantitativ bestimmen. Die Möglichkeit mittels LLS präzise Bindungskonstanten im mM-Bereich zu bestimmen, in dem ITC und biologische Assays üblicherweise versagen, und dies sogar für wenig lösliche Verbindungen, ermöglicht es, bereits früh Struktur-Wirkungs-Beziehungen aufzustellen und damit Anhaltspunkte für eine chemische Optimierung eines Hits zu liefern.