Darobactine mit Überlegener Antibiotischer Aktivität Generiert durch Strukturgeleitetes Kryo-EM-Biosynthese-Design**
Eine frühere Version dieses Manuskripts ist auf einem Preprint-Server hinterlegt worden (https://doi.org/10.26434/chemrxiv-2022-z96fq).
Abstract
In den letzten Jahrzehnten ist die Entwicklungspipeline neuer Antibiotika gegen Gram-negative Bakterien nahezu zum Erliegen gekommen, da die meisten entdeckten Kandidaten Defizite im Bereich der Wirksamkeit, ihrer pharmakokinetischen Eigenschaften oder eine zu hohe Toxizität aufwiesen. Die Darobactine, eine vielversprechende neue Peptid-Klasse, binden an BamA, ein zuvor von Antibiotika nicht adressiertes Zielprotein der äußeren Membran. Wir haben bereits gezeigt, dass durch Biosynthese-Modifikationen in einem heterologen Wirt neuartige Darobactine mit erhöhter antibakterieller Aktivität erzeugt werden können. Nun verwenden wir eine optimierte Reinigungsmethode und präsentieren Kryo-EM-Strukturen des Bam-Komplexes mit Darobactin 9 (D9), welche als Basis für die biotechnologische Herstellung von zwanzig neuen Darobactinen, einschließlich halogenierter Analoga, dient. Das neu entwickelte Darobactin 22 bindet enger an BamA und übertrifft das bereits hohe Aktivitätsprofil von D9 gegen klinisch relevante Krankheitserreger, wie Carbapenem-resistente Acinetobacter baumannii, um das bis zu 32-fache, ohne erkennbare Anzeichen von Toxizität.
Einleitung
Die stockende Entwicklung neuer Antibiotika in Kombination mit einer wachsenden Weltbevölkerung und einer zunehmenden antimikrobiellen Resistenz (AMR) bei Bakterien, führt zu einer weltweiten Antibiotikakrise.1, 2 Jüngste Studien ergaben, dass 1,27 Millionen Todesfälle im Jahr 2019 allein auf bakterielle AMR zurückzuführen sind, während bakterielle Infektionen mit multiresistenten Keimen zu 5 Millionen Todesfällen bei bereits vorerkrankten Patienten beitrugen.3 Darüber hinaus gingen während der aktuellen Coronavirus-Pandemie (COVID-19) die viralen Infektionen häufig mit einer bakteriellen Koinfektion einher.4, 5 Insbesondere Koinfektionen mit Carbapenemase produzierenden Erregern erhöhen die Sterblichkeitsrate bei Patienten und trugen zum Tod von über 6 Millionen COVID-19-Patienten seit dem Ausbruch der Pandemie im Jahr 2020 bei.6 Die anspruchsvolle Forschung zur Entdeckung und Entwicklung neuer Antibiotikaklassen, insbesondere gegen Gram-negative Erreger, die lebensbedrohliche Infektionen verursachen, führt nur selten zu vielversprechenden Kandidaten.2, 7 Folglich steigt der Bedarf an neuen Medikamenten gegen pathogene Gram-negative Bakterien wie Escherichia coli (E. coli), Acinetobacter baumannii (A. baumannii), Klebsiella pneumoniae (K. pneumoniae) und Pseudomonas aeruginosa (P. aeruginosa) kontinuierlich an.5, 8
Derzeit befinden sich einige Kandidaten zur Behandlung von Infektionen, die durch Gram-negative Erreger verursacht werden, in der präklinischen und klinischen Entwicklungsphase. Dabei handelt es sich jedoch meist um bereits bekannte Wirkstoffklassen oder um neuartige chemische Substanzen, die gängige Zielstrukturen angreifen, wie z.B. das am Ribosom wirkende Breitspektrumantibiotikum Odilorhabdin, welches ursprünglich aus Xenorhabdus nematophila isoliert wurde.9 Die Darobactine, die ebenfalls von einem entomopathogenen Bakterium, Photorhabdus khanii,10 produziert werden, gehören vermutlich zu den vielversprechendsten neuen antibakteriellen Wirkstoffen der letzten 50 Jahre,2 da sie ein neuartiges Breitspektrum-Target in Gram-negativen Krankheitserregern blockieren. Der Wirkmechanismus überwindet gängige Resistenzen von Pathogenen und die Effektivität konnte sowohl in vitro, als auch in vivo nachgewiesen werden.2 Diese Familie bizyklischer Heptapeptide gehört zur biosynthetischen Klasse der ribosomal synthetisierten und posttranslational modifizierten Peptide (RiPPs).10 Darobactine unterscheiden sich von anderen neuen Antibiotikakandidaten, wie dem kürzlich veröffentlichten Macolacin,11 da sie die kommensalen menschlichen Darmbakterien der Familie Bacteriodes nicht abtöten, eine vorteilhafte Eigenschaft im Hinblick auf die beabsichtigte Anwendung.10 Durch die selektive Bindung am β-Faltblatt des äußeren Membranproteins (OMP) BamA stört Darobactin die Integration von OMPs in die Membran, indem es das OMP Signalpeptid nachahmt.12 Infolgedessen ist BamA nicht in der Lage, das “laterale Tor” zu schließen, was sich auf die mechanischen, kinetischen und energetischen Eigenschaften des gesamten Komplexes auswirkt.13, 14 Um den Wert von BamA als lohnendes antibakterielles Ziel hervorzuheben, wurden in vitro Darobactin-resistente E. coli-Mutanten mit mutiertem BamA erzeugt, die eine deutlich verringerte Fitness und Virulenz aufwiesen. Dies deutet auf das Fehlen einer bereits bestehenden Resistenz und ein geringeres Risiko einer schnellen Resistenzentwicklung hin, zumindest in E. coli.12, 15, 16
Darobactin wurde bisher nur in geringen Mengen im nativen Wirt produziert, was für eine größere industrielle Fermentation unzureichend ist.10 Die Totalsynthese über mehrere Schritte mit relativ geringen Ausbeuten wurde erst kürzlich für Darobactin A (DA) entwickelt und erfordert den Einsatz toxischer Chemikalien.17, 18 Interessanterweise bieten kürzlich veröffentlichte heterologe Expressionsplattformen des Darobactin Biosynthese-Genclusters (BGC) ausreichende Produktionsraten in E. coli-Stämmen bei der Nutzung von kostengünstigen Standard-Kulturmedien.10, 16, 19 Das Darobactin BGC besteht aus zwei für die Produktion essentiellen Genen. Das eine kodiert für das Vorläuferpeptid DarA und das andere für das radikalische S-Adenosylmethionin (rSAM)-Enzym DarE, das die bizyklische Verbrückung katalysiert.20 Heterologe Produktionsraten von bis zu 25 mg L−1 für DA und 3 mg L−1 für das deutlich aktivere Darobactin 9 (D9), welches durch Modifizierung des Biosynthesewegs erzeugt wurde, öffnen die Tür für eine effiziente Produktion im großen Maßstab.19 Während die Kryo-EM-Struktur von DA im Komplex mit BamA bereits beschrieben wurde,12 gibt es wenige Daten zu Struktur-Wirkungsbeziehungen (SAR).
In diesem Artikel beschreiben wir eine SAR-Studie, die durch die Strukturaufklärung des BamABCDE (BAM)-D9-Komplexes mittels Kryo-EM initiiert wurde und das Design neuer Darobactin-Kernpeptidsequenzen in silico ermöglichte. Eine neue, schnelle und kostengünstige Strategie zur Modifizierung des Kernpeptids, die auf der “Overlap Extension”-Polymerase Ketten Reaktion (OE-PCR) Methode basiert, erleichterte die Generierung von fünfzehn neuen darA Genvarianten und die Herstellung mehrerer halogenierter Tryptophanderivate durch Fütterungsexperimente, was zu vielversprechenden Derivaten mit verbesserter Bioaktivität im Vergleich zu DA führte, wobei beide an der Zyklisierung beteiligten Tryptophane konserviert blieben.20 Vor allem Darobactin 22 (D22) zeigt deutlich verbesserte minimale Hemmkonzentrationen (MHK) gegen klinisch relevante Krankheitserreger, wie z.B. gegen klinische Isolate von Carbapenem-resistenten A. baumannii (bis zum 32-fachen) und P. aeruginosa (bis zum 4-fachen). Eine Einzelpartikel-Kryo-EM-Strukturaufklärung ermöglichte die Analyse des Bindungsverhaltens des aktivsten neuen Derivats im Vergleich zu den etablierten DA und D9.
Ergebnisse und Diskussion
Fünf native Darobactine (DA bis DE) wurden bereits beschrieben.10, 19, 21 In einer vorherigen Arbeit unserer Gruppe wurden weitere nicht natürliche Derivate (D1 bis D21) hergestellt, darunter ein Derivat, bei dem das endständige Phenylalanin (DA, W1N2W3S4K5S6F7) durch ein Tryptophan (D9, W1N2W3S4K5S6W7) ersetzt wurde (siehe Abbildung 1 a–b). Die signifikant erhöhte antibakterielle Aktivität des modifizierten D9 im Vergleich zum nativem DA war das Resultat eines gentechnischen Eingriffs in den Darobactin-Biosyntheseweg und bildete eine Grundlage für neue gezielte strukturelle Verbesserungen.19 Um festzustellen, ob D9 dieselbe Bindungsstelle wie DA besitzt, haben wir zunächst die Kryo-EM-Struktur des E. coli BAM-Komplexes mit daran gebundem D9 gelöst. Während die durchschnittliche Gesamtauflösung bei 3,4 Å lag, ergab die mit Cryosparc geschätzte, lokale Auflösung um die Bindungsregion von D9 3,0 Å (Abbildung 1 c, d; Abbildung S1 a–e; Tabelle S7). Die Bindung an das β-Faltblatt zeigte eine große Ähnlichkeit mit der selbigen DA Bindung am BAM-Komplex,12 wie in Abbildung 1 d, sowie in der überlappenden Elektronendichte beider Verbindungen (Abbildung S2) gezeigt wird. D9 bindet auch an das β1-Faltblatt des lateralen BamA Tores mit mehreren Wasserstoffbrückenbindungen in antiparalleler β-Faltblattkonformation und ahmt so eine β-Faltblattverlängerung nach, wodurch der funktionelle Bereich von BamA blockiert wird (Abbildung 1 c, d; Abbildung S1 f, g).12 Darüber hinaus sind die N- und C-terminalen Enden von D9 auch über elektrostatische und hydrophobe Wechselwirkungen fest an BamA verankert (Abbildung 1 e, f). Die sich ergebenden Abstände für die Etablierung von Wasserstoffbrückenbindungen von D9 zu Atomen der BamA Bindungsstelle liegen genau innerhalb der erwarteten Werte und lassen vermuten, dass veränderte Kernpeptide zu hochaktiven Darobactin-Derivaten wie den bereits veröffentlichten Darobactinen DB, D4, D9, D11, D14 und D16 führen.19 Bemerkenswert ist, dass D9 näher an BamA gebunden ist, wie ein Vergleich mit nativem DA zeigt (Abbildung 1 d, g, h), was durch zusätzliche Seitenketten- und Rückgratwechselwirkungen von l-Tryptophan an Position 1 mit NH von I430, an Position 6 mit der Carbonylgruppe von N422 und an Position 7 mit der Carbonylgruppe von S425 erklärt werden könnte (Abbildung 1 g, h). Die neuartigen Wasserstoffbrückenbindungsinteraktionen und die damit einhergehenden Änderungen der D9-Orientierung gehen auf Kosten der Wasserstoffbrückenbindungsinteraktion von l-Asparagin an Position 2 mit dem Carbamoylsauerstoff der Seitenkette (Oδ2) von N427 einher. Jedoch bildet die C-terminale Carboxylgruppe des terminalen Tryptophans eine verkürzte Wasserstoffbrückenbindung mit NHδ2 von N422 im Gegensatz zu l-Phenylalanin (Abbildung 1 g, h). Zusätzlich ist die aromatische Gruppe des C-terminalen Tryptophans von D9 deutlich in die hydrophobe Tasche hinein verlagert, die von den Seitenketten F426 und V444 gebildet wird, wodurch möglicherweise starke hydrophobe Wechselwirkungen entstehen (Abbildung 1 e). Infolgedessen könnte dies im Vergleich zu nativem DA die Fähigkeit verbessern das laterale BamA Tor zu verschließen. Zusammengenommen führen die zusätzlichen Interaktionsstellen von D9 zu einer engeren Bindung12 und liefern damit (1) strukturelle Belege und somit die Grundlage der 8-fach höheren Aktivität von D9 gegenüber DA und (2) den konzeptionieren Beweis, dass zusätzliche chemische Veränderungen an DA die Bindung und die biologische Aktivität weiter verbessern könnten.
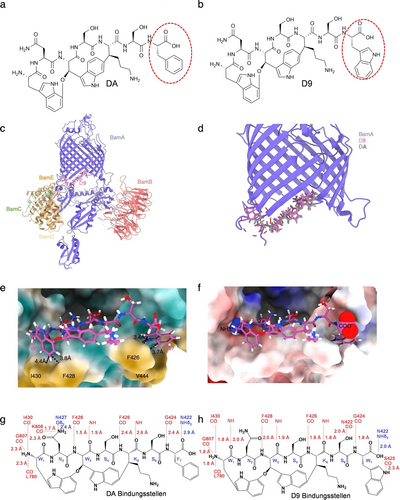
a–b) Strukturformeln von DA und D9. Das Phenylalanin an der DA Position 7 (F7) und das Tryptophan von D9 sind durch einen rot gestrichelten Kreis gekennzeichnet. c) Dreidimensionale Struktur des an BAM gebundenen D9, aufgelöst durch Kryo-EM. d) Vergrößerte Ansicht von D9, welches an der Oberfläche von BamA gebunden ist, verglichen mit DA. e–f) Vergrößerte Ansicht des BAM-D9 Komplexes, welche die lokale (hydrophobe) Umgebung (grün=hydrophil; orange=hydrophob) in der Bindungstasche und das positive (blau) und negative (rot) elektrochemische Potenzial im BAM-D9-Interaktionsbereich zeigt. g–h) 2D-Schema der BamA-Darobactin-Wasserstoffbrückenbindungsinteraktionen. Kanonische β-Strang-Wasserstoffbrücken (rot), Seitenkettenwechselwirkungen (blau) und Abstandsvariationen beim Vergleich von DA12 (g) und D9 (h) wurden kalkuliert.
Für die Entwicklung neuer nicht-nativer Darobactin-Derivate haben wir die derzeit verfügbaren Daten berücksichtigt, einschließlich der relevanten Wechselwirkungen von D9 (WNWSKSW) (Abbildung 1) und DA (WNWSKSF) mit dem BAM-Komplex und der Bioaktivitätsdaten Darobactin-haltiger Extrakte (DA bis DE und D1 bis D21) sowie von reinem DA, DB und D9.10, 19, 21 Um eine ordnungsgemäße Darobactin-Zyklisierung und Ligand-Rezeptor-Interaktion zu gewährleisten (Abbildung 1 g, h), wurden die Positionen 1, 2 und 3 des Darobactins nicht verändert. Insbesondere wurden Manipulationen an der Aminosäureposition 2 vermieden, da diese in der Vergangenheit zu verringerten Produktionsraten und verminderter antibakterieller Aktivität geführt haben.19, 21 Folglich ergaben sich aus dem Bindungsverhalten von DA und D9 am E. coli BAM-Komplex die Positionen 4, 6 und 7 als die flexibelsten Positionen für weitere Gensequenz-Optimierungen, ohne die Zielbindung generell zu beeinträchtigen (Abbildung 1 g, h). Daher haben wir fünfzehn neue Derivate mit Änderungen an diesen Positionen entwickelt (Tabelle 1): Die verstärkte Interaktion des C-terminalen Tryptophans an Position 7 im Vergleich zum kleineren Phenylalanin initiierte die Entwicklung von D26 bis D28, mit terminalem l-Histidin. Die Erwartung war, dass der aromatische Imidazolring des Histidins starke Wechselwirkungen mit den benachbarten Aminosäuren des BamA bilden könnte, ähnlich, wie die aromatischen Ringe des l-Phenylalanins oder l-Tryptophans (Abbildung 1 a, b).

Der Vergleich der drei Derivate mit dem nativen DB (WNWTKRF) und dem zuvor veröffentlichten nicht-natürlichen Derivat D16 (WNWSKAF)19 ermöglichte es uns, die Auswirkungen des Austauschs von l-Serin an Position 6 gegen entweder l-Arginin oder apolarem l-Alanin zu untersuchen. Darüber hinaus führte die erhöhte Aktivität von DB und D4 (WNWTKSF) im Vergleich zu DA zum Austausch von l-Serin gegen l-Threonin an Position 4.19, 21 Die signifikante Aktivitätsverbesserung durch den Ersatz des terminalen l-Phenylalanins durch l-Tryptophan führte zur Entwicklung weiterer D9 Derivate mit unterschiedlichen Aminosäuren an den Positionen 4 und 6. Folglich wurden die Derivate D22 bis D25 und D36 bis D38 mit l-Arginin, l-Histidin, l-Serin, l-Alanin, l-Lysin oder l-Threonin an Position 6 und l-Threonin an Position 4, wie im aktiveren DB vorhanden,21 verändert, um den Einfluss der einzelnen Positionen zu bewerten. Das Ersetzen von l-Serin an Position 4 durch l-Alanin schien weniger Einfluss auf die Aktivität zu haben, wie bei D14 (WNWAKSF) beobachtet wurde,19 was möglicherweise auf das Fehlen einer direkten Seitenketteninteraktion an Position 4 zurückzuführen ist (Abbildung 1 g, h). Um den Einfluss von Position 4 weiter zu untersuchen, wurden D29 bis D32 entworfen, die den verwandten D22 bis D24 und D6 nachempfunden sind.
Der Wechsel der Aminosäure an Position 5 von l-Lysin zu l-Arginin führte zu niedrigeren Produktionsausbeuten und geringerer Aktivität, wie die Extrakt-Aktivität in unserer früheren Publikation und die Analyse von reinem DD (WNWTRSF) durch Böhringer et al. zeigte.19, 21 Diese Daten stehen jedoch im Widerspruch zu dem in unseren Kryo-EM-Experimenten ermittelten Darobactin-Bindungsverhalten. Das Rückgrat von l-Lysin an Position 5 ist nur an der Interaktion mit dem β1-Faltblatt von BamA an F426 mit seiner Carbonyl- und an A427 mit dem Stickstoff der Amidgruppe beteiligt (Abbildung 1 g, h).12 Die l-Lysin-Seitenkette mit ihrer terminalen Aminogruppe ist nicht an der Darobactin-BamA-Interaktion beteiligt, sondern in den äußeren Bereich des β-Faltblattes orientiert (Abbildung 1 d–f; Abbildung S1 f). Kaur et al. fanden heraus, dass die positive Ladung der Lysin-Seitenkette mit den Phosphateinheiten von Cardiolipin und 1-Palmitoyl-2-oleoyl-sn-glycero-3-phosphoglyc-erol interagiert,12 was auch für Arginin gelten sollte. Dementsprechend entwurfen wir D39 mit l-Threonin an Position 4, l-Arginin an Position 5 und 6 und l-Tryptophan an Position 7 (WNWTRRW). Die entworfenen Darobactin-Kernsequenzen, die für D22 bis D32 und D36 bis D39 kodieren (Abbildung S3), wurden durch Einführung von Punktmutationen mittels OE-PCR in pNOSO-darABCDE-9 (DSM 33802) erzeugt. Eine ausführliche Erläuterung ist in den Methoden zu finden (Abbildung S4; Tabelle S2). Die anschließende massenspektrometrische (MS) Analyse (Tabelle S3; Abbildung S5–36) und die Bioaktivitätsevaluation der Rohextrakte zur Priorisierung der aktivsten Derivate wurden, wie in unserer früheren Veröffentlichung beschrieben, durchgeführt, in welcher D9 entdeckt wurde.19 Verschiedene Masse-Ladungszustände, die in der Elektrospray-Ionisation (ESI) MS beobachtet werden können, wurden in kombinierten, extrahierten Ionen Chromatogrammen (EIC) verwendet, um vergleichbare Daten, für die Fläche unter der Kurve (AUC) zu erhalten, da die Einführung basischer Aminosäuren die am häufigsten vorkommenden Ionenspezies von den doppelt geladenen zu den dreifach geladenen Spezies verschoben hat (Tabelle 1). Die Aktivitätsbestimmung zeigte eindeutig eine Verbesserung der antibakteriellen Aktivität aller neu erzeugten Derivate im Vergleich zu DA und eine mindestens vergleichbare Aktivität zu D9, mit Ausnahme der Derivate, die das terminale l-Histidin beinhalten (D26 bis D28). Der Austausch gegen l-Histidin scheint die Wirksamkeit aufzuheben, was auf das leicht positive elektrochemische Potenzial im C-terminalen Bindungsbereich von BamA (Abbildung 1 f) zurückzuführen sein könnte und wiederum die Bindung von Histidin in der hydrophoben Bindungstasche verhindern könnte. Die Aktivität aller anderen getesteten Derivate mit einem terminalen l-Tryptophan ist jedoch vielversprechend. Interessanterweise wiesen die Aktivitätstests auf einen starken Effekt von Änderungen an Position 6 hin, was beim Vergleich der Aktivitätsdaten von D22 mit D25 und D36 erkennbar ist. Dieser Effekt war bei A. baumannii DSM 30008 am stärksten ausgeprägt und bei P. aeruginosa aufgrund der allgemein höheren Potenz weniger offensichtlich. Insgesamt war die SAR aufgrund von Änderungen an Position 6 in E. coli und K. pneumoniae weniger deutlich. Vor allem die SAR von D37 und D38 deutet darauf hin, dass A. baumannii empfindlicher auf die Veränderung der Position 6 reagiert. Dies könnte ein Hinweis auf mehr erregerspezifische Wechselwirkungen oder andere Faktoren sein, die die antibakterielle Wirksamkeit von Darobactinen beeinflussen. Aus dem direkten Vergleich von D22 mit D31 und D32 lässt sich schließen, dass Änderungen an Position 4 von l-Threonin zu l-Alanin oder verlängerten l-Cystein-Resten, wie bereits beschrieben,19 nur geringe Auswirkungen auf die Aktivität haben.
Die NMR-Strukturaufklärung ergab, dass das l-Cystein in diesen Fällen über eine Disulfidbindung mit einem anderen l-Cystein und Milchsäure verbunden ist (Abbildung S76–85, Tabelle S13, S14). Interessanterweise zeigte D39 mit l-Arginin anstelle von l-Lysin als ringbildender Aminosäure eine stärkere antibakterielle Aktivität. Dieser Befund steht im Widerspruch zu der 16-fachen Aktivitätsabnahme beim Vergleich von DA und DD,19, 21 stimmt aber mit den Vorhersagen überein, die auf dem erwarteten Bindungsmodus beruhen (Abbildung 1 d, h), was auf andere Gründe für die Aktivitätsabnahme von DD schließen lässt.21 Unsere Ergebnisse bezüglich der SAR von Rohextrakten sind in Abbildung 2 zusammengefasst. Während die Daten der Rohextrakte mit Vorsicht zu genießen sind,19 stimmten die Ergebnisse der mit gereinigten Darobactin-Derivaten durchgeführten MHK-Tests vollständig mit den Extraktdaten überein (Tabelle 2).
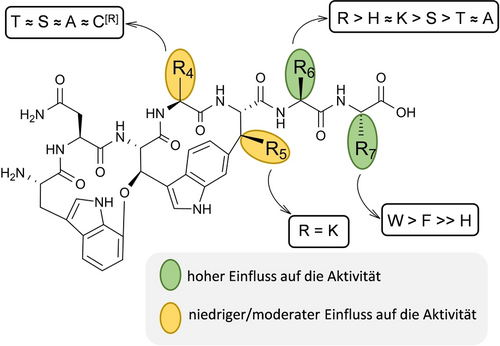
Zusammenfassung der Aminosäuremodifikationen und ihres Einflusses auf die antibakterielle Aktivität. In der SAR Studie wurden die Aminosäure-Positionen 4, 5, 6 und 7 des Heptapeptids verändert. Die getesteten Aminosäuren sind als Ein-Buchstaben-Abkürzungen dargestellt (wobei C[R] das l-Cystein-Addukt bezeichnet) und nach ihrer antibakteriellen Bioaktivität gegen A. baumannii DSM-30008 geordnet, basierend auf einem MHK-Test mit Rohextrakten (Tabelle 1). “≈” vergleichbare Aktivität; “>” oder “≫” höhere Aktivität.
Ein weiterer Ansatz zur Modifizierung von Darobactin ist die Veränderung der Tryptophane, z.B. durch Halogenierung. Die Positionierung des Tryptophan-Indolrings ist wesentlich für die Blockierung des β-Fasses durch Darobactin.12 Wir haben die Möglichkeit in Betracht gezogen, dass die Halogenierung dieser zentralen Aminosäuren die Bioaktivität erheblich verändern könnte. Fünf neue Derivate von D9 wurden durch Fütterung (siehe Hintergrundinformationen) von halogenierten l-Tryptophanen hergestellt, von denen zwei 5-Chlor- und drei 6-Fluor-l-Tryptophan enthielten (Abbildung S37–40). Aufgrund der geringen Ausbeuten konnten jedoch nicht alle Derivate isoliert werden.
Das Derivat mit der höchsten Ausbeute und 6-Fluor-l-Tryptophan an Position 3 des Kernpeptids konnte gereinigt werden, und seine Struktur wurde durch NMR-Spektroskopie bestätigt (Abbildung S86–90; Tabelle S15). Die entsprechende Aktivitätsanalyse der gereinigten, einfach fluorierten Derivate D9–6F1 und D9–6F7 zeigte eine mit dem reinen D9 vergleichbare Aktivität mit leicht verbesserter Aktivität gegen E. coli und K. pneumoniae und leicht verminderter Aktivität gegen P. aeruginosa. (Abbildung S41). Generell erweitern Modifikationen der Darobactin bildenden Aminosäuren, wie z.B. durch Halogenierung, das Spektrum der strukturellen Vielfalt neuer Darobactine und beeinflussen möglicherweise das Bindungsverhalten und die pharmakokinetischen Eigenschaften, wie z. B. für Fluorchinolone bekannt.22 Diese Entdeckungen werden, zusammen mit der Entwicklung eines verbesserten Produktionsverfahren der halogenierten Darobactine, Teil einer separaten Studie sein. Daher haben wir uns in der aktuellen Phase dieses Projekts auf die Reinigung und Charakterisierung neuer nicht halogenierter Derivate mit vielversprechender Rohextraktaktivität konzentriert (Tabelle 1, Abbildung 2).
Um die vielversprechenden Aktivitätsdaten der Rohextrakte zu verifizieren, wurden bestimmte Derivate mit erhöhter Aktivität für die technische Produktion und Isolierung ausgewählt. Bei der Optimierung der Produktion von D22 wurde der handelsübliche E. coli BL21-Gold (DE3) getestet, welcher für eine bessere
Expressionskontrolle entwickelt wurde (Agilent Technologies, Kalifornien, USA). Die Produktion von Darobactin in uninduzierten Kulturen war deutlich reduziert, weshalb der neue Stamm, aufgrund der Autotoxizitätsprobleme von E. coli als besser geeignet eingestuft wurde. Beim Vergleich von E. coli BL21 (DE3) (10,5 (±1,0) mg L−1) und E. coli BL21-Gold (DE3) (9,0 (±1,2) mg L−1) wurde zwar kein signifikanter Unterschied im D22-Produktionstiter festgestellt, wir entschieden uns jedoch dafür im Folgenden E. coli BL21-Gold (DE3) als Wirt zu nutzen. Dies ermöglichte eine kontrolliertere Fermentation und damit eine bessere Reproduzierbarkeit im Gegensatz zu einer signifikant erhöhten unkontrollierten Expression im uninduzierten E. coli BL21 (DE3) Wirt, wie in Groß et al. verwendet.19 Weiterhin wurde ein optimiertes Reinigungsverfahren entwickelt. Es unterscheidet sich insbesondere in der Nutzung des schwachen Kationenaustauscher Harzes Dowex MAC-3 gefolgt von einer Ammoniak-Elution anstelle des zuvor genutzten XAD16-N Harzes.19 Mit diesem modifizierten Arbeitsschritt konnten wir nach einem anfänglichen C18-Flash-Chromatographieschritt den Reinigungsaufwand für die Darobactine auf einen einzigen präparativen C18-Schritt reduzieren, was zu einer Steigerung der D22-Ausbeute von 0,8 mg L−1 Kultur auf 3,0 bis 3,7 mg L−1 (3,8- bis 4,6-fach) führte. Das detaillierte Reinigungsverfahren ist im Anhang zu finden.
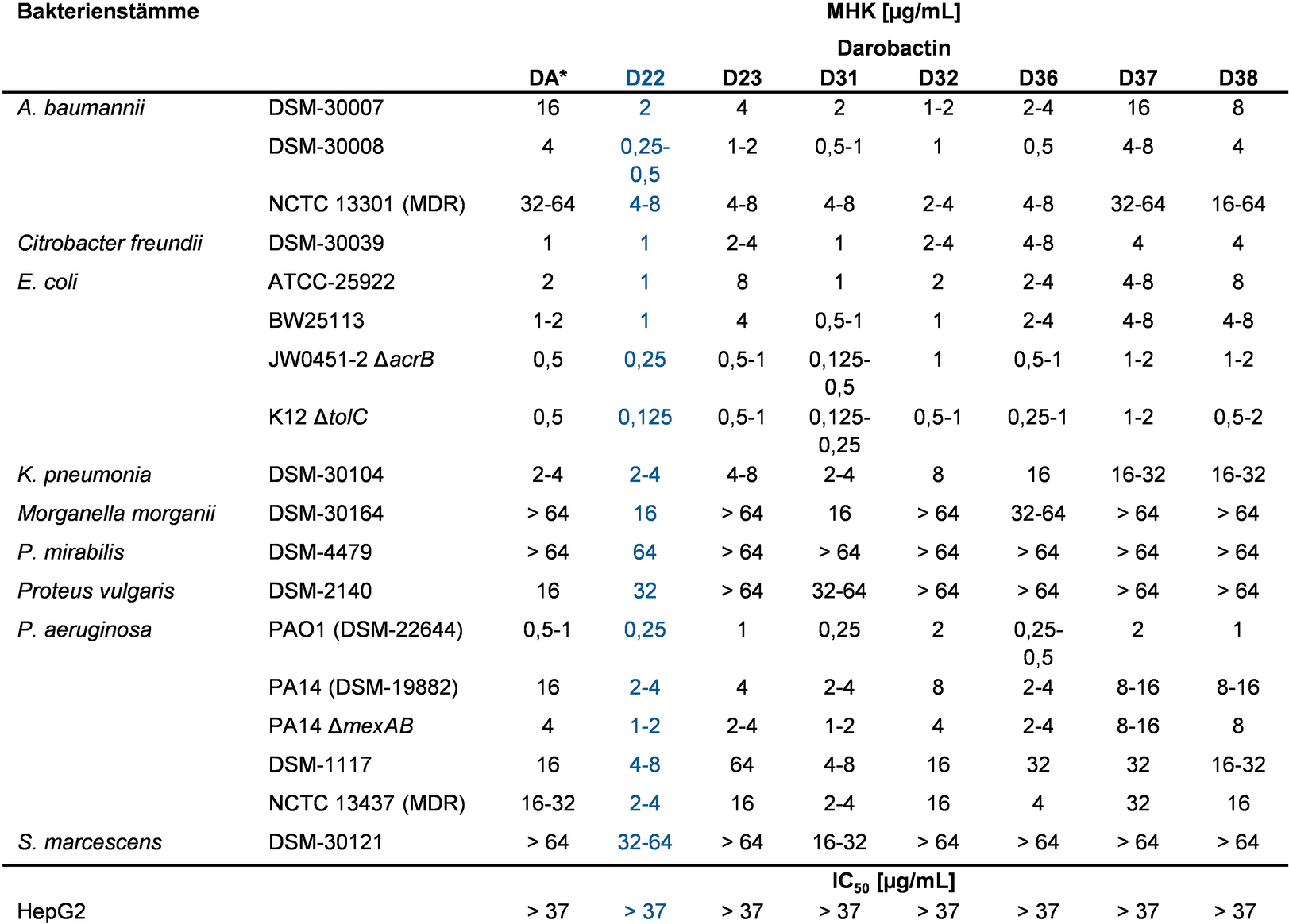
Die gereinigten Darobactine D22, D23, D31, D32, D36, D37 und D38 wurden mittels NMR strukturell verifiziert (Abbildung S46–75, Tabelle S7–12) und gegen eine Reihe verschiedener Gram-negativer Erreger getestet, darunter A. baumannii, E. coli, K. pneumoniae und P. aeruginosa. Die Aktivität wurde mit zuvor veröffentlichten Daten zur DA Aktivität10 und den oben dargestellten Rohextraktergebnissen verglichen (Tabelle 1). Darobactine sind erwartungsgemäß nicht gegen Erreger mit phylogenetisch entfernten BamA Proteinen wie Proteus mirabilis oder Serratia marcescens aktiv (Tabelle 2; Abbildung S42).12 Viele neue Derivate zeigen jedoch eine vielversprechende antibakterielle Aktivität gegen die Mehrzahl der getesteten Erreger (Tabelle 2) und stimmen mit den zuvor ermittelten MHK-Daten der Rohextrakte überein (Tabelle 1). Die Derivate D22 und D31 zeichnen sich durch eine bis zu achtfach höhere Aktivität gegen die getesteten Krankheitserreger im Vergleich zu nativem DA aus. Darüber hinaus wurde eine bis zu zweifach höhere Aktivität im Vergleich zu D9 festgestellt, vor allem gegen Acinetobacter-Stämme.19 Der direkte Vergleich von D22, D31 und D32 unterstreicht zudem den geringen Einfluss von Veränderungen der Aminosäureposition 4 der Kernsequenz. Der wesentliche Einfluss von Änderungen an Position 6 zeigt sich im Vergleich der unterschiedlichen Aktivitäten von D22 und D38 sowie durch den Aktivitätsvergleich von D919 und D37. Der Wechsel von l-Serin zu l-Threonin oder von l-Arginin zu l-Threonin führte zu einer deutlichen Verringerung der bakteriziden Aktivität (Tabelle 2), während eine basische Seitenkette die Aktivität erhöhen kann, wie der Vergleich von DA mit D36 zeigt. Die Aktivität ist generell stark speziesspezifisch, was insbesondere bei den K. pneumoniae-Stämmen zu detektieren ist. Darüber hinaus wurde keine Zytotoxizität gegen die humane Zelllinie HepG2 beobachtet, vergleichbar zu publizierten Daten von DA und D9.19, 10
Zur Untersuchung der SAR von D22, welches das beste Aktivitätsprofil aller bekannten Darobactine aufwies,10, 19, 21 wurde eine Kryo-EM-Ko-Strukturbestimmung des BAM-Komplexes gebunden mit D22 initiiert, um den möglicherweise veränderten Bindungsmodus (MoB) zu untersuchen. Die strukturelle Grundlage der überlegenen Aktivität von D22 wurde durch eine Einzelpartikel-Ko-Strukturaufklärung mittels Kryo-EM des E. coli BAM-D22-Komplexes (Abbildung 3) analysiert und mit der Ko-Struktur von BAM-D9 (Abbildung 1) verglichen. Die Kryo-EM-Struktur des E. coli BAM-Komplexes wurde mit einer Gesamtauflösung von 3,0 Å aufgelöst (Abbildung 3 a; Abbildung S43; Tabelle S6). Der MoB von D22 ist vergleichbar mit der von D9, jedoch mit leichten Konformationsänderungen von N2 und W7, die wahrscheinlich durch die Substitution von T4 bzw. R6 verursacht werden (Abbildung 3 b). Das C-terminale Tryptophan ist tiefer in die hydrophobe Bindungstasche geschoben und bildet im Vergleich zu BamA-D9 eine noch engere Wasserstoffbrücke mit dem Stickstoff des Indolrings und S425 (Abbildung 3 b–e; Abbildung 1 h). N422 von BamA bildet weiterhin mit der C-terminalen Carbonylgruppe von D22 eine NHδ2-Seitenketteninteraktion. Darüber hinaus bildet N422 eine zusätzliche Bindung mit der Guanidingruppe von R6, was wahrscheinlich durch die leichte Verschiebung des C-terminalen Tryptophans in der Bindungstasche begründet ist. Eine Seitenketteninteraktion von Q441 aus dem β-Strang 2 ist im Kontrast zu D9 zu erkennen (Abbildung 3 e). Die beschriebenen Konformationsänderungen sind wahrscheinlich für die erhöhte Bindungsaffinität und folglich die verbesserte Bioaktivität, zumindest gegen A. baumannii Stämme, verantwortlich. Darüber hinaus geht die signifikant veränderte Interaktion und Ausrichtung am C-terminalen Ende nicht auf Kosten einer der drei N-terminalen Wasserstoffbrückenbindungsinteraktionen, wodurch das ordnungsgemäße Schließen des lateralen Tores von BAM aufrechterhalten bleibt (Abbildung 3 c, d). Die Tatsache, dass T4 nicht an der direkten Bindung mit den β-Faltblättern von BamA beteiligt ist, macht diesen Rest attraktiv für den Austausch mit anderen Aminosäuren oder anderer Komponenten zur Verbesserung der pharmakologischen Eigenschaften. Zusammengenommen offenbaren die unterschiedlichen Darobactin-BamA-Bindungsinteraktionen von DA, D9 und D22 wahrscheinlich die strukturelle Grundlage für die verstärkte Aktivität der konstruierten Darobactine, die enger mit BamA gepaart sind, um die β-Faltblattverlängerung zu imitieren und den BAM-Komplex im inaktiven Zustand zu halten, indem das laterale Tor fest verschlossen wird (Abbildung 1, 3).14 Diese struktur- und aktivitätsbasierten Erkenntnisse werden durch die Analyse der unterschiedlichen Bindungskinetiken von DA und D9 im Vergleich zu D22 an E. coli BamA untermauert, die KD-Werte von 0,28 μM, 0,3 μM bzw. 0,16 μM ergaben (Abbildung S44). Dennoch müssen umfangreichere Aktivitätsprofilierungen durchgeführt werden, um eine validere Korrelation zwischen der erhöhten Aktivität von D22 und den strukturellen Veränderungen im Vergleich zu DA und D9 herzustellen.
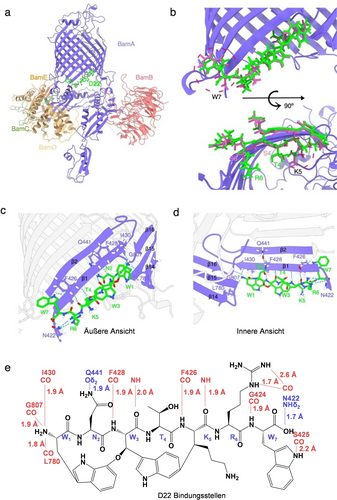
a) Kryo-EM-Struktur des an den BAM-Komplex gebundenen D22. b) Vergrößerte Ansicht von D22 (grün), das an die Oberfläche von BamA gebunden ist, im Vergleich zu D9 (rosa). Die Positionen mit variablen Konformationen sind durch einen rot gestrichelten Kreis gekennzeichnet. Die variablen Aminosäuren an der gleichen Position wurden ebenfalls in verschiedenen Farben angezeigt. c–d) Vergrößerte Ansicht von BamA-D22 von außen und innen. Aminosäuren, die an Wasserstoffbrückenbindungen beteiligt sind, werden als Stäbchen dargestellt und mit einer Buchstabenabkürzung gekennzeichnet. Grüne Striche markieren Wasserstoffbrückenbindungspaare. e) 2D-Schema der BamA-D22 Wasserstoffbrückenbindungsinteraktionen. Kanonische ß-Strang-Wasserstoffbrückenbindungen sind in rot und Seitenkettenwechselwirkungen in blau dargestellt.
Da sich D22 durch seine überlegene Aktivität, insbesondere gegen getestete Acinetobacter-Stämme, auszeichnet (Tabelle 2), wurde dieses Derivat in einem MHK-Assay mit schwer zu behandelnden klinischen Carbapenem-resistenten Acinetobacter baumannii (CRAB) Isolaten getestet, bei denen die meisten der üblicherweise verwendeten Antibiotika nicht mehr wirksam sind (Tabelle S4). Die Aktivitäten von DA, D9 und D22 wurden direkt mit Colistin (COL) verglichen, einem Antibiotikum der letzten Wahl zur Behandlung von durch CRAB verursachten Infektionen, welches Nephrotoxizität verursachen kann.23 Bemerkenswerterweise erwies sich D22 als hochwirksam gegen alle neun getesteten klinischen CRAB-Stämme (MHK von 0,06 bis 0,5 μg mL−1) (Tabelle 3) und zeigte eine deutlich höhere Wirksamkeit als die bislang beste Substanz D9 (und DA), sowie eine nahezu äquivalente antibakterielle Aktivität mit Colistin. Außerdem zeigte D22 eine deutlich bessere Aktivität als andere klinisch verwendete Antibiotika (Tabelle S4).24 Insbesondere war die in vitro Wirksamkeit von D22 gegen CRAB um mindestens eine Größenordnung besser als bei D9. Diese deutliche Verbesserung ist umso beeindruckender, da D9 im Vergleich zu nativem DA bereits eine vierfach verbesserte Aktivität aufweist.

Darüber hinaus wurde die Abtötungskinetik von DA und D22 bei 2-, 4- und 8-facher MHK gegen E. coli MG1655 (Abbildung 4 a, b) und A. baumannii NCTC13301 (Abbildung 4 c, d) über einen Zeitraum von 24 Stunden untersucht. D22 zeigt bei 4-facher und 8-facher MHK eine rasche Abtötung von E. coli MG1655 nach einer frühen statischen Phase von bis zu 2 Stunden, gefolgt von einer bakteriziden Aktivität mit einer Abtötung von mehr als 3 logarithmischen Einheiten (KBE mL−1) innerhalb von 2 bis 4 Stunden. Die beobachtete anfängliche bakteriostatische Wirkung lässt sich möglicherweise durch Daten von Ritzmann et al. erklären, die zeigten, dass die Bindung von Darobactin am lateralen Tor zu einer erhöhten Stabilität durch “Einfrieren” von BamA und folglich zu keiner unmittelbaren bakteriziden Wirkung führt.14 DA und D22 mit 2-, 4- und 8-facher MHK wirkten im Abtötungsexperiment mit E. coli MG1655 in den ersten 8 Stunden bakterizid. Das Ausmaß der bakteriziden Wirkung war konzentrationsabhängig. Dennoch wurde unter den Bedingungen der 2-fachen und 4-fachen MHK nach 24 Stunden ein erneutes Wachstum der Bakterien beobachtet, während dies bei der 8-fachen MHK nicht der Fall war (die für DA und D22 angegebenen MHK-Werte betrugen 2 μg mL−1 bzw. 1 μg mL−1).
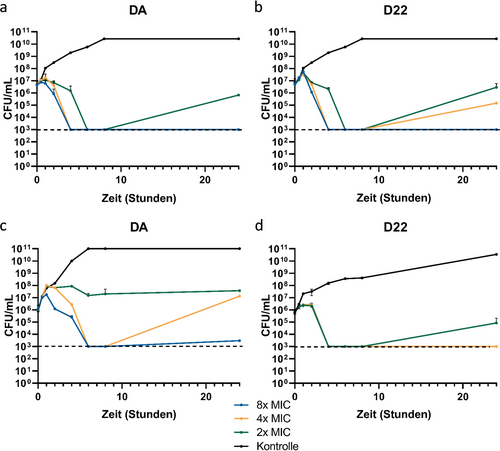
Abtötungskinetiken (time kill curves, TKC) belegen die bakterizide Wirkung von Darobactinen. a–b) TKC von E. coli MG1655 mit DA und D22 bei 2-, 4- und 8-facher MHK. c–d) TKC von A. baumannii NCTC13301 mit DA und D22 bei 2-, 4- und 8-facher MHK. Die Daten sind als Mittelwert±SD angegeben (n=3).
Imai et al. berichteten nicht über ein erneutes Wachstum von E. coli nach der DA-Behandlung, allerdings war der Zeitrahmen ihres Experiments auf 8 Stunden bei 16-facher MHK begrenzt, was einen direkten Vergleich erschwert.10 Sie ermittelten außerdem eine minimale bakterizide Konzentration (MBC) von 8 μg mL−1 gegen E. coli, was mit unseren Ergebnissen für DA und D22 mit MBCs von 16 bzw. 8 μg mL−1 wiederum völlig übereinstimmt. Erfreulicherweise konnten wir bei der Wiederholung der Abtötungskinetik unter hohen Inokulum-Bedingungen (≈5×108 KBE mL−1) zeigen, dass die überlebenden Bakterien sehr wahrscheinlich schlafende “Persister” Zellen sind25 und keine Resistenz entwickelt haben, da repräsentative Kolonien aus diesem Experiment keine MHK-Verschiebungen aufwiesen (Tabelle S5). Bei einem Vergleich der Abtötungskinetik beider Darobactine gegen A. baumannii NCTC13301 zeigt D22 seine klare Überlegenheit gegenüber DA (Abbildung 4 c, d). Für Letzteres stellten wir eine dauerhafte bakteriostatische Wirkung bei 2-facher MHK fest, und ein erneutes Wachstum wurde bei beiden ursprünglich bakteriziden Konzentrationen (4-fache und 8-fache MHK) beobachtet. Im Gegensatz dazu zeigte D22 eine bakterizide Wirkung bei Konzentrationen bis zur 4-fachen MHK und verhinderte das erneute Wachstum 24 Stunden nach der Behandlung vollständig (MBC von 32 μg mL−1).
In Anbetracht der Tatsache, dass A. baumannii leicht Resistenzen erwirbt, was CRAB zu einem Erreger mit höchster Priorität für die Entwicklung neuer Antibiotika macht,8, 26 sind die Ergebnisse von D22 sehr vielversprechend. Es zeigt eine mit Colistin vergleichbare in vitro Aktivität gegen klinische CRAB-Isolate, während Colistin schwere Nebenwirkungen aufweisen kann. D22 zeigte keine toxische Wirkungen bei in vitro Tests mit HepG2-Zellen (Tabelle 2) und bei ersten in vivo Tests mit dem hochsensiblen Zebrafisch-Larvenmodell bis zu 500 μg mL−1 (Abbildung S45), was das Anwendungspotenzial unterstreicht und die weitere Entwicklung hin zu einem potenziellen Antibiotikum zur klinischen Anwendung möglich erscheinen lässt.
Zusammenfassung
Frühere Darobactin-SAR-Studien konzentrierten sich auf native Darobactine21 oder basierten auf deren Analyse.19 Die Aufklärung des MoB von DA und D9 an BamA in Verbindung mit Strukturdaten kürzlich veröffentlichter Darobactine12, 14, 19, 21 ermöglichte einen gezielteren Ansatz, bei dem wir zwanzig neue Derivate konstruierten, darunter zwölf Derivate mit terminalem l-Tryptophan, die eine ähnliche oder höhere Aktivität als DA aufweisen (Abbildung S3). Nach unseren Erkenntnissen scheinen strukturelle Veränderungen an Position 4 nur einen geringen Einfluss zu haben, während die Positionen 6 und 7, die für die Wechselwirkungen mit BamA notwendig sind, einen großen Einfluss auf die Aktivität zu haben scheinen und als potenzieller Ausgangspunkt für die Verbesserung der pharmazeutischen Eigenschaften durch strukturelle Veränderungen genutzt werden könnten. Die bisher vielversprechendste Verbindung, die auf der Grundlage unserer SAR-Studie entwickelt wurde, ist das biosynthetisch hergestellte Darobactin D22, das die antibakterielle Aktivität aller bisher identifizierten nativen Darobactine übertrifft. Dies ist bemerkenswert, da adaptierte Naturstoffe im Laufe der Evolution über Millionen von Jahren entstanden sind, was zu einer strukturellen Vielfalt und Zielspezifität für Pilze, Eukaryoten und Bakterien geführt hat.26, 27 Das gentechnisch hergestellte Derivat D22 ist gegen ein breites Spektrum Gram-negativer bakterieller Krankheitserreger hochwirksam und übertrifft damit sogar den früheren, biosynthetisch hergestellten Spitzenreiter D9, der bereits die Aktivität des nativen DA übertraf.19 Hervorhebend zeigt D22 eine bakterizide Aktivität gegen schwer zu behandelnde CRAB und ist äquipotent zu dem hochwirksamen, jedoch nephrotoxischen Antibiotikum Colistin, welches als eines der letzten Mittel klinisch eingesetzt wird. Darüber hinaus erwies sich D22 in ersten Versuchen in vitro (HepG2) und in vivo (Zebrafischlarven) als nicht toxisch. Weitere Studien werden die optimierte Wirkung von D22 in Bezug auf mechanistische, kinetische und energetische Eigenschaften untersuchen, z.B. in Bezug auf die Linker-Region und die acht β-Faltblätter, verglichen mit DA.14 Künftige Studien sollten auch einen Vergleich mit dem strukturell variablen BAM-Komplex von A. baumannii-Stämmen umfassen, dessen Ko-Struktur mit Darobactinen noch nicht gelöst wurde. Unsere Daten zur Aktivität und Toxizität von D22 sind jedoch ermutigend, um weitere präklinische Studien in Angriff zu nehmen. Die derzeit etablierten Protokolle für die Kultivierung in E. coli eignen sich zwar für die schnelle und valide Erzeugung neuartiger, sogar halogenierter Darobactine in für Forschungs- und erste Entwicklungszwecke geeigneten Mengen,16, 19 doch sind sie aufgrund der geringen Selbstresistenz des Gram-negativen heterologen Wirts E. coli kaum in der Lage, die Produktion für künftige Studien sicherzustellen. Ein Wechsel des heterologen Produzenten zu einem Gram-positiven Stamm wie Bacillus subtilis, der erfolgreich in industriellen Fermentationen eingesetzt wird, könnte höhere Darobactin-Titer ermöglichen. Alternativ könnten auch die kürzlich veröffentlichten Totalsynthesen für DA anwendbar sein, die jedoch derzeit ebenfalls unter Ausbeuteproblemen leiden.18, 17
Zusammenfassend lässt sich sagen, dass unser SAR-Ansatz strukturelles Wissen über die MoB der Darobactine lieferte und zu neuen Derivaten mit einer bis zu 32-fach höheren Aktivität gegen CRAB führte, verglichen mit dem potentesten bekannten Derivat D9.19 Das neue Derivat D22 erwies sich in seiner Aktivität gegen bestimmte Gram-negative Krankheitserreger als vergleichbar mit klinisch verwendeten Antibiotika. Folglich führte unser Ansatz zu einem neuen Derivat der Darobactine, welches eine erhöhte antibakterielle Aktivität aufweist. D22 ist besonders aktiv gegen kritisch priorisierte Gram-negative Erreger, was zu weiteren präklinischen Studien mit diesem vielversprechenden Molekül ermutigt, um hoffentlich eventuell einen Wirksamkeitsnachweis beim Menschen für diese spannende und vielversprechende Molekülklasse zu erbringen.
Danksagung
Wir danken Alexandra Amann und Viktoria George für die Durchführung der MHK-Tests und Dr. F. P. Jake Haeckl, Dr. Daniel Krug, Dr. Peter Sullivan und Dr. Jordan Espenshade für hilfreiche Korrekturen. Die Kryo-EM-Optimierungen und die Datenerfassung wurden in der Kryo-EM-Anlage am CSSB Hamburg durchgeführt (mit Unterstützung der Universität Hamburg, des Universitätsklinikums Hamburg-Eppendorf und der DFG-Förderkennzeichen INST152/772-1, 152/774-1, 152/775-1, 152/776-1 und 152/777-1 FUGG). Das Hochleistungsrechnen wurde durch den Zugang zum HPC am DESY/Hamburg (Deutschland) ermöglicht. Dieses Projekt wurde mit Mitteln der Behörde für Wissenschaft, Forschung und Gleichstellung der Freien und Hansestadt Hamburg am Institut für Struktur- und Systembiologie des Universitätsklinikums Hamburg-Eppendorf (UKE), sowie durch das Deutsches Elektronen Synchrotron (DESY), gefördert. Unser besonderer Dank gilt Dr. Rory Hennell James für seine Anregungen bei der Modellierung von Liganden. Wir danken auch Dr. Stephan Niebling vom EMBL am CSSB für seine Hilfe bei der ITC-Datenanalyse und Prof. Dr. Sebastian Hiller für die Bereitstellung des Expressionsplasmids mit BamABCDE. Open Access Veröffentlichung ermöglicht und organisiert durch Projekt DEAL.
Interessenkonflikt
Die Autoren erklären, dass keine Interessenkonflikte vorliegen.
Open Research
Data Availability Statement
Die Daten, die die Ergebnisse dieser Studie unterstützen, sind auf begründete Anfrage beim Autor erhältlich.