Multi-Wellenlängen-Photopolymerisation von stabilen Poly(katecholamin)-DNA-Origami-Nanostrukturen**
Eine frühere Version dieses Manuskripts ist auf einem Preprint-Server hinterlegt worden (https://doi.org/10.33774/chemrxiv-2021-dvkpb).
Abstract
Die Herstellung von präzisen Multikomponenten-Polymerhybriden ist eine chemische Herausforderung bei der Bottom-up-Synthese komplexer Nanostrukturen. Wir nutzen die einzigartige Adressierbarkeit der DNA-Origami-Technik, um ein auf mehrere Wellenlängen ansprechendes Photopolymerisationssystem mit Nanometerauflösung zu realisieren. Durch die positionsgenaue Immobilisierung verschiedener Photosensibilisatoren auf dem Origami-Grundgerüst und deren Aktivierung bei verschiedenen, für den jeweiligen Sensibilisator charakteristischen Wellenlängen können wir sequenzielle Polymerisationsprozesse präzise steuern. Die angeregten Photosensibilisatoren erzeugen reaktive Sauerstoffspezies, welche wiederum die Polymerisation der Katecholamine Dopamin und Norepinephrin einleiten. So bilden sich polymere Schichten an festgelegten Positionen auf der DNA-Origamistruktur, wodurch der polyanionische Charakter der DNA-Objekte verändert, ihre Aufnahme in Zellen gefördert und gleichzeitig ihre Integrität bewahrt wird. Die von uns vorgestellte Methode bietet eine schnelle Plattform für den Zugang zu komplexen 3D-Nanostrukturen durch die Anpassung von Material und biologischen Grenzflächen.
Einleitung
Die räumliche Kontrolle und Konstruktion von Objekten mit einer Auflösung im Nanometerbereich ist für die Miniaturisierung von intelligenten Materialien und Geräten unerlässlich. Sowohl in der Materialwissenschaft als auch in der Biomedizin besteht Bedarf an Werkzeugen zur Konstruktion von dreidimensionalen Multikomponenten-Substrukturen, um das Verständnis dafür zu erweitern, wie Oberflächenmuster und Objektkonturen die Grenzflächenkräfte modulieren. Die Konstruktion von nanostrukturierten Oberflächen auf weichen Materialien, die frei anpassbar sind, stellt jedoch ein Nadelöhr dar, da es an Werkzeugen für deren präzises Design fehlt. Dieses Problem wird noch verschärft, wenn die Muster viel kleiner sind als die Wellenlänge des Lichts, wo Top-down-Ansätze wie die Lithografie an ihre Grenzen stoßen. Auf dieser Längenskala bieten Bottom-up-Strategien auf Grundlage der Selbstassemblierung eine logische Ergänzung für die Herstellung von strukturierten, weichen Nanomaterialien. Im Gegensatz zu den Limitierungen anderer Systeme, die auf synthetischen Polymeren oder Peptiden basieren, verfügt die DNA-Nanotechnologie über die notwendige Präzision, um nanostrukturierte Oberflächen passgenau herzustellen.1 In Verbindung mit dem DNA-Origami-Strukturdesign2 haben Konzepte zur Untersuchung von Epitopen,3 Proteinanordnungen,4 plasmonischen Geräten,5 und Biosensoren6 in jüngster Zeit zu entscheidenden Erkenntnissen in der Nanomedizin und Biophysik geführt.
In der Polymerchemie und -strukturierung haben Fortschritte bei der radikalischen und oxidativen Polymerisation sowie bei der Organisation von Polymerketten dazu beigetragen, dass diese mit der DNA-Origami-Technik, die strikten Bedingungen unterliegt, kombiniert werden können.7 Umgekehrt resultierte aus der Kombination von DNA-Nanostrukturen mit geladenen Molekülen und Polymeren eine erhöhte Stabilität unter wässrigen, physiologischen Bedingungen sowie sogar in organischen Lösungsmitteln. Diese neuen Eigenschaften waren für die rasche Expansion der DNA-Origami-Plattform in den letzten Jahren maßgeblich verantwortlich.8 Das Potential von Polymer-DNA-Objekten ist allerdings durch die strukturierte Positionierung von Polymeren auf DNA-Origami-Gerüsten mittels Hybridisierung bei weitem noch nicht ausgeschöpft.9
In dieser Arbeit konnten wir Photopolymerisationsreaktionen bei mehreren Wellenlängen auf DNA-Nanostrukturen durchführen, um Polymermuster und mehrschichtige Strukturen mit hoher räumlicher Präzision herzustellen (Abbildung 1). Diese Studien bauen auf Vorarbeiten auf, in denen wir zeigen konnten, dass Dopamin (DA) durch den Photosensibilisator Protoporphyrin IX (PPIX), das in einer G-Quadruplex(G4)-Sequenz gebunden vorliegt, photopolymerisiert werden kann, wodurch Polydopamin an festgelegten Positionen gebildet wird.10 Darauf aufbauend konnten wir nun durch die Verwendung von G4-Sequenzen mit den Photosensibilisatoren Eosin Y (EY) und Methylenblau (MB), die durch grünes (525 nm) bzw. rotes (625 nm) Licht aktiviert werden, eine breite Wellenlängenflexibilität für die Anregung und die lokale Produktion reaktiver Sauerstoffspezies (ROS) realisieren. Zusammen mit dem durch blaues Licht angeregten PPIX-Sensibilisator (410 nm) erzeugen diese drei Katalysatorzentren ROS bei ihrer jeweiligen charakteristischen Wellenlänge und leiten so die lokale Polymerisation von Katecholaminen ein. Anhand von Dopamin und Norepinephrin (NE) konnten wir demonstrieren, dass beide Monomere sequenziell und in definierten Mustern und Dimensionen polymerisiert werden können. Durch die Bildung einer Polymerschicht ändern sich die ansonsten polyanionischen Eigenschaften des DNA-Origamis und erscheinen weniger elektronegativ, wodurch eine Aufnahme in lebende Zellen ermöglicht wird, die durch Fluoreszenz-Kolokalisierungsstudien verfolgt werden konnte. Unser Ansatz ermöglicht eine schnelle und einfache Synthese von Multikomponenten-Polymermustern auf DNA-Origami mit präzisen Formen und Abmessungen. Die Anpassung der Grenzflächen von DNA-Polymer-Objekten durch eine gezielte Oberflächenmodulation ist für verschiedene Anwendungen von entscheidender Bedeutung, z. B. für die Wechselwirkung mit Membranen und für die zelluläre Aufnahme beispielsweise für den Medikamententransport.
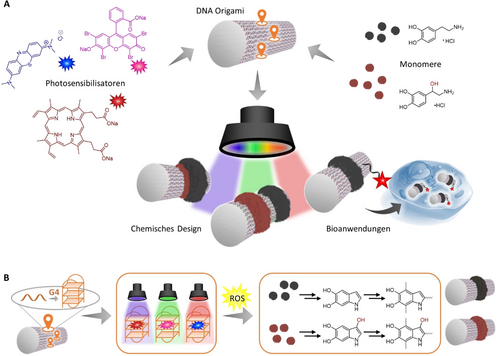
A) Durch die Kombination von verschiedenen Photosensibilisatoren und zwei verschiedenen Katecholaminmonomeren kann eine Multiwellenlängen-Photopolymerisation auf DNA-Origami erreicht werden. Auf diese Weise können Polymere unter zeitlicher und räumlicher Kontrolle an festgelegten Stellen auf der DNA-Nanostruktur gebildet werden. Polymer-DNA-Hybridstrukturen können genutzt werden, um Wechselwirkungen an der zellulären Schnittstelle zu modulieren. B) Die Reaktionszentren, bestehend aus G-Quadruplex-Strukturen (G4) und eingelagerten Photosensibilisatoren, erzeugen reaktive Sauerstoffspezies (ROS) bei ihrer charakteristischen Wellenlänge und leiten so die Polymerisation ein. Mechanismus und Struktur sowohl von Polydopamin11 als auch von Norepinephrin12 sind vielfältig und noch immer Gegenstand aktueller Forschung. Aus Gründen der Übersichtlichkeit sind hier nur einige repräsentative Strukturen abgebildet. Weitere Informationen über den Mechanismus, die Zwischenstufen und die vorherrschenden Wechselwirkungen finden sich in den Hintergrundinformationen (Abbildung S2).
Ergebnisse und Diskussion
Um dreidimensionale DNA-Origami-Nanoröhren als Gerüststrukturen für die Photopolymerisation zu verwenden, wurden G-Quadruplex-Strukturen auf der DNA-Oberfläche in bestimmten Mustern angeordnet. Die katalytischen Zentren können durch Wahl eines passenden Photosensibilisators, der innerhalb des G-Quadruplex sitzt (5′-GGG TA GGG C GGG TT GGG-3′), realisiert und auf Wellenlängenselektivität abgestimmt werden. Der so gebildete PPIX-G4-Komplex erzeugt unter Weißlichtbestrahlung ROS, die ihrerseits die Oxidation und Polymerisation von Dopamin auslösen. Um die bereits bekannte Selbstpolymerisation von Dopamin in Lösung zu unterdrücken und die Polymerbildung lokal auf der DNA-Origami-Oberfläche zu kontrollieren, ist es entscheidend, in einer leicht sauren Umgebung (pH 6.5) zu arbeiten. Dabei haben wir festgestellt, dass nur energiereiches, blaues Licht (410 nm) genügend Energie besitzt, um die Polymerisation von Dopamin zu initiieren (Hintergrundinformationen Abbildung S1). Die Anregung der Q-Banden von PPIX-G4 im sichtbaren Spektrum des Lichts reichte nicht aus, um genügend oxidierte Dopaminspezies zu bilden und so die Polymerisation voranzutreiben. Wellenlängenspezifität im grünen und roten Bereich wiederum wurde durch die Aufnahme von EY13 und MB14 in das G4-Motiv erreicht. Die Fähigkeit der einzelnen Katalysatoren, Singulett-Sauerstoff (1O2) zu erzeugen, wurde mit einem Test auf der Grundlage von Imidazol und p-Nitrosodimethylanilin (RNO) untersucht.15 Sowohl MB als auch EY zeigten eine höhere Effizienz bei der Produktion von 1O2 als PPIX, was sich in einer 7- bzw. 11-mal schnelleren Entfärbung von RNO widerspiegelt (Hintergrundinformationen Abbildung S3). Anschließend wurde versucht, die Polymerisation mit EY-G4 und MB-G4 auf DNA-Origami-Nanoröhren bei den jeweiligen Wellenlängen (EY: 525 nm, MB: 625 nm) zu initiieren. Dafür wurde das Origami-Gerüst mit einem zentralen Ring aus G4-Sequenzen ausgestattet, welche die Photosensibilisatoren beherbergen. Bei Verwendung von 10 mm Dopamin in 100 mm Puffer (pH 6.5) zeigte die UV/Vis-Spektroskopie nach drei Stunden eine erfolgreiche Polymerisation zu Polydopamin (pDA) (Hintergrundinformationen Abbildung S4A). Die Bildung von Zwischenprodukten wie Dopaminochrom (320 nm), oxidierten Oligomeren (480 nm) und schließlich pDA (700 nm) konnte anhand ihrer charakteristischen Absorptionswerte verfolgt werden.7c Die räumliche Kontrolle über die Polymerisation und die daraus resultierende Nanostruktur wurde durch Rasterkraftmikroskopie (AFM) überprüft (Hintergrundinformationen Abbildung S4B). Es wurde erfolgreich ein Polymerring an jenen Positionen gebildet, an welchen die G4-Sequenzen angefügt wurden, was zeigt, dass der Wechsel der katalytischen Zentren und der Anregungswellenlängen die Kontrolle über die Polymerisationsreaktion nicht beeinträchtigt. Im Vergleich dazu zeigte die Reaktionskinetik der oxidativen Polymerisation, dass die Bildung jedes Zwischenprodukts (Dopaminochrom, Oligomere usw.) einschließlich pDA für EY effizienter war (Hintergrundinformationen Abbildung S4C). Trotz der beobachteten Unterschiede in der Reaktionskinetik wies die Analyse des topologischen Höhenprofils mittels AFM keine signifikanten Höhenunterschiede zwischen den verschiedenen Photosensibilisatoren auf (Hintergrundinformationen Abbildung S4D).
Anschließend untersuchten wir verschiedene Muster und den Einfluss der Größe der angebrachten G4-Zentren auf den Polymerisationsprozess. Zunächst entwarfen wir eine Origami-Nanoröhre mit einem diagonalen G4-Motiv von ähnlicher Dichte wie das Standard-Ringmuster (Hintergrundinformationen Abbildung S5). Nach der Polymerisation konnten wir die Bildung von pDA auf den Nanoröhren entlang des vorhergesehenen Musters feststellen, abhängig von der Beobachtungsperspektive. Die fehlende Symmetrie bedeutet, dass die Ausrichtung der DNA-Origami-Strukturen auf der Glimmeroberfläche einer inhärenten Zufälligkeit unterliegt und somit den Abbildungsprozess beeinflusst. Aus diesem Grund halten wir Ringmuster für die zuverlässigste Methode, um eine robuste Charakterisierung zu ermöglichen. Darüber hinaus haben wir die Korrelation zwischen der Polymerbildung und der Breite des Ringsystems durch direkten Vergleich der in Abbildung 2 dargestellten Strukturen untersucht. Die entsprechenden AFM-Bilder zeigen, dass bei lediglich 22 photokatalytischen Zentren die Polymerisationsreaktionen nicht auf jedem DNA-Objekt zuverlässig ausgelöst werden und die lokal gebildeten ROS für weitere Oxidationsreaktionen wahrscheinlich nicht ausreichten. Darüber hinaus ist die Höhe der untersuchten Polymerringe im Vergleich zur Standard-Origami-Nanoröhre (44 G4-Sequenzen) geringer. Hier werden Gesamthöhen von typischerweise 10–15 nm erreicht. Verdoppelt man die Anzahl der G4-Strukturen auf 88, so wachsen die Polymerringe auf fast jedem Origami gleichmäßig und die Gesamthöhe ist ähnlich wie bei den standardmäßig 44 katalytischen Zentren. Unsere Ergebnisse deuten darauf hin, dass in der Tat eine minimale Anzahl von gebündelten katalytischen Zentren erforderlich ist, um die Polymerisation einzuleiten. Was die Aktivität dieser Zentren betrifft, so scheint es keine Obergrenze zu geben, die das System einschränkt. Eine vollständige Bedeckung der Origamioberfläche mit G4-Sequenzen würde jedoch einerseits weitere Modifikationen einschränken und andererseits die Integrität der Origami-Struktur aufgrund höherer Spannungen beim Aufrollen der DNA-Nanoröhre beeinträchtigen. Auch die Art des neu gebildeten Polymers spielt für die Stabilität der DNA-Objekte eine wichtige Rolle. Wird pDA gebildet, so neigen die mit Polymerringen versehenen DNA-Origami-Objekte aufgrund der starken Adhäsion von pDA zur Aggregation (Hintergrundinformationen Abbildung S6). Daher wurde Norepinephrin als Dopamin-Analogon eingeführt, um gut dispergierbare Nanoobjekte zu erhalten, die auch in komplexen Medien ohne Bildung von Aggregaten stabil bleiben. Norepinephrin gehört ebenfalls zur Familie der Katecholamine, und Poly(norepinephrin) (pNE) zeigt ähnliche materialunabhängige Modifizierungsfähigkeiten wie pDA, jedoch mit einer ultraglatten Oberflächenmorphologie.12c, 16 Chemisch gesehen besitzt NE eine zusätzliche Hydroxygruppe, und diese Zunahme der hydrophilen Wechselwirkungen könnte möglicherweise einen wichtigen Beitrag zur kolloidalen Stabilität der gebildeten Nanostrukturen leisten. Im Gegensatz zu pDA zeigte die Polymerisation zu pNE auf DNA-Origami unter Verwendung aller drei Photosensibilisatoren eine starke Präferenz für EY und MB (Hintergrundinformationen Abbildung S7). Diese Beobachtungen, die durch UV/Vis-Spektroskopie gemacht wurden, deuten darauf hin, dass die Oxidation von pNE einen leistungsfähigeren Photosensibilisator erfordert, um die Polymerisationsreaktion zu initiieren. Ebenso zeigte die Bildung von pNE auf dem DNA-Origami, dass die Polymerisation mit EY und MB als Photosensibilisator effizienter war.
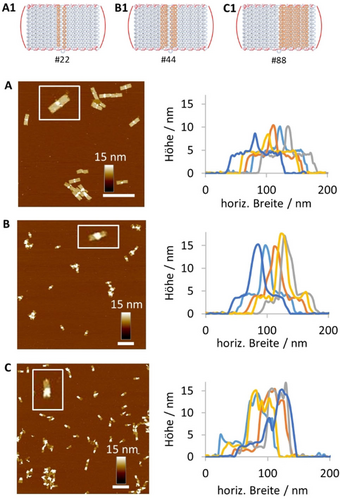
Studien über die Korrelation zwischen der Polymerbildung und der Anzahl der katalytischen G4-Zentren. (A1–C1) Es wurden DNA-Origami-Nanoröhren entworfen, die einen Ring aus 22, 44 bzw. 88 G4-Sequenzen tragen. Die Nanoröhren werden durch Hybridisierung der Faltstränge (in Rot dargestellt) aufgerollt. A–C) AFM-topografische Bilder der Origami-Nanoröhren mit pDA-Ring zeigen, dass eine minimale Anzahl von 44 katalytischen Zentren erforderlich ist, um die Polymerisation zuverlässig einzuleiten.
In Studien zur dynamischen Lichtstreuung (DLS) führte die Bildung eines pNE-Rings zu einer deutlichen Verschiebung der intensitätsbasierten Größenverteilung hin zu höheren hydrodynamischen Durchmessern, was sich auch in den z-Mittelwerten zeigt (Abbildung 3). Darüber hinaus sind sowohl unfunktionalisierte Origami-Strukturen als auch Origami mit pNE-Ring für mindestens drei Tage stabil und zeigen keine Agglomeration (Hintergrundinformationen Abbildung S8).
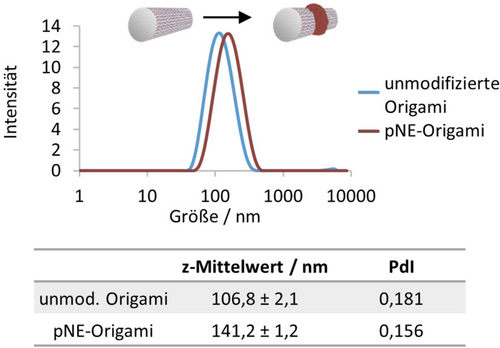
DLS-Charakterisierung von unmodifizierten Origami-Nanoröhren und Origami-Nanoröhren mit pNE-Ringen. Das Polymerwachstum auf dem Origami führt zu einer deutlichen Verschiebung der intensitätsgewichteten Größenverteilung und der z-Mittelwerte. Die Zahlen sollten nur als qualitativer Hinweis betrachtet werden, da die DLS nach den Prinzipien kugelförmiger Objekte arbeitet, was diese Origami-Strukturen nicht genau abbildet.
Zusätzlich ergänzt die Agarosegel-Elektrophorese (AGE) die Charakterisierung der Origami-Objekte vor und nach der Polymerisation der pDA- und pNE-Polymere (Hintergrundinformationen Abbildung S9).
Auf der Grundlage der ermittelten Reaktionsbedingungen und Wellenlängenselektivitäten für DA und NE wurden sequenzielle Polymerisationsschritte zur Herstellung von Multikomponenten-Nanostrukturen durchgeführt. Die DNA-Origami wurden mit MB-G4s beladen, und NE und DA wurden nacheinander bei 625 nm für jeweils zwei Stunden polymerisiert (Abbildung 4A). Die erfolgreiche Polymerisation wurde für beide Bestrahlungsphasen anhand der charakteristischen Profile für pNE und pDA nachgewiesen (Hintergrundinformationen Abbildung S10). Die erste Bestrahlungsphase führte zur Bildung einer pNE-Schicht von 5.2 nm±1.5 nm Höhe (Abbildung 4B). Danach wurden überschüssiges NE und oxidierte Nebenprodukte durch Spinfiltration entfernt und durch DA ersetzt. In der zweiten Bestrahlungsphase bildete sich die pDA-Schicht, die eine zusätzliche Höhenzunahme von 4.3 nm±1.8 nm (Gesamthöhe: 9.5 nm±1.8 nm) bewirkte (Abbildung 4B). Die Höhenzunahme der einzelnen Komponenten korreliert gut mit ihren individuellen Einzelpolymerisationen. Um nachzuweisen, dass die zweite Bestrahlungsphase die Polymerisation von DA und nicht von bestehendem pNE (oder dessen adsorbierten Oligomeren) ausgelöst hat, wurde ein Kontrollversuch ohne Zugabe von DA durchgeführt. In diesem Fall wurden keine zusätzlichen Polymere gebildet (Hintergrundinformationen Abbildung S11). Die Obergrenze der geschichteten Komponenten wird durch den Zugang der Monomere zu den katalytischen Zentren bestimmt. Bei einer Gesamthöhe von etwa 10–15 nm kann die Polymerisation nicht mehr durch den Photosensibilisator-G4-Komplex gesteuert werden.
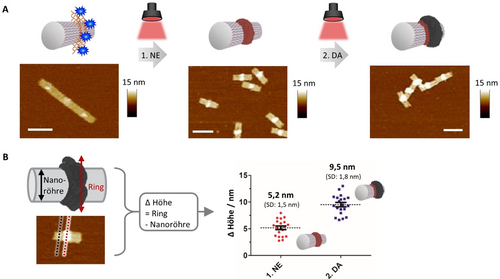
Ein zweistufiger Polymerisationsprozess demonstriert das Potenzial des Origami-Systems, eine schichtweise Bildung von Polymeren zu induzieren. A) DNA-Origami-Röhrchen werden mit Methylenblau als Photosensibilisator inkubiert und bei 625 nm bestrahlt. In der ersten Beleuchtungsphase wird Norepinephrin (NE) polymerisiert, gefolgt von einem Reinigungsschritt, um überschüssiges NE zu entfernen, und Dopamin (DA) wird als zweites Monomer hinzugefügt, das ebenfalls unter Rotlichtbestrahlung polymerisiert. AFM-Bilder zeigen das topografische Profil der DNA-Origami-Objekte: Das Polymer ist auf die G4-Muster aufgedruckt. Maßstabsbalken entsprechen 100 nm. B) Um die Höhe der Polymerschichten quantitativ zu bestimmen, wurden die z-Werte aufgezeichnet und wie dargestellt berechnet (n=20; Fehlerbalken sind SEM).
Als Nächstes demonstrierten wir die Wellenlängen-Orthogonalität für eine zweistufige Polymerisation, um Reaktionsselektivität bei einer bestimmten Wellenlänge zu erreichen. Zunächst wurde untersucht, ob MB und EY sich bei entgegengesetzten Wellenlängen inaktiv verhalten. MB zeigte keine Bildung von oxidierten DA-Spezies unter Bestrahlung bei 525 nm, und umgekehrt verhielt sich EY bei 625 nm nicht aktiv (Hintergrundinformationen Abbildung S12). Um sicherzustellen, dass prinzipiell eine fortlaufende Aktivierung jedes Photosensibilisators kontinuierlich die Polymerisation von Dopamin auslösen könnte, wurden die DNA-Origami-Röhrchen an jedem Röhrenende mit Ringmustern von EY-G4 und MB-G4 bestückt. Die Polymerisation von DA (10 mm, pH 6.5) wurde durch sequenzielle Bestrahlung bei 625 nm und 525 nm für jeweils 3 Stunden veranlasst. Die Bildung von oxidierten Zwischenprodukten und pDA wurde durch UV/Vis-Spektroskopie überprüft (Hintergrundinformationen Abbildung S13A). Kontrollversuche stellten sicher, dass die Integrität des DNA-Origami-Röhrchens durch die verlängerte Bestrahlung und durch die von den Photosensibilisatoren erzeugten ROS nicht eingeschränkt wurde (Hintergrundinformationen Abbildung S13B).
Um die Methodik zu erweitern, haben wir die Beladung mit den EY- und MB-Photosensibilisatoren schrittweise entkoppelt, um zu zeigen, dass die DNA-Hybridisierung auch nach der Polymerisation robust und zuverlässig ist (Abbildung 5). Im ersten Schritt wurde MB auf einem DNA-Origami-Röhrchen angebracht, das mit einem einzigen Ring aus G4-Sequenzen ausgestattet war. Die Bestrahlung bei 625 nm für 2 Stunden in Anwesenheit von NE (10 mm, pH 6.5) bildete den ersten Polymerring, der durch AFM und UV/Vis-Spektroskopie nachgewiesen werden konnte (Abbildung 5B,C).
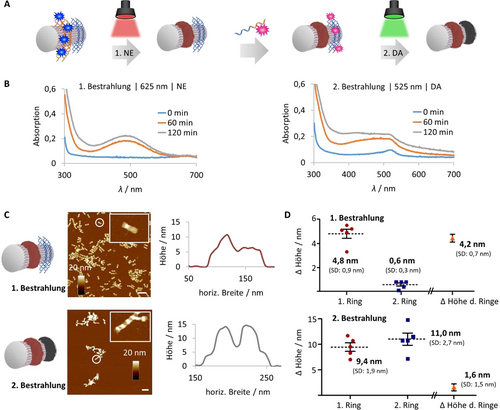
In einer Weiterentwicklung des zweistufigen Polymerisationsverfahrens wurden DNA-Origami-Röhrchen mit zwei verschiedenen Photosensibilisatoren ausgestattet und mit zwei verschiedenen Monomeren inkubiert, die bei unterschiedlichen Wellenlängen die Polymerbildung an verschiedenen Ringen auslösten. A) DNA-Origami-Röhrchen wurden mit einem Ring aus G4-Sequenzen (orange) und einem Ring aus überstehenden, einzelsträngigen DNA-Sequenzen (blau) versehen. MB wurde auf die G4-Sequenzen geladen, NE wurde hinzugefügt und bei 625 nm bestrahlt. Nach der ersten Bestrahlungsphase wurde NE durch Spinfiltration entfernt, EY-G4 über Hybridisierung an den komplementären Ring angebracht, und die pDA-Bildung wurde bei 525 nm ausgelöst. B) UV/Vis-Spektren zeigen die charakteristischen Profile für die Bildung von pNE bzw. pDA. C) AFM-Aufnahmen der Origami-Nanoröhren nach jedem Schritt wurden durchgeführt, um die Bildung des ersten und des zweiten Polymerrings zu verfolgen und zu vergleichen. Maßstabsbalken entsprechen 200 nm. Repräsentative Höhenprofile von 1-Ring- und 2-Ring-Strukturen sind abgebildet. D) Die Histogramme zeigen, dass im ersten Schritt nur ein Polymerring gebildet wird, während im zweiten Schritt ein zweiter Ring wächst. Beide Ringe haben ähnliche Höhen (n=5; Fehlerbalken sind SEM).
Überschüssiges NE wurde durch Spinfiltration entfernt und der zweite Photosensibilisator, EY-G4, wurde mit Hilfe einer Temperaturrampe auf das gegenüberliegende Ringmuster desselben DNA-Röhrchens hybridisiert. Der zweite Polymerisationsschritt wurde bei 525 nm für 2 Stunden mit DA (10 mm, pH 6.5) als Monomer durchgeführt, um die finale Nanostruktur zu erhalten, bei der pNE und pDA jeweils einen einzelnen Ring besetzen. Die Oxidationsprofile sowohl von NE als auch von DA in diesem Zweikomponentensystem zeigten eine konsistente Polymerisationskinetik im Vergleich zum Einkomponentensystem. Darüber hinaus kann durch den Vergleich der durchschnittlichen Polymerhöhen jeder Bestrahlungsperiode die sequenzielle Ringbildung verfolgt werden (Abbildung 5D). Im ersten Schritt wurde nur an einer Stelle des Origami Polymer gebildet (4.8±0.9 nm), was sich auch deutlich im Höhenunterschied zwischen diesem Polymerring und dem benachbarten Ringmuster zeigt (Δ von 4.2±0.7 nm). Im zweiten Schritt wurde ein zweiter Polymerring an der vorgesehenen Stelle gebildet, dessen Höhe die Dimensionen des zuvor gezüchteten Rings erreichte. Beide Polymere weisen eine ähnliche Höhe auf (Δ von 1.6±1.5 nm). Es ist daher wichtig anzumerken, dass der vorhandene Ring als zusätzliches Keimbildungszentrum fungiert, so dass aktivierte Spezies von dem benachbarten Ring dorthin diffundieren können, was zu einer weiteren Höhenzunahme während der Aktivierung des zweiten Rings führt. Weiterhin ist die Anwesenheit aller reaktiver Komponenten, d. h. der Monomere, der Photosensibilisatoren auf dem DNA-Origami und der Lichtquelle, für die Bildung der erwünschten hybriden Nanostruktur unerlässlich. Kontrollversuche nur mit Monomeren ohne Bestrahlung oder ohne eingebetteten Photosensibilisator zeigten keine Polymerisation (Hintergrundinformationen Abbildung S14). Sowohl bei den gekoppelten als auch bei den entkoppelten Methoden konnten wir demonstrieren, dass der Zugang zu anspruchsvollen und aus mehreren Komponenten bestehenden, dreidimensionalen DNA-Polymer-Hybriden einfach zu erreichen ist.
Darüber hinaus kann die Bildung dieser Polymermuster zur Anpassung der Oberflächenchemie des DNA-Origami genutzt werden. Grundsätzlich sind DNA-Origami-Strukturen aufgrund des Polyphosphat-Rückgrats stark anionisch und benötigen daher zweiwertige Kationen zur Stabilisierung in wässriger Lösung.17 Unter physiologischen Bedingungen sind DNA-Origami-Strukturen anfällig für den Abbau durch Nukleasen und zu geringe Konzentrationen von zweiwertigen Kationen.18 Weiterhin hat auch die DNA-Nanostruktur selbst einen großen Einfluss auf die Stabilität der Objekte unter diesen Bedingungen.19 Dreidimensionale Strukturen beispielsweise verlangsamen die Verdauungsgeschwindigkeit von Nukleasen im Vergleich zu entsprechenden 2D-Objekten erheblich.20 Vor diesem Hintergrund haben wir unsere DNA-Strukturen, d. h. unmodifziertes Origami und Origami mit Polymerringen, den Bedingungen im Zellmedium ausgesetzt, wie sie bei Studien zur Zellaufnahme gegeben sind. Die nativen DNA-Strukturen weisen ein höheres Maß an Fragmentierung auf als pNE-Origami, wenn sie 24 Stunden lang bei 37 °C in Zellmedium inkubiert werden (Hintergrundinformationen Abbildung S15).
Neben der fehlenden Stabilität unter physiologischen Bedingungen verhindert auch der polyanionische Charakter der DNA die zelluläre Aufnahme aufgrund von Abstoßungskräften gegen die negativ geladene Zellmembran. Zu den bestehenden Strategien zur Bewältigung dieser Herausforderung gehört das Anbringen von zielgerichteten Einheiten (z. B. Peptide, Proteine und Aptamere), die die rezeptorvermittelte Endozytose fördern.21 Allerdings könnten die Polymermuster auf den Origami-Nanostrukturen die lokale Ladungsabstoßung verringern, wodurch ein Transport durch die Zellmembran ermöglicht werden könnte. Die entsprechend modifizierten DNA-Origami-Nanostrukturen wurden entweder mit pNE oder pDA synthetisiert und mit Alexa-647®-Oligonukleotiden versehen (Hintergrundinformationen Abbildung S16). Bei 10 nm wurde mittels konfokaler Laser-Scanning-Mikroskopie eine effiziente, zelluläre Aufnahme von pNE-Origami in A549-Lungenadenokarzinomzellen nach 24-stündiger Inkubation beobachtet (Hintergrundinformationen Abbildung S17). Bei vergleichbaren pDA-Origami wurde jedoch eine erhebliche Aggregation festgestellt, welche auf die bekannte Adhäsionseigenschaft von pDA zurückzuführen ist. In Übereinstimmung mit der Literatur zeigen nur mit Alexa-647®-markierte DNA-Origami-Nanostrukturen keine Aufnahme in Zellen, ebenso wenig wie das freie Alexa-647®-Oligonukleotid. Um die Stabilität der internalisierten Origami-Proben weiter zu charakterisieren, wurde ein weiterer Fluorophor, Alexa-488®, an das gegenüberliegende Ende des Röhrchens angebracht, um Kolokalisierungsstudien zu ermöglichen (Schema in Abbildung 6A). Das doppelt markierte Origami wurde durch AGE und anschließende Gelextraktion charakterisiert (Hintergrundinformationen Abbildung S19). Durch Überlagerung beider Kanäle im Mikroskop wird die Kolokalisierung der beiden Fluorophore deutlich sichtbar, was darauf hindeutet, dass ein großer Teil der pNE-Origami-Strukturen nach der Internalisierung intakt bleibt (Hintergrundinformationen Abbildung S18). In ähnlicher Weise zeigten Kontrollen mit unmodifizierten Origami-Strukturen keine zelluläre Aufnahme, während das Aggregationsverhalten von pDA-Origami abermals deutlich erkennbar auftrat. Auch wenn die Fluoreszenz-Kolokalisierung der pNE-Origami-Proben positiv war und die Kontrollen die Möglichkeit ausschließen sollten, dass auch fragmentierte Komponenten aufgenommen werden, muss überprüft werden, ob sich das Material tatsächlich innerhalb der Zellen befindet. Mehrere Berichte haben gezeigt, dass das nachgewiesene Fluoreszenzsignal möglicherweise lediglich von an der Oberfläche haftendem oder abgebautem Material stammt.21c, 22 Aus diesem Grund wurden mit DNA-Origami behandelte Zellen mit DNase I inkubiert, um jegliche Anlagerungen an der Zellmembran abzubauen. Im Falle von pDA-Origami wurden die Aggregate durch die Behandlung mit DNase I entfernt, was bestätigte, dass die Fluoreszenzsignale größtenteils von membrangebundener Probe stammten (Abbildung 6B, Hintergrundinformationen Abbildung S18, S20). Bei den pNE-Origami wurde zwar auch eine deutliche Verringerung der Fluoreszenzintensität beobachtet, [21c] die z-Stapel-Analyse zeigte jedoch, dass die pNE-Origami erfolgreich internalisiert wurden (Abbildung 6B,C). Diese Experimente verdeutlichen, dass die adhärenten Eigenschaften von pDA vorherrschend waren und dass das Aggregationsverhalten die Aufnahme der Konjugate in die Zellen verhindert hat. Die kollektiven Beobachtungen deuten darauf hin, dass die Gegenwart der pNE-Schicht die abstoßenden Wechselwirkungen des DNA-Origami abschwächt und die Aufnahme in die Zellen erleichtert. Anhand dieser Zellexperimente konnten wir zeigen, dass Polymermuster auf DNA-Origami genutzt werden können, um Wechselwirkungen an der zellulären Schnittstelle zu verändern und zu modulieren, wodurch die Aufnahme von DNA-Objekten ermöglicht wird.
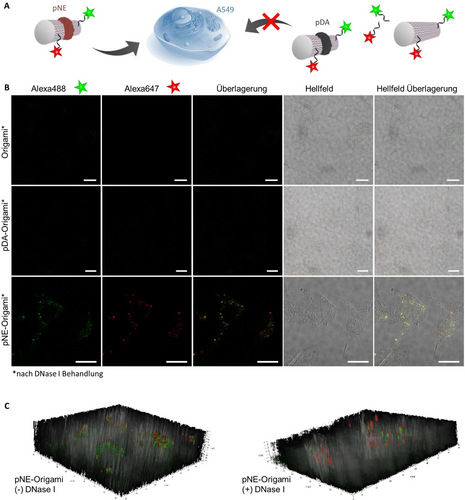
Zellstudien zur Kolokalisierung der Origami-Nanostrukturen. A) Schematische Darstellung der Alexa488- und Alexa647-Modifikation von bloßen DNA-Origami-Röhrchen sowie von pNE- und pDA-modifizierten Röhrchen. B) Konfokale Laser-Scanning-Aufnahmen von A549-Zellen, die 24 Stunden lang mit Alexa488- und Alexa647-doppelmarkierten DNA-Origami-Proben inkubiert wurden, wurden nach DNase-I-Behandlung aufgenommen. Maßstabsbalken entsprechen 20 μm. C) Z-Stapel von mit pNE-Origami inkubierten Zellen ohne und mit DNase-I-Behandlung zeigen, dass pNE-Origami in die Zelle aufgenommen werden. Kontrollbilder nur für Puffer und Alexa-Oligonukleotide sind in der Hintergrundinformationen zu finden (Abbildung S18A).
Zusammenfassung
Im Rahmen dieser Arbeit haben wir das strukturierte und schichtweise Wachstum verschiedener Polymere (pNE und pDA) auf DNA-Origami-Nanoröhren unter Verwendung von Licht verschiedener Wellenlängen erforscht. Die Wechselwirkung von G4 mit verschiedenen Photosensibilisatoren (PPIX, EY und MB) wird hierbei genutzt, um ihre Position auf der DNA-Origami-Plattform präzise festzulegen. Infolgedessen kann die Aktivität jedes Photokatalysators von einem aktiven zu einem ruhenden Zustand, und umgekehrt, gesteuert werden. Darüber bietet die Herstellungsmethode eine hohe Flexibilität, sodass die Abfolge der Photopolymerisationsreaktionen und/oder der Hybridisierungsschritte leicht geändert werden kann, ohne dass sich dies auf ihre Effizienz auswirkt. Das Ausmaß der Polymerbildung kann durch UV/Vis-Spektroskopie und AFM-Bildgebung direkt verfolgt werden, was eine strukturelle Anpassung in z-Richtung erleichtert. Darüber hinaus veränderten die Polymermuster den intrinsischen polyanionischen Charakter des DNA-Origami, während ihre Integrität erhalten blieb. Durch die Modulation der Abstoßungskräfte gegenüber der Zellmembran könnten diese Hybridobjekte für biologische Anwendungen genutzt werden. Zusammengenommen ist diese Plattform ein wertvolles Instrument zur Konstruktion komplexer Polymer-Origami-Architekturen, das die Untersuchung maßgeschneiderter Oberflächenmuster in den Nanowissenschaften und der Biomedizin ermöglicht.
Danksagung
Die Autoren danken der Deutsche Forschungsgemeinschaft (DFG) für die finanzielle Unterstützung – Projekt Nr. 407426226 – TRR 234 CataLight (B01) und dem Max-Planck-Bristol Centre for Minimal Biology. Open Access Veröffentlichung ermöglicht und organisiert durch Projekt DEAL.
Interessenkonflikt
Die Autoren erklären, dass keine Interessenkonflikte vorliegen.