Die Erzeugung von Tyrosinaseaktivität in einer Catecholoxidase erlaubt die Identifizierung der für die C-H-Aktivierung in Typ-III-Kupferenzymen verantwortlichen Aminosäurereste
Abstract
Tyrosinasen (TYRs) katalysieren die Hydroxylierung von Phenolen und die Oxidation der resultierenden o-Diphenole zu o-Chinonen, während Catecholoxidasen (COs) nur die letztere Aktivität aufweisen. Die Auronsynthase (AUS) reagiert nicht mit klassischen Tyrosinasesubstraten wie Tyramin und l-Tyrosin, jedoch hydroxyliert sie ihr natürliches Substrat: das Isoliquiritigenin. Der strukturelle Unterschied zwischen TYRs, COs und AUS, der zu ihren unterschiedlichen katalytischen Aktivitäten führt, ist noch immer ein Rätsel. Zu dessen Klärung wurde eine Bibliothek von 39 Mutanten der AUS von Coreopsis grandiflora (CgAUS) erstellt. Aktivitätsstudien an diesen zeigten, dass die Reaktivität der drei konservierten Histidine (HisA2, HisB1 und HisB2) durch ihre benachbarten Reste (HisB1+1, HisB2+1) und den Wasserträgerrest bestimmt wird, womit diese entweder als stärkere Basen reagieren oder/und in einer für das Verschieben von Substratprotonen günstigen Position stabilisiert werden. Diese Studie liefert das Verständnis für die CH-Aktivierung basierend auf dem Typ-III-Kupferzentrum, das in zukünftigen biotechnologischen Prozessen verwendet werden kann.
Die Typ-III Kupferproteinfamilie beinhaltet die Enzyme Tyrosinase (TYR), Catecholoxidase (CO) und Auronsynthase (AUS), die unter dem Oberbegriff Polyphenoloxidasen (PPOs) zusammengefasst sind.1, 2 Sie sind in Archaeen, Bakterien, Pilzen, Tieren und Pflanzen allgegenwärtig.3-5 TYRs sind bifunktionelle PPOs, die die o-Hydroxylierung von Monophenolen katalysieren (Monophenolaseaktivität, EC 1.14.18.1), an die eine anschließende Zwei-Elektronen-Oxidation der resultierenden o-Diphenole zu o-Chinonen gekoppelt ist (Diphenolaseaktivität, EC 1.10.3.1),2 wohingegen COs nur die letztere Diphenolase-Reaktivität zeigen. Die dazwischen liegende AUS katalysiert in vivo die Bildung von Auronen und Wildtyp-AUS aus Coreopsis grandiflora (CgAUSwt) zeigt eine schwache Hydroxylaseaktivität gegenüber ihrem natürlichen Substrat Isoliquiritigenin6, während sie nicht mit den klassischen TYR-Substraten l-Tyrosin und Tyramin reagiert und daher als CO eingestuft wurde.6, 7 PPOs sind an einem breiten Spektrum wichtiger natürlicher Reaktionen beteiligt wie dem Bräunungsprozess von Früchten,8 der Hautfarbe von Tieren bis hin zu menschlichen Krankheiten wie Albinismus,9 Melanome10 und neurodegenerative Erkrankungen (Parkinson).11
Die Suche nach dem strukturellen Unterschied zwischen TYRs und COs als Grundlage für ihre unterschiedlichen katalytischen Aktivitäten dauert nunmehr seit Jahrzehnten an. Die sehr große Übereinstimmung der aktiven Zentren der PPOs von Bakterien bis hin zu Tieren wurde durch 102 PDB-Einträge, die 18 PPOs aus 16 Organismen abdecken (Stand Juli 2020), beschrieben. Die Diskussion über die Aminosäuren, die für die unterschiedlichen Reaktivitäten entscheidend sind, wurde in neueren Artikeln zusammengefasst.12, 13-16 Die erste Erklärung für die fehlende Monophenolaseaktivität in COs war das Vorhandensein eines Phenylalanins, des sogenannten Torwächters oberhalb von CuA (Abbildung 1), der in der Süßkartoffel-CO (IbCO)-Struktur vorhanden ist.17, 18 Die erste Kristallstruktur einer pflanzlichen TYR aus Juglans regia (JrPPO1) zeigte jedoch auch das Vorhandensein dieses sperrigen Phenylalanins, was die Idee des Torwächters als den den Substratzugang blockierenden Rest widerlegte.19 Eine neuere Theorie besagt, dass die Deprotonierung des monophenolischen Substrats und folglich die Tyrosinaseaktivität von einem hochkonservierten Wassermolekül (Abbildung 1), das durch ein Asparagin (HisB1+1) neben dem ersten CuB-koordinierenden Histidin (HisB1) stabilisiert wird und durch ein konserviertes Glutamat, den sogenannten Wasserträger (Abbildung 1), aktiviert wird, herrührt.14 Der Asparaginrest an Position HisB1+1 kann jedoch nicht die alleinig verantwortliche Aminosäure sein, da Tyrosinaseaktivität gegenüber den klassischen Substraten für Pilz- (AbPPO4),20 Apfel- (MdPPO1 und MdPPO3),13, 21, 22 Larrea tridentata- (LtPPO)23 sowie Tomaten- (SlPPO1) tyrosinase,24 nachgewiesen wurde, die alle kein Asparagin an Position HisB1+1 enthalten. Kürzlich wurden zwei nicht konservierte Aminosäuren identifiziert und als “Aktivitätsregulatoren” bezeichnet, die neben den konservierten ersten (HisB1+1; Thr253 in CgAUSwt) und zweiten CuB-koordinierenden Histidinresten (HisB2+1; Arg257 in CgAUSwt) angeordnet sind, welche sich auf einer α-Helix vor dem katalytischen Zentrum der PPO (Abbildung 1) befinden.12a, 13 Mutagenesestudien an JrPPO1 und Taraxacum officinale-PPOs (ToPPO2, eine TYR und ToPPO6, eine CO) haben den Einfluss dieser Aminosäurereste auf die Tyrosinaseaktivität nachgewiesen.15, 16 Jedoch berichtet bisher keine Studie über die Erzeugung von Hydroxylaseaktivität in einer CO.
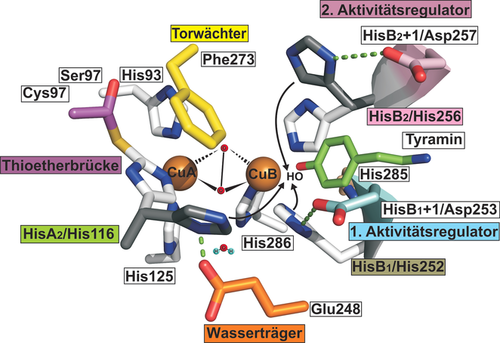
Aktives Zentrum der CgAUS-Mutante (Cys97Ser/Thr253Asp/Arg257Asp). Die mutierten Aktivitätsregulatoren Asp253 (Cyan, HisB1+1), Asp257 (Violett, HisB2+1), der Thioetherbrückenbestandteil Cys97 (lila, Ser97 in der Mutante), der Torwächter Phe273 (gelb) und der Wasserträger Glu248 (orange) sind hervorgehoben. Dargestellt sind die sechs konservierten Histidine His93 (HisA1), His116 (HisA2) und His125 (HisA3) von CuA und His252 (HisB1), His256 (HisB2) und His286 (HisB3) von CuB (Kohlenstoff in Weiß und Stickstoff in Blau). Das His285 (7. Histidin, weizenfarben) ist dargestellt und das Wasser neben Glu248 repräsentiert das konservierte Wasser, welches in eine Reihe hochauflösender Kristallstrukturen modelliert wurde.
Bislang haben die zahlreichen veröffentlichten Kristallstrukturen von Typ-III-Kupferproteinen und die erheblich geringere Anzahl von Mutationsstudien die Ursache für die Hydroxylaseaktivität in Typ-III-Kupferenzymen noch nicht aufgeklärt. Daher ist ein anderer Ansatz erforderlich, um diese ungeklärte Frage anzugehen. Dazu haben wir eine Bibliothek von 39 CgAUS-Mutanten erstellt, die sich auf die Reste neben den konservierten Histidinen (HisA2, HisB1 und HisB2), die sich in unmittelbarer Nähe des Substrats, und somit vor dem aktiven Zentrum befinden (Abbildung 1), konzentriert. In ähnlicher Weise wurden ausgewählte Enzyme mittels Umgestaltung des aktiven Zentrums mit einer neuen Aktivität ausgestattet (z. B. Esterase zu Aldolase oder Epoxidhydrolase sowie Untersuchungen zur Rolle von Phenylalanin und Tyrosin in Ammoniaklyasen bzw. Mutasen).25-27 Darüber hinaus sind Mutationen, die auf den Torwächter (Phe273), den Wasserträger (Glu248), das 7. Histidin (His285) und die zwei Cysteine (Cys31 und Cys32), die die konservierten Disulfidbrücken bilden (Abbildungen S1 und S2), abzielen in der Supporting Information zusammengefasst. Die CgAUS-Mutanten wurden heterolog in E. coli exprimiert, bis zur Homogenität gereinigt (Abbildung S3) und auf ihre Reaktivität mit den klassischen monophenolischen Substraten Tyramin (Abbildung S4) und l-Tyrosin (Abbildung S5) sowie den entsprechenden Diphenolen Dopamin (Abbildung S6) und l-Dopa (Abbildung S7) getestet. Die Km- und kcat-Werte für Tyramin und Dopamin, der Kupfergehalt aller Mutanten sowie die Fähigkeit der Mutanten zur Bildung des Oxyaddukts wurden photometrisch bestimmt (Tabelle 1, Tabelle S1). Alle öffentlich verfügbaren Kristallstrukturen von Typ-III-Kupferenzymen wurden bei der Interpretation unserer Daten berücksichtigt.
Mutanten |
Kupfer |
Dopamin |
Tyramin |
H2O2ϵ345 |
||
|---|---|---|---|---|---|---|
|
% |
kcat [s−1] |
Km [mm] |
kcat [s−1] |
Km [mm] |
[m−1 cm−1] |
CgAUSwt |
46.1±1.0 |
556±27.2 |
8.63±0.28 |
kA |
kA |
5190 |
|
||||||
Bestandteile der Thioetherbrücke |
||||||
Cys97Ala |
42.4±1.4 |
5.84±0.31 |
1.59±0.09 |
0.14±0.01 |
3.03±0.18 |
– |
Cys97Gly |
34.1±2.3 |
43±2.5 |
7.53±0.45 |
0.12±0.01 |
2.96±0.16 |
– |
Cys97Asp |
52.7±0.6 |
1.54±0.07 |
1.68±0.07 |
0.07±0.00 |
1.05±0.02 |
– |
Cys97Asn |
45.7±0.6 |
1.24±0.06 |
0.80±0.05 |
0.05±0.00 |
2.11±0.08 |
– |
Cys97Ser |
59.3±0.5 |
15±1.26 |
1.26±0.04 |
0.55±0.04 |
3.59±0.33 |
7600 |
|
||||||
HisB1+1 (1. Aktivitätsregulator) |
||||||
Thr253Asp |
45.0±0.9 |
530±33.7 |
1.93±0.12 |
2.14±0.16 |
30.9±7.27 |
3800 |
Thr253Asn |
83.6±2.5 |
850±46.5 |
4.99±0.38 |
1.19±0.14 |
11.5±2.69 |
19 840 |
Thr253Glu |
46.7±5.2 |
1394±90.7 |
3.59±0.22 |
0.21±0.02 |
11.5±1.06 |
7490 |
Thr253Gly |
72.8±0.9 |
337±24.3 |
8.74±0.92 |
0.07±0.00 |
2.22±0.14 |
8600 |
Thr253Ser |
52.7±0.0 |
500±29.0 |
6.62±0.37 |
0.01±0.00 |
1.18±0.09 |
8570 |
Thr253Cys |
56.9±1.8 |
312±14.8 |
11.1±0.33 |
0.04±0.00 |
4.32±0.28 |
14 220 |
Thr253Ala |
56.1±3.2 |
140±7.2 |
5.80±0.30 |
0.05±0.00 |
1.48±0.04 |
7850 |
Thr253Ile |
58.2±0.8 |
27±1.1 |
13.6±0.72 |
kA |
kA |
9980 |
Thr253Lys |
4.8±0.8 |
20±3.2 |
38.6±7.88 |
kA |
kA |
– |
|
||||||
HisB2+1 (2. Aktivitätsregulator) |
||||||
Arg257Asp |
11.1±0.8 |
1380±95.3 |
1.26±0.13 |
8.26±0.48 |
4.01±0.29 |
10 470 |
Arg257Leu |
64.5±2.1 |
2245±125 |
5.91±0.25 |
kA |
kA |
14 890 |
Arg257Ile |
66.3±1.2 |
1660±93.6 |
3.57±0.17 |
kA |
kA |
11 060 |
Arg257Gly |
79.6±1.8 |
1264±63.7 |
3.54±0.26 |
kA |
kA |
3490 |
|
||||||
HisB1+1 and HisB2+1 (1. und 2. Aktivitätsregulatoren) und Bestandteile der Thioetherbrücke |
||||||
Thr253Asp Arg257Asp |
43.5±1.7 |
171±10.2 |
0.24±0.02 |
9.48±0.55 |
1.09±0.09 |
4570 |
Thr253Asp Arg257Gly |
32.8±0.4 |
662±30.0 |
1.31±0.04 |
1.91±0.11 |
4.59±0.25 |
8610 |
Thr253Gly Arg257Leu |
33.8±0.8 |
535±39.9 |
4.00±0.46 |
0.05±0.00 |
0.75±0.07 |
7690 |
Thr253Ser Arg257Gly |
33.0±0.0 |
430±26.1 |
2.37±0.26 |
0.01±0.00 |
0.73±0.10 |
5660 |
Thr253Gly Arg257Val |
52.1±0.4 |
859±52.9 |
1.29±0.09 |
0.02±0.00 |
0.46±0.03 |
15 990 |
Thr253Gly Arg257Thr |
10.6±1.6 |
191±9.11 |
2.09±0.09 |
0.56±0.03 |
15.5±1.03 |
2620 |
Cys97Ser Thr253Asp Arg257Asp |
40.2±0.6 |
19±1.22 |
0.06±0.00 |
6.52±0.39 |
0.02±0.00 |
3700 |
Mutationen neben dem konservierten HisB1-Rest (1. Aktivitätsregulator): Es wurden neun Mutanten (Asp, Asn, Glu, Gly, Ser, Cys, Ala, Ile und Lys) hergestellt, die auf den nicht konservierten HisB1+1-Rest (genannt 1. Aktivitätsregulator; Thr253 in CgAUSwt) abzielen und hinsichtlich ihrer Hydroxylaseaktivität gegen Tyramin untersucht (Tabelle 1 und Abbildung S4). Die Reaktionsraten mit Tyramin von Thr253Asp, Thr253Asn und Thr253Glu, die entweder eine (deprotonierte) Carbonsäure (Asp und Glu) oder ein Carboxamid (Asn) enthielten, waren besonders hoch (kcat=2,14; 1,19 bzw. 0,21 s−1) und bemerkenswerterweise in der gleichen Größenordnung wie bei Wildtypen pflanzlicher TYRs z. B. Apfel MdPPO1-3 (kcat=9,5; 0,92 bzw. 1,0 s−1).13 Die Mutanten Thr253Gly, Thr253Ser, Thr253Cys und Thr253Ala zeigten signifikant niedrigere kcat-Werte, besitzen jedoch zweifellos Hydroxylaseaktivität, während Thr253Ile und Thr253Lys keine Monophenolaseaktivität aufwiesen und demzufolge COs blieben (Tabelle 1). Die Aminosäurereste His252 und sein Nachbar an Position 253 liegen aufgrund der Drehung der α-Helix relativ nahe beieinander (2,8 Å in CgAUSwt His252-Thr253, PDB: 4Z14) (Abbildung S8). Die höhere Hydroxylaseaktivität in Thr253Asp, Thr253Asn und Thr253Glu wird durch Wasserstoffbrückenbindungen erklärt, die die Aminosäurereste an Position 253 mit His252 (HisB1+1) bilden können und welche die Basizität des konservierten His252 erhöhen. Daher werden die ankommenden monophenolischen Substrate durch His252 deprotoniert, das als Base reagiert (Abbildung 2). Die schnellere Hydroxylierungsrate von Thr253Asp gegenüber Thr253Glu (2,14 gegenüber 0,21 s−1) zeigt die Bedeutung der genauen Position der aktivierenden Aminosäureseitenkette des HisB1+1-Restes. Die Glutaminsäure mit ihrer zusätzlichen CH2-Gruppe ist 4,7 Å von HisB1 entfernt und zeigt eine schwächere Wasserstoffbrückenwechselwirkung als Asp253 oder Asn253, die sich in einem Abstand von 2,6 bzw. 2,8 Å befinden (Abbildung S9). Die Wechselwirkungen von Asp oder Asn mit His ähneln katalytischen Triaden, wie sie in den gut charakterisierten Proteasen Chymotrypsin (PDB: 4H4F, Asp102-His57-Ser195 als Säure-Base-Nucleophil) und Papain (PDB: 1PPN, Asn175-His159-Cys25 als Stabilisator-Base-Nucleophil, Abbildung S10) auftreten.28 Eine Bildung von Schiff‘schen Basen im Übergangszustand und ein durch Glutamat unterstützter Protonentransfer während der Katalyse wurde für viele Reaktionsmechanismen berichtet, z. B. bei Aldolasen.29-31
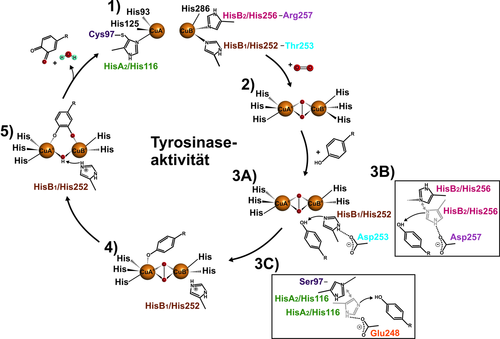
Tyrosinaseaktivität in Typ-III-Kupferenzymen. 1) Die Deoxyform von Typ-III-Kupferzentren (CuI-CuI) bindet molekularen Sauerstoff und geht dabei in die katalytisch kompetente Oxyform (CuII-CuII) über 2). Drei der sechs konservierten Histidine (HisA2, HisB1 und HisB2) sind für die Deprotonierung des ankommenden monophenolischen Substrats verantwortlich. 3 A) In der Thr253Asp-Mutante wird die Basizität von HisB1 durch den benachbarten Aminosäurerest Asp253 (HisB1+1) erhöht, was die Deprotonierung des ankommenden Substrats verbessern kann. 3 B) In ähnlicher Weise nähert sich HisB2 in der Arg257Asp-Mutante Asp257 (HisB2+1) und wird basischer. 3 C) In der Cys97Ser-Mutante wird HisA2 durch Entfernen der Thioetherbrücke freigesetzt und ist somit flexibel genug, um sich dem Wasserträger Glu248 zu nähern, basischer zu werden und das ankommende Substrat zu deprotonieren. 4) Das deprotonierte Monophenol ist nun kompetent für katalytisch produktive Wechselwirkungen mit der Oxyform des Typ-III-Kupferzentrums. 5) Die ortho-Hydroxylierung des Phenolats erfolgt über eine elektrophile aromatische Substitution, und die anschließende Zwei-Elektronen-Oxidation ergibt ein ortho-Chinon und Wasser. Diese beiden Elektronen reduzieren das Typ-III-Kupferzentrum in seine Deoxyform und schließen so den Katalysezyklus.
Mutationen neben dem konservierten HisB2-Rest (2. Aktivitätsregulator): Von den vier Mutanten (Asp, Leu, Ile und Gly), die auf den HisB2+1-Rest (2. Aktivitätsregulator) abzielen, gewann nur Arg257Asp Tyrosinaseaktivität, allerdings mit einer faszinierenden Umsatzrate (kcat=8,26 s−1). Die Erzeugung von Monophenolaseaktivität in Arg257Asp wird durch den Ersatz des positiv geladenen Arg257-Rests in CgAUSwt durch das negativ geladene Asp257 verursacht, wodurch die Basizität von His256 (HisB2) ähnlich der Aktivierung von His252 (HisB1) durch Asp253 erhöht wird. Die Seitenkette von Asp257 ist jedoch 6,3 Å vom konservierten His256 entfernt und kann daher die Basizität des konservierten His256 von dieser Position aus nicht direkt beeinflussen. Ungeachtet dessen sind die genannten Abstände nicht absolut zu verstehen, da eine große Flexibilität der beiden Kupferionen CuA und CuB und damit der konservierten Histidine mehrfach als Voraussetzung für die Funktionalität in PPOs beschrieben wurde.32, 33 Aufgrund dieser Flexibilität des konservierten His kann His256 näher an Asp257 heranrücken (2,6 Å) und eine Wasserstoffbrücke ausbilden, und somit Teil des Ladungsaustauschnetzwerks zur Deprotonierung des ankommenden Substrats werden (Abbildung S11).
Doppelmutanten an den HisB1+1- und HisB2+1-Resten (1. und 2. Aktivitätsregulatoren): Die Thr253Asp/Arg257Gly-, Thr253Gly/Arg257Leu- und Thr253Gly/Arg257Thr-Mutanten ahmten die charakterisierten und verifizierten TYRs AbPPO4,34 LtPPO23 und MdPPO313 nach. Passend zu ihren jeweiligen Aktivitätsregulatorkombinationen sind alle drei Doppelmutanten mit Tyrosinaseaktivität ausgestattet (Tabelle 1). Basierend auf den Ergebnissen, die für die Einzelmutanten an den HisB1+1- und HisB2+1-Resten erhalten wurden, wurde erwartet, dass die Doppelmutante Thr253Asp/Arg257Asp die stärkste TYR ist und tatsächlich zeigt sie die höchste Reaktionsrate auf Tyramin (kcat=9,48 s−1, Tabelle 1). Damit zeigt sich, dass die negativ geladenen Asparaginsäuren an den Positionen der Aktivitätsregulatoren TYRs mit bisher maximaler Tyrosinaseaktivität liefern.
Die Thioetherbrücke zwischen dem Cysteinschwefel und dem konservierten HisA2-Rest: CgAUSwt enthält eine Thioetherbrücke zwischen dem Cysteinschwefel (Cys97) und dem Cϵ des konservierten HisA2 (His116, siehe Abbildung 1). Diese Bindung fixiert das konservierte HisA2 und soll den Elektronentransfer unterstützen.18, 19 Die Thioetherbrücke ist konserviert in pflanzlichen und pilzlichen PPOs,7, 13, 33 während sie normalerweise in PPOs von Bakterien und Säugetieren fehlt (Abbildung S12).14 Fünf Mutanten (Ala, Gly, Asp, Asn und Ser anstelle von Cys97) wurden hergestellt, um die Bildung der Thioetherbrücke zu verhindern. Alle fünf Mutanten zeigten Hydroxylaseaktivität (Tabelle 1), die vermutlich auf die Freisetzung von HisA2 von der Thioetherbrücke zurückzuführen ist (Abbildung 1).32 Mutanten ohne Thioetherbrücke ermöglichen es HisA2 sich dem Eingang des dinuklearen Kupferzentrums zu nähern, als Base zu fungieren und zusätzlich seine Basizität durch Wechselwirkung mit dem konservierten Wasserträger Glu248 zu erhöhen (Abbildung 1). Die Dreifachmutante Cys97Ser/Thr253Asp/Arg257Asp, die auf die drei in dieser Arbeit untersuchten Hauptpositionen abzielte (Abbildung 1), zeigte eine geringere Aktivität auf Tyramin (kcat=6,52 s−1) als die Doppelmutante Thr253Asp/Arg257Asp (9,48 s−1, Tabelle 1), was darauf hindeutet, dass HisA2 die durch HisB1 und HisB2 verursachte Deprotonierung des Substrats nicht signifikant erhöht. Obwohl nicht schneller, so war doch die katalytische Effizienz der Mutante Cys97Ser/Thr253Asp/Arg257Asp mit ≈326 s−1 mm−1 im Vergleich zu Thr253Asp/Arg257Asp mit ≈8,7 s−1 mm−1 aufgrund der hohen Spezifität der Dreifachmutante für Tyramin (Km=0,02 mm, Tabelle 1) deutlich höher.
Der Torwächter (Phe273), der Wasserträger (Glu248), das 7. Histidin (His285) und die Cys31- und Cys32-Reste: Diese Studie berichtet auch über Mutanten, die den Torwächter (Phe273), den Wasserträger (Glu248), das 7. Histidin (His285) sowie die Cys31- und Cys32-Reste abändern. Einige dieser Mutanten haben einen geringen Einfluss auf die CH-Aktivierung, üben jedoch einen signifikanten Einfluss auf die Gesamtaktivität des Enzyms aus, welche in den meisten Fällen beeinträchtigt ist (siehe SI).
Flexibilität der dem Substrat zugewandten konservierten Histidine als Voraussetzung für die CH-Aktivierung in Typ-III-Kupferzentren: In PPOs ist die Flexibilität der beiden Kupferionen hoch, was durch kristallographische Daten verschiedener Oxidationszustände des Typ-III-Kupferzentrums (oxy-, met- und deoxy-Formen) gestützt wird. Die Strukturen von oxy (PDB: 4Z13) und deoxy CgAUS: (PDB: 4Z14) weisen einen Unterschied von ≈1 Å im Kupfer-Kupfer-Abstand auf. Bei der Bildung der Oxyform bewegen sich beide Kupferatome (CuA um 0,5 Å und CuB um 0,7 Å) von ihrer ursprünglichen Deoxykupferposition.7 Kürzlich haben Matoba et al. die bakterielle TYR von Streptomyces castaneoglobisporus untersucht und sie zeigten, dass die Flexibilität der beiden Cu-Ionen einen Einfluss auf die Position der konservierten Histidine hat.32 Darüber hinaus haben Fujieda et al. einen ähnlichen Einfluss der Kupferionenflexibilität auf das konservierte His von Aspergillus oryzae TYR gezeigt und basierend auf kristallographischen Studien wurde vorgeschlagen, dass die Deprotonierung des Substrats wahrscheinlich durch HisA3 (His103) aufgrund seiner verlorenen Wechselwirkungen mit CuA während der Substratinteraktion erfolgt.35 Darüber hinaus zeigte die Kristallstruktur der aktiven Pilz-TYR AbPPO433 zwei CuB-Konformationen mit einem Abstand von 2,3 Å zwischen ihnen. Die zweite CuB-Position zeigt die Wechselwirkung mit einer vierten Imidazolgruppe (drei mit den konservierten Histidinen und eine weitere mit dem 7. Histidin) an. Daher kann in AbPPO4 das 7. Histidin als Komplexierungsreserve reagieren und die Flexibilität des Kupferions und der Histidine unterstützen.33 Alle diese Untersuchungen zeigen, dass PPOs ein flexibles Dikupferzentrum enthalten, in dem die beiden Kupferionen unterschiedliche Positionen einnehmen können und folglich die konservierten Histidine neben der Kupferkoordination für die zusätzliche Aufgabe der Substratdeprotonierung zur Verfügung stehen.
Bildung des Oxykomplexes als Hauptschritt im Monophenolasezyklus: Alle CgAUS-Mutanten wurden mit H2O2 titriert und die Ausbildung ihrer Oxyform durch Bestimmung des Absorptionskoeffizienten ϵ345 (Tabelle 1 und S1) und der Anzahl der zur Sättigung benötigten Äquivalente untersucht (Tabelle S1). Alle Mutanten, die auf die Positionen HisB1+1 und HisB2+1 (Thr253 und Arg257) abzielten und Monophenolaseaktivität zeigten, bildeten ebenfalls ein Oxyaddukt. Die Mutanten Thr253Ile, Arg257Leu und Arg257Ile konnten monophenolische Substrate nicht hydroxylieren, obwohl sie ein starkes durch H2O2 induziertes Oxyaddukt zeigten (Tabelle S1). Daher ist die Fähigkeit von PPOs, ein stabiles Oxyaddukt zu bilden, lediglich der erste Schritt im Monophenolasezyklus (Abbildung 2).
Monophenolasezyklus für Typ-III-Kupferenzyme (Abbildung 2): Deoxy-CgAUS (CuI) bindet molekularen Sauerstoff und bildet die Oxy-Form (CuII) (1→2). Das ankommende monophenolische Substrat muss deprotoniert werden, was von einem der drei konservierten Histidine HisA2, HisB1 und HisB2 vor dem aktiven Zentrum (2→3 A, 3 B und 3 C) unterstützt wird. Aufgrund der Flexibilität der beiden Kupferionen dienen die abgelösten Histidine als Basen (3). Die Reaktivität der drei Histidine (HisA2, HisB1 und HisB2) wird durch ihre benachbarten Reste (HisB1+1, HisB2+1 und den Wasserträger) eingestellt, um entweder als stärkere Basen zu reagieren oder/und eine Position zu stabilisieren, die für das Abstrahieren von Substratprotonen geeignet ist (3). Das deprotonierte Substrat bindet an CuA32 oder in der Mitte der beiden Kupferionen,7 was zur Hydroxylierung führt (4). Das Dikupferzentrum wandelt sich nach Übertragung eines Sauerstoffatoms auf das Substrat (5) in die met-Form um, und die Oxidation des diphenolischen Substrats führt zum endgültigen Chinonprodukt, während das Enzym für einen neuen Katalysezyklus in die Deoxyform zurückkehrt (5→1).
Implikationen: Zunächst wurden die Reste identifiziert, die die Substratspezifität in Typ-III-Kupferproteinen steuern. Die an diesen Positionen vorhandenen Aminosäuren verursachen die Ungleichheit der strukturell ähnlichen aktiven Zentren in TYRs und COs. Zweitens haben wir gezeigt, wie ein Enzym vom CO-Typ durch Mutation der Reste HisB1+1, HisB2+1 oder des Cys der Thioetherbrücke in eine TYR umgewandelt werden kann. Schließlich tragen unsere Ergebnisse zum grundlegenden Verständnis des Monophenolase-Reaktionszyklus bei (Abbildung 2). Die vorliegende Studie erklärt zum ersten Mal, dass die Hydroxylaseaktivität in Typ-III-Kupferenzymen ein Ergebnis der Flexibilität der drei konservierten HisA2, HisB1 und HisB2 ist, die es ihnen ermöglicht, als Basen zu fungieren und monophenolische Substrate zu deprotonieren, also im Gegensatz zu COs in TYRs die CH-Aktivierung zu initiieren. Die Steuerung der C−H-Aktivierungsreaktion wird sich grundlegend auf mehrere wichtige Anwendungen in den Bereichen Medizin (Melanom), Biotechnologie, Bioremediation, Nacherntetechnologie, Textiltechnologie, Weinherstellung und anderen wichtigen Bereichen auswirken.
Acknowledgements
Die Forschung wurde vom Österreichischen Wissenschaftsfonds (FWF) P25217 und P32326 sowie von der Universität Wien finanziert. Wir danken Rosa Heydenreich B.Sc. für ihre Unterstützung bei der Proteinexpression.
Conflict of interest
Die Autoren erklären, dass keine Interessenkonflikte vorliegen.