Isoxazol-Nukleoside als Bausteine für eine plausible Proto-RNA
Abstract
Die Frage, wie sich die RNA als ein Träger der genetischen Information entwickelt hat, ist von grundlegender Bedeutung für unser Verständnis des Ursprungs des Lebens. Das RNA-Molekül ist viel zu komplex, als das es in einem einzigen Evolutionsschritt hat entstehen können, was darauf hindeutet, dass es Proto-RNAs (erste Vorläufer der RNA) gegeben haben könnte, die sich im Laufe der Zeit zu der heutigen RNA entwickelt haben. Hier zeigen wir, dass Isoxazol-Nukleoside, die aus Hydroxylamin, Cyanoacetylen, Harnstoff und Ribose gebildet werden, plausible Vorläufer für RNA sind. Das Isoxazol-Nukleosid kann sich innerhalb eines RNA-Strangs zu Cytidin umlagern, was zu einer Erhöhung der Paarungsstabilität führt. Wenn die Proto-RNA ein kanonisches seed-Nukleosid mit definierter Stereochemie enthält, kann das seed-Nukleosid die Konfiguration des anomeren Zentrums kontrollieren, das sich, während der in-RNA-Umwandlung bildet. Die Ergebnisse zeigen, dass sich die RNA nach der Strangbildung aus evolutionär primitiven Vorläufern wie Isoxazol-Ribosiden entwickelt haben könnte.
Alle bisher entwickelten Konzepte, um zu erklären, wie sich RNA-Oligonukleotide auf der frühen Erde gebildet haben könnten, gehen von der präbiotischen Bildung von Nukleosiden aus, die in einer enzymfreien Reaktion oligomerisieren.1-6 Diese Konzepte beruhen auf der Vorstellung, dass die Nukleoside zunächst in präbiotischen Reaktionen entstanden sein müssen, dann phosphoryliert wurden7-10 und anschließend oligomerisierten (Abbildung 1a).2, 11-15 Dieses Szenario erfordert komplexe mehrstufige präbiotische Reaktionen zur Erzeugung der Nukleoside als Voraussetzung für die RNA-Bildung.16 Die Komplexität dieses Szenarios hat schon früh zu der Vorstellung geführt, dass die RNA durch einen chemischen Evolutionsprozess aus einer viel einfacheren Proto-RNA (erster Vorläufer der RNA) entstanden sein könnte.17-22 In diesem Sinne ist es denkbar, dass es eine Proto-RNA gab, die aus Proto-Nukleosiden (Vorläufer-Nukleosiden) aufgebaut war, die sich innerhalb der Proto-RNA zu RNA umgelagert haben. Es ist auch denkbar, dass diese Proto-Nukleoside eine gemischte Proto-RNA bildeten mit einigen eingebauten kanonischen RNA-Nukleosiden als seed-Nukleotide.23 Die Proto-RNA-Nukleoside könnten sich dann umlagern, um schließlich RNA-Stränge zu bilden, die ausschließlich aus kanonischen Nukleotiden bestehen (Abbildung 1a). Durch den Abbau dieser umgelagerten RNA, der auf der frühen Erde mit Sicherheit stattgefunden hat, würden die kanonischen Nukleoside freigesetzt, so dass diese als seed-Nukleotide in wachsende Proto-RNA wieder eingefügt werden konnten. Hier zeigen wir, dass ein solches Konzept tatsächlich RNA hervorbringen kann und erbringen den Nachweis, dass die Stereoinformation der kanonischen seed-Nukleotide das stereochemische Ergebnis der Umlagerung von Proto-RNA zu RNA steuern kann. Ein Hindernis für ein solches Konzept ist, dass die glykosidischen Bindungen in den meisten RNA-Vorläufer-Nukleosiden zu labil sind, so dass keine stabile Strangbildung stattfinden kann. Dies ist ein besonderes Problem für nichtzyklische Vorläufer-Nukleoside, die typischerweise ein saures anomeres H-Atom am glykosidischen N-Atom aufweisen. Ausnahmen hiervon sind jedoch auf Harnstoff basierende Proto-Nukleoside, bei denen das saure H-Atom durch eine interne H-Bindung stabilisiert wird.24
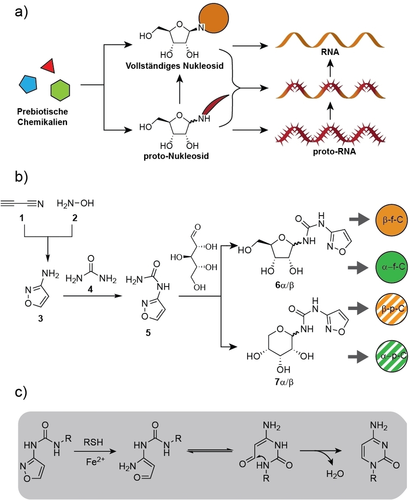
a) Die klassische präbiotische Chemie liefert zunächst die vollständigen Nukleoside, die dann in RNA umgewandelt werden. Alternativ könnten die Proto-Nukleoside eine Proto-RNA oder eine gemischte RNA-Proto-RNA gebildet haben, aus der die RNA entstand. b) Synthese der 3-Harnstoff-Isoxazol-Nukleoside 6 und 7. Nach dem Einbau in die RNA lagert sich 6α/β direkt in der RNA zu Cytidin um. c) Mechanismus der Reaktion.
Ein kürzlich vorgestelltes Konzept (Abbildung 1b), wie das Pyrimidin-Nukleosid Cytidin auf der frühen Erde entstanden sein könnte, basiert auf der Reaktion von Cyanoacetylen (1) mit Hydroxylamin (2) zu 3-Aminoisoxazol (IO3, 3).25 Es wurde gezeigt, dass 3 mit Harnstoff (4) zu N-Isoxazolyl-Harnstoff 5 reagiert, der anschließend mit Ribose zu den entsprechenden Furanosiden (6α,β) und Pyranosiden (7α,β) reagieren kann. Durch die anschließende reduktive N−O-Bindungsspaltung mit Thiolen in Gegenwart katalytischer Mengen von Fe2+ erhält man Cytidin (Abbildung 1c).25-28 Hier berichten wir, dass, die durch N−O-Spaltung induzierte Umlagerung des ursprünglich gebildeten N-Isoxazolyl-Harnstoffes 5 auch direkt in der RNA stattfinden kann, wenn die Verbindung in (RNA)-Proto-RNA-Strukturen eingebaut ist. Diese “in-RNA”-Reaktion erschafft ein neues Stereozentrum. Spannenderweise konnten wir beobachten, dass die Konfiguration bereits vorhandener kanonischer seed-Nukleoside das stereochemische Ergebnis der “in-RNA”-Zyklisierungsreaktion kontrolliert, was zu einer Amplifizierung der stereochemischen Information führt.
Um zunächst die Möglichkeit zu untersuchen, die 6α/β zu Cytidin Umlagerung direkt in der RNA durchzuführen, haben wir die IO3 Phosphoramidite α-8 und β-8 hergestellt und beide Bausteine in die verschiedenen RNA-Stränge eingebaut (Tabelle 1). Die Synthese von 8 ist in Schema 1 dargestellt. Sie begann mit 1-Azidoribose 9, die zunächst mit (tBu)2SiCl2 (DMF, RT, 78 %) 3′-5′-geschützt wurde, um 10 zu erhalten. Durch die anschließende Schützung der 2′-OH-Gruppe mit TBS-Cl (DMF, Imidazol, RT, 78 %) wurde 11 erhalten. Es folgte eine one-pot-Reduktion des Azids zum Amin (Pd/C, kat. XPhos, Toluol, CO, 5 bar, 60 °C) und die Reaktion des Amins mit IO3 3, um das Ribofuranosid 12 als die erwarteten α/β-Gemische zu erhalten. Die beiden IO3-Anomere α-12 und β-12 konnten durch Flash-Säulenchromatographie getrennt werden. Die Nukleoside wurden anschließend unabhängig voneinander zu 13 entschützt (HF-py, CH2Cl2, 0 °C) und in das 5′-Dimethoxytrityl-geschützte Produkt 14 umgewandelt (DMT-Cl, Pyridin, RT). Die 5′-DMT-2′-TBS-geschützten IO3-Riboside 14 wurden schließlich in die Phosphoramidite α-8 und β-8 umgewandelt. Während wir uns zunächst Sorgen über die Stabilität der Isoxazol-Riboside machten, lernten wir während der Synthese, dass alle IO3-Nukleoside erstaunlich stabil sind. Ähnlich wie bei den zuvor berichteten Harnstoff-Nukleosiden,24 haben wir während der Reaktion keine Deglykosylierung festgestellt, was angesichts der aciden NH-Gruppe am anomeren Zentrum überraschend ist.
Sequenz Name |
Sequenz (5′→3′) |
berechnet [M−H]− |
gefunden (m/z) |
|---|---|---|---|
S1 |
5′-CUU ACβIO3 CUG A-3′ |
3094.4 |
3094.6 |
A-S1 |
5′-CUU ACA CUG A-3′ |
3102.4 |
3102.3 |
G-S1 |
5′-CUU ACG CUG A-3′ |
3118.4 |
3118.3 |
C-S1 |
5′-CUU ACC CUG A-3′ |
3078.7 |
3078.3 |
U-S1 |
5′-CUU ACU CUG A-3′ |
3079.4 |
3079.5 |
A-R1 |
5′-UCA GAG UAA G-3′ |
3205.5 |
3205.4 |
G-R1 |
5′-UCA GGG UAA G-3′ |
3221.5 |
3221.2 |
C-R1 |
5′-UCA GCG UAA G-3′ |
3181.5 |
3181.8 |
U-R1 |
5′-UCA GUG UAA G-3′ |
3182.4 |
3182.1 |
S2 |
5′-βCβCIO3 βCβCIO3 βCβC-3′ |
2409.4 |
2409.3 |
S3 |
5′-αCαCIO3 αCαCIO3 αCαC-3′ |
2409.4 |
2409.1 |
S4 |
5′-αCβCIO3 αCβCIO3 αCβC-3′ |
2409.4 |
2408.6 |
S5 |
5′-IO3IO3IO3 IO3–3′ |
1222.2 |
1221.9 |
S6 |
5′-IO3IO3IO3 IO3βC-3′ |
1526.2 |
1526.2 |
S7 |
5′-IO3IO3IO3 IO3αC-3′ |
1526.2 |
1526.2 |
S8 |
5′-GGU βIO3GA CC3′ |
2538.4 |
2535.5 |
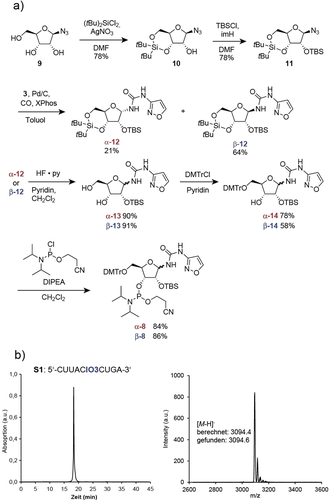
a) Synthese des IO3-Phosphoramidits α,β-8 und Bedingungen. b) HPL-Chromatogramme und MALDI-TOF-Massenspektren des als Beispiel gezeigten Oligonukleotids S1, das mit dem IO3-Phosphoramidit β-8 synthetisiert wurde.
Um zu untersuchen, ob ein Einbau der IO3-Nukleoside in RNA möglich ist, verwendeten wir Standard-Festphasen-Oligonukleotidchemie und die Phosphoramidite α-8 und β-8. Die RNA-Synthese war ohne große Anpassungen des RNA-Synthese- und Entschützungsprotokolls möglich. Wir mussten lediglich die Kopplungszeit von α-8 und β-8 auf 600 s verlängern. Die Entschützung erfolgte zunächst mit Ammoniumhydroxid/Methylamin (1 : 1), gefolgt von einem zweiten Entschützungsschritt mit TEA⋅3HF. Schema 1b zeigt das HPLC-Chromatogramm des synthetisierten RNA-Strangs S1 als Beispiel, der ein einzelnes β-konfiguriertes IO3 enthält. Das MALDI-TOF-Massenspektrum beweist die Integrität der erhaltenen RNA S1. Mit diesem experimentellen Beweis für den möglichen und effizienten Einbau des IO3-Nukleosids in die RNA haben wir als nächstes dieses Protokoll zur Herstellung der RNA-Stränge S1 – S8 verwendet, die mehrere IO3-Nukleoside in gemischten Sequenzen enthalten.
Um die “in-RNA”-Umlagerung der IO3-Nukleoside zu C zu untersuchen und das stereochemische Ergebnis der Reaktion zu studieren, stellten wir zunächst den RNA-Strang S2 (Abbildung 2a) her, der zwei 3-Isoxazol-Nukleoside enthält, die in ein 8mer mit 6 zusätzlichen Cytidinen eingebettet sind. Für die Synthese verwendeten wir die Phosphoramidit-Mischung α/β-8. Um das Verhältnis nach der RNA-Synthese und der HPL-chromatographischen Aufreinigung des Strangs zu untersuchen, verdauten wir S2 nach der Synthese mit dem New England BioLabs Nucleoside digestion mix (Kat.-Nr. NEB M0649S). Das erhaltene Nukleosidgemisch wurde anschließend mittels LC-HESI-MS analysiert (grauer Kasten in Abbildung 2a). Wie erwartet, ist das Vorhandensein von β-C-Nukleosiden deutlich zu erkennen. Neben β-C wurden zwei zusätzliche Signale für die beiden Isoxazol-Nukleoside (α-IO3,β-IO3) nachgewiesen. Die Integration der Signale zeigt ein α/β-Verhältnis von 1 : 4. Als nächstes behandelten wir den Isoxazol-RNA-Strang S2 mit Dithiothreitol (DTT) und katalytischen Mengen von Fe2+ bei 90 °C für 2 h. Die HPL-chromatographische Analyse der Transformation zeigte eine saubere spot-to-spot-Konversion (Abbildung 2a). Während der ursprüngliche RNA-Strang S2 eine Retentionszeit von 33 min hatte, verschwand dieses Signal vollständig zugunsten eines neuen HPLC-Signals bei 23 min, das dem reduzierten Produktstrang S2red entspricht. Wichtig ist die Beobachtung, dass nur ein einziges Signal detektiert wurde und nicht eine Reihe von Signalen, wie es für ein diastereoisomeres Produktgemisch zu erwarten wäre. Um das stereochemische Ergebnis zu untersuchen, verdauten wir den RNA-Strang S2red erneut und analysierten das Nukleosidgemisch mittels LC-HESI-MS (Abbildung 2b). Dieses Experiment zeigte zu unserer Überraschung nur ein Signal für die Verbindung β-C. Die Co-Injektion von α-C bestätigte dieses experimentelle Ergebnis. α-C wird eindeutig nicht während der N−O-Bindungsspaltung und der Umlagerung zu C im RNA-Strang gebildet. Dieses Ergebnis zeigt, dass das α/β-Gemisch des IO3-Nukleosids in der RNA ausschließlich durch “Verstärkung” der von den seed-Cytidinen bereitgestellten stereochemischen Information zu β-C umgelagert wurde.
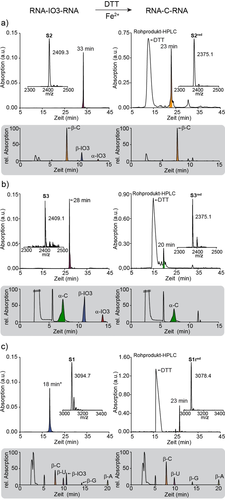
Reaktion der IO3-haltigen Stränge mit DTT und Fe2+ und HPLC-, MALDI-, HPLC-MS-Analysen (bei 223 nm; graue Klammern) der N−O-Spaltung und Zyklisierungsreaktion (links: Edukt; rechts: Produkt). a) Ergebnis des β-homo-C-Strangs S2; b) des α-homo-C-Strangs S3; c) des Strangs S1. A=Adenosin, C=Cytidin, G=Guanosin, IO3=3-Aminoisoxazol, U=Uridin. *HPLC-Gradient: 0–40 % Puffer B.
Um diesen stereochemischen Amplifikationsschritt zu belegen, stellten wir als nächstes den RNA-Strang S3 her, bei dem wir die flankierenden β-Cs von S2 durch entsprechende α-Cs ersetzten. Der RNA-Strang S3 wurde wiederum in ausgezeichneter Qualität synthetisiert (Abbildung 2b). Die Verdauung und LC-HESI-MS-Analyse vor der N−O-Bindungsspaltung zeigt erneut das Vorhandensein der IO3-Nukleoside als α/β-Gemisch. Als wir die N−O-Bindungen von S3 öffneten und das Nukleosidgemisch nach dem Verdau analysierten, beobachteten wir zu unserer Überraschung wieder nur ein einziges HPLC-Signal. Diesmal wurde nur α-C detektiert, was zeigt, dass die “in-RNA”-Umlagerung des α/β-Gemischs von IO3 nur noch das α-konfigurierte Produkt lieferte. Auch hier muss die Stereoinformation des anomeren Zentrums der seed-Nukleoside das stereochemische Ergebnis der Zyklisierungsreaktion bestimmt haben (Abbildung 2b). Als nächstes führten wir dieselbe Reaktion mit Strang S1 durch, um zu zeigen, dass die stereochemische Verstärkung auch mit anderen kanonischen Basen erreicht wird. Wie erwartet, wiesen wir nur β-konfiguriertes C in unserem Produktstrang S1red nach (Abbildung 2c). Diese Daten zeigen, dass die flankierenden Basen offensichtlich stereochemische Informationen übertragen, so dass die Zyklisierungsreaktion mit hoher Stereospezifität abläuft. Wir glauben, dass der Informationstransfer durch eine Optimierung der Basenstapelkräfte verursacht wird. Eine stereochemisch homogene α oder β-RNA kann in wässriger Lösung bessere Basenstapelungskräfte aufbauen.
Um zu beweisen, dass das Vorhandensein von seed-Nukleosiden eine Voraussetzung für die stereochemische Amplifikation ist, haben wir zwei Kontrollversuche durchgeführt. Zunächst verwendeten wir den Strang S4, ein 8mer Oligonukleotid, welches sowohl α- als auch β-seed-Cytidin im Verhältnis von 1 : 1 enthält. Die Proto-RNA S5 hingegen enthielt keine seed-Cytidine. Nach Behandlung dieser Stränge mit DTT und Fe2+ zeigte die anschließende LC-HESI-MS-Analyse erwartungsgemäß die Bildung einer 1 : 1-Mischung von α/β-Cytidinen (siehe SI).29
Auf der Grundlage dieses Ergebnisses entwarfen wir als nächstes zwei 5mere mit nur einem informationstragenden Nukleosid in Form eines einzelnen β-C (S6) oder α-C (S7) an der 3′-Position. Die LC-HESI-MS-Analyse der Verdauungsprodukte nach der N−O-Bindungsspaltung und der anschließenden Umlagerung ergab nun für beide Stränge Gemische von α/β-C. Es ist jedoch eine deutliche stereochemische Tendenz zu beobachten. Für den β-C tragenden Strang S6red wurde ein α/β-Verhältnis von 1 : 3 und für den α-C tragenden Strang S7red ein α/β-Verhältnis von 2 : 1 ermittelt. Dies zeigt, dass ein einziges stereochemisch definiertes seed-Nukleotid bereits ausreicht, um das stereochemische Ergebnis der Zyklisierungsreaktion zu beeinflussen.
Damit eine chemische Verbindung als instruktives Proto-Nukleosid fungieren kann, muss das Nukleosid zur Stabilität der RNA beitragen und Informationen kodieren können.30, 31 Daher untersuchten wir, ob das Nukleosid β-IO3 in der Lage ist, informative Wechselwirkungen mit einer der anderen RNA-Basen herzustellen. Für diese Studie synthetisierten wir verschiedene Gegenstränge zu S1 (A/C/G/U-R1), bei denen wir die dem IO3 gegenüberliegende Base variierten und die Schmelzpunkte bestimmten. Außerdem stellten wir vier S1 Referenzstränge (A/C/G/U-S1) her, bei denen wir die IO3 durch eine der anderen kanonischen Basen ersetzten (Abbildung 3a). Der Duplex mit dem Basenpaar G : C (G-S1:C-R1) an der entsprechenden Position weist erwartungsgemäß den höchsten Schmelzpunkt von 55 °C auf. Das Basenpaar A : U an dieser Position bildet einen Duplex (A-S1:U-R1), der bei Tm=48 °C schmilzt. Für den Duplex G-S1:U-R1 mit einem G : U Wobble-Basenpaar an der entsprechenden Position wurde ein Schmelzpunkt von 43 °C gemessen. Als wir die Basenpaare IO3-S1:R1 untersuchten, stellten wir fest, dass die IO3:G-Paarung einen Duplex IO3-S1:G-R1 mit einem Schmelzpunkt von 45 °C erzeugt, der sogar höher lag als der für das G : U-Wobble-Basenpaar. Noch wichtiger ist, dass die Selektivität der Paarung bemerkenswert ist. Während der IO3:G-Duplex bei 45 °C schmilzt, bildeten alle anderen Basenpaare Duplexe mit Schmelzpunkten von 31 °C oder darunter, was einen ΔTm von mindestens 14 °C ergibt. Dies ist eine ungewöhnliche Unterscheidung zwischen der produktiven IO3:G-Situation und allen anderen Basenpaarungsmöglichkeiten.
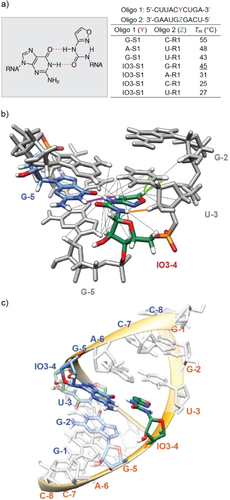
a) Chemische Struktur und Basenpaarungseigenschaften eines IO3:G-Basenpaares und Zusammenfassung der Tm-Analysen für kanonische und IO3-haltige Oligonukleotide. Die Lösungen wurden mit 10 mM Natriumphosphat (pH 7) und 150 mM NaCl gepuffert. b) Vorhergesagte Orientierung des IO3 mit allen NOE-Kontakten (schwarze Linien) und den wichtigsten Kontakten für die Strukturbestimmung hervorgehoben (IO3-4H3:G5H1 (lila), IO3-4H1:U3H2′ (orange), IO3-4H7:U3H5 und IO3-4H7:U3H6 (grün)). c) Strukturmodell von S8, das die nicht-kanonische Basenpaarung zwischen G-5 (blau) und IO3-4 (grün) zeigt.
Um den Grund für die konstruktive Interaktion des IO3:G-Basenpaares zu untersuchen, entwarfen wir zwei palindromische 8mer-RNA-Stränge S8 und Can8 (siehe Supporting Information), die nach der Hybridisierung entweder G : U- oder IO3:G-Wobble-Basenpaare an den beiden zentralen Positionen in jedem Duplex bilden (GGUXGACC). Diese Stränge wurden in mg-Mengen hergestellt, und die gebildeten Duplexe wurden mittels NMR-Spektroskopie analysiert (Abbildung S7). Die hohe Ähnlichkeit der chemischen Verschiebung, die bei beiden Proben sowohl in homo- als auch in heteronuklearen Experimenten beobachtet wurde, zeigte uns, dass die beiden Palindrome, Doppelstränge mit einer A-ähnlichen Gesamtkonformation bilden, die typisch für RNA-Duplexe ist (Abbildung S7–S11, Tabelle S1). 2D 1H–1H NOESY wurde anschließend eingesetzt, um die Konformation der IO3-Base aufzuklären (Abbildung 3b, siehe Hintergrundinformationen für erweiterte Methoden). Die wahrscheinlichste Orientierung wurde durch Analyse der cross peaks in der Iminoregion bestimmt. Es ist eine beträchtliche Verschiebung des G-5H1 Protons nach oben zu beobachten (von 12.1 auf 11.0 ppm), was auf das Vorhandensein einer schwachen Wasserstoffbindung von G-5 zu IO3–4 hinweist (Abbildung S11). Der intensive cross peak zwischen G-5H1 und IO3-4H3 (violette Linie in Abbildung 3b), der sogar bei einer kurzen Mischzeit von 40 ms nachgewiesen werden konnte, zeigt, dass sich die beiden Basen gegenüberstehen. Diese Beobachtung impliziert, dass IO3-4H3 in Richtung des zweiten Strangs und damit weg vom Zuckerphosphatgerüst zeigt. Ein cross peak zwischen den Protonen G-5H1 und IO3-4H8 bestätigt, dass der Isoxazolring von IO3-4 in Richtung G-5 zeigt. Ein cross peak mit dem Zuckerproton U-3H2′ (orange Linie in Abbildung 3b) deutet außerdem darauf hin, dass das IO3-4H1 in Richtung des Zuckerphosphatgerüsts ragt, ähnlich wie die H8-Protonen in Adenin und Guanin oder die H5-Protonen von Cytosin oder Uracil. Schließlich weisen die starken cross peaks zwischen IO3-4H7 und den aromatischen Protonen von U-3 (grüne Linien in Abbildung 3b) und die schwächeren cross peaks von IO3-4H8 zu diesen Protonen darauf hin, dass IO3-4 und U-3 gestapelt sind. Dieses spricht dafür, dass der aromatische Ring in Richtung der Hauptfurche ausgerichtet ist.
Dies führt zu einer Ausrichtung, bei der IO3-4H1 eine intramolekulare Wasserstoffbrücke mit IO3-4O4 bilden kann. Bei einer solchen Anordnung zeigt die Carbonylgruppe in Richtung des Guanins, was, in Übereinstimmung mit den Schmelzpunktstudien, eine produktive Basenpaarungsinteraktion mit G-5H1 ermöglicht. Insgesamt zeigt diese Analyse, dass IO3 eine flache Konformation annimmt, in der die NH-CO-Einheit zur Gegenbase G zeigt, um konstruktive H-Bindungen zu bilden. Es ist die Harnstoff-Substruktur, die H-Bindungen mit der G-Gegenbase eingeht. Die Tatsache, dass sich nur zwei H-Bindungen bilden können, steht im Einklang mit dem niedrigeren Schmelzpunkt. Die 2D-1H–1H-NOESY-Daten wurden für Strukturberechnungen verwendet (Abbildung 3c). Das für das modifizierte 8mer erhaltene Niedrigenergie-Strukturmodell zeigt, dass das IO3:G-Basenpaar sterisch perfekt in die Doppelstrangstruktur eingebaut ist. Interessant ist die Tatsache, dass die interne H-Bindung zwischen dem Isoxazol N und dem Harnstoff N−H die Struktur stark stabilisiert. Das IO3-Nukleosid bildet 2 Wasserstoffbrückenbindungen mit der G-Gegenbase, die sich bei der Umlagerung zu C auf 3 Wasserstoffbrücken erhöhen, was mit einer weiteren Stabilisierung des Basenpaares einhergeht. Dies könnte die treibende Kraft in der Evolution gewesen sein.
Zusammenfassend zeigen wir, dass Isoxazol-Harnstoff-Nukleoside, die unter präbiotisch plausiblen Bedingungen aus Cyanoacetylen, Hydroxylamin, Harnstoff und Ribose25 gebildet werden, ein Basenpaar mit G bilden können, das eine mit einem U : G-Wobble-Basenpaar vergleichbare Stabilität aufweist, die auf zwei instruktiven H-Bindungen beruht (Abbildung 3a). Noch wichtiger ist, dass sich das Basenpaar IO3:G selektiv bildet, da alle anderen Kombinationen (IO3:A/C/U) wesentlich schwächer sind. Wir zeigen, dass die N−O-Bindung des Isoxazol-Nukleosids innerhalb eines Proto-RNA-Strangs mit Thiolen und katalytischen Mengen von Fe2+ gespalten werden kann. Diese Spaltung löst eine Zyklisierungs-Eliminierungs-Kaskade aus (Abbildung 1c), die zu β- oder α-Cytidin führt. Wichtig ist das Vorhandensein von α- oder β-seed-Nukleosiden, welche das stereochemische Ergebnis der Umlagerungsreaktion bestimmen können.
Wir wissen nicht, ob solche IO3-Nukleoside einst Bestandteile einer frühen Proto-RNA-Welt waren, aber es wird allgemein angenommen, dass der RNA einfachere Vorläuferstrukturen vorausgingen.16, 30 Hier zeigen wir, dass Isoxazol-Nukleoside hervorragende Kandidaten für solche Vorläufernukleoside und Proto-RNAs sind. Da sich die Proto-Nukleoside auch in Lösung zu C umlagern können, liegt die Vermutung nahe, dass IO-Nukleoside und die kanonischen α-/β-Nukleoside simultan existiert haben könnten, um Proto-RNA-Stränge zu bilden, die einige kanonische seed-Cytidine enthalten haben könnten. Diese Nukleoside könnten dann das stereochemische Ergebnis der Umlagerung gesteuert haben, die das Basenpaar G:IO3 in das besser paarende Basenpaar G : C umwandelte. Da sich α- und β-Cs mit der gleichen Wahrscheinlichkeit bilden, bieten unsere Daten keine Lösung für die Frage, wie es zur Homo-Chiralität in der RNA kam.32 Die Daten zeigen jedoch, dass die stereochemische Information durch “in-RNA-Reaktionen” von Vorläufernukleosiden zu kanonischen Nukleosiden amplifiziert worden sein könnte, dieses Ergebnis kann zum Nachdenken über den Ursprung der Homo-Chiralität anregen.
Danksagung
Wir danken Stefan Wiedemann für die Synthese der Referenzverbindungen und Pia Heinrichs für die Unterstützung bei den NMR-Messungen der Oligonukleotide. Wir danken auch Dr. Luis Escobar und Jonas Feldmann für wertvolle Diskussionen. Diese Arbeit wurde von der Deutschen Forschungsgemeinschaft (DFG) über CRC1309 (Projekt-ID 325871075, A04), CRC1361 (Projekt-ID 893547839, P02) und CRC1032 (Projekt-ID 201269156, A5) finanziell unterstützt. Dieses Projekt wurde vom Europäischen Forschungsrat (ERC) im Rahmen des Forschungs- und Innovationsprogramms Horizont 2020 der Europäischen Union unter der Fördervereinbarung Nr. 741912 (EpiR) gefördert. Wir sind dankbar für die zusätzliche Finanzierung durch die Volkswagen-Stiftung (EvoRib). Open Access Veröffentlichung ermöglicht und organisiert durch Projekt DEAL.
Interessenkonflikt
Die Autoren erklären, dass keine Interessenkonflikte vorliegen.
Open Research
Data Availability Statement
Die Daten, die die Ergebnisse dieser Studie unterstützen, sind in den Hintergrundinformationen zu diesem Artikel verfügbar.